Top-down design bringer nye DNA-strukturer til live
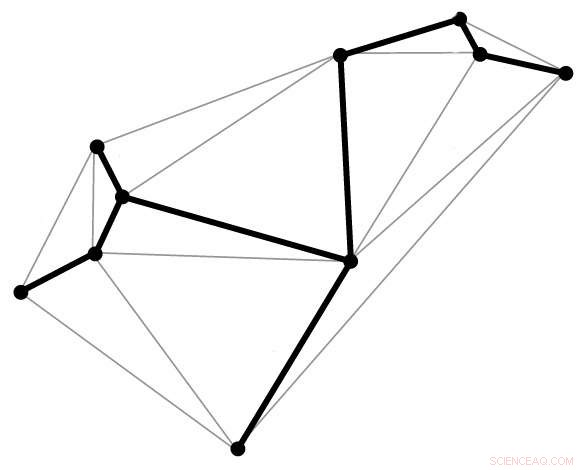
Den fede linje, kendt som et spændingstræ, følger den ønskede geometriske form, at røre ved hvert toppunkt kun én gang. En spændende træ -algoritme bruges i den nye DNA origami -metode til at kortlægge den korrekte routingsti for DNA -strengen. Kredit:Public Domain
Blandt de værdifulde besiddelser i Londons Wellcome Library er en grov blyantskitse lavet i 1953 af Francis Crick. Tegningen er en af de første, der viser DNA's dobbelthelixstruktur - naturens plan for design af havsnegle, mennesker, og enhver anden levende form på jorden.
De færreste kunne dog have forudset, at DNA's simple egenskaber ved selvsamling, og dens alsidige informationskapacitet, kunne bruges til mange formål, som Watson og Crick aldrig havde forestillet sig, (eller faktisk, af naturen selv).
I ny forskning, der vises i den forskudte onlineudgave af tidsskriftet Videnskab , Mark Bathe, sammen med kollegerne Hao Yan fra ASU og Wah Chiu fra Baylor College of Medicine beskriver en ny metode til at designe geometriske former bygget ud fra DNA. De præsenterer en ny variant på en teknik kendt som DNA-origami, hvor DNA'ets baseparringsegenskaber udnyttes til konstruktion af bittesmå strukturer i 2 og 3 dimensioner.
"En vigtig udfordring inden for DNA-nanoteknologi er at designe eventuelle ønskelige strukturer på en top-down måde, uden megen menneskelig input vedrørende detaljer om DNA-strengfoldningsbaner, " siger Yan.
Hans samarbejdspartnere på MIT, ledet af Mark Bathe, udviklet en computeralgoritme til at designe DNA-nanostrukturer ved kun at indtaste en målform. De konstruerede en softwareplatform, der kan beregne og udlæse nødvendige DNA-strenge for at danne designerarkitekturer. Dannelsen af disse strukturer blev derefter systematisk karakteriseret og bekræftet eksperimentelt på de tre institutter. "Dette viser virkelig tværfaglig kollaborativ videnskab over hele landet, " siger Yan
Yan leder Biodesign Center for Molecular Design and Biomimetics ved Arizona State University.
Andre verdener
Holdet designer nyttige strukturer i en forbløffende lille skala. (En nanometer er en milliardtedel af en meter eller omtrent på størrelse med et sukkermolekyle.) Specialiserede billeddannelsesteknikker, inklusive atomkraft- og kryo-elektronmikroskopi bruges til at visualisere de resulterende former.
Den beskrevne forenklede teknik lover at udvide brugen af DNA-origami betydeligt ud over specialistsamfundet og udvide rækken af mulige anvendelser inden for biomolekylær videnskab og nanoteknologi. Disse omfatter brugen af nanopartikler til lægemiddellevering og cellemålretning, konstruktion af robotter i nanoskala, der er i stand til at udføre forskellige aktiviteter inden for medicin og industri og design af skræddersyede optiske enheder.
En af de mere spændende innovationer i horisonten involverer brugen af DNA som et lagringsmedium - et med retentionstider i millioner af år. (Et enkelt gram DNA kan lagre omkring 700 terabyte information - en mængde svarende til 14, 000 50-gigabyte Blu-ray-diske. Yderligere, sådan nukleinsyrehukommelse kunne potentielt drives med en brøkdel af den energi, der kræves til andre informationslagringsmuligheder.)
Formskiftende
Den nye designmetode, som kan producere praktisk talt enhver polyedrisk form, er afhængig af en top-down strategi, som begynder med et omrids af den ønskede form og arbejder baglæns i etaper for at definere den nødvendige DNA-sekvens, der vil foldes korrekt for at danne det færdige produkt.
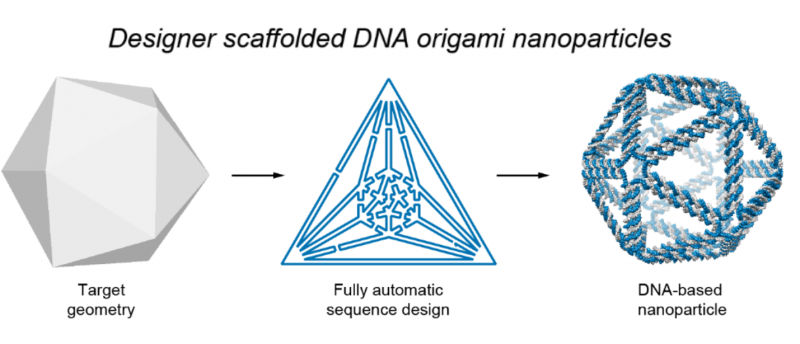
Illustration viser den grundlæggende proces, der bruges til at designe DNA origami -nanostrukturer. Først, der laves en wireframe af det tilsigtede måldesign. Softwaren oversætter derefter dette til en plan for routing af DNA-stilladser og hæftestrenge, som samles for at danne den ønskede form. Kredit:Biodesign Institute
Den autonome proces udføres ved hjælp af et softwareprogram designet af forfatterne. Kendt som DAEDALUS (for DNA Origami Sequence Design Algorithm for User-Defined Structures), programmet udfører omvendt design af vilkårlige DNA origami nanoformer, baseret på et input wireframe mesh, (en visuel fremstilling af den lukkede, 3-dimensionel geometrisk overflade).
Programmet er ikke kun brugervenligt, men meget alsidig, producerer former, der ikke er begrænset til sfærisk topologi, (dvs. lukket, tosidede strukturer uden grænser eller huller). Når målformularen er blevet beskrevet som et netværk af noder og kanter, DNA-stilladsstrenge af tilpasset længde og sekvens genereres ved hjælp af teknologi kendt som asymmetrisk polymerasekædereaktion.
Den nye undersøgelse beskriver fremstillingen af en række geometriske DNA-objekter, inklusive 35 polyedriske former (platonisk, Arkimedean, Johnson og catalanske faste stoffer) 6 asymmetriske strukturer, og fire polyedre med ikke-sfærisk topologi, ved hjælp af omvendte designprincipper. Metoden kan producere nanoformer med høj kvalitet og stabilitet uden den normale besværlige proces med manuelt at designe basepar til at danne den tilsigtede målstruktur.
Ind i folden
DNA-origami bringer den gamle japanske metode til papirfoldning ned til molekylær skala. Det grundlæggende er enkle:Tag en længde enkeltstrenget DNA og led det til en ønsket form, fastgørelse af strukturen ved hjælp af kortere såkaldte hæftestrenge, som binder sig på strategiske steder langs den længere længde af DNA. Metoden bygger på det faktum, at DNA's fire nukleotidbogstaver - A, T, C, &G holder sammen på en konsekvent måde; Som altid parring med Ts og Cs med Gs.
DNA-molekylet i sin karakteristiske dobbeltstrengede form er ret stift, sammenlignet med enkeltstrenget DNA, som er fleksibel. Af denne grund, enkeltstrenget DNA giver et ideelt blondelignende stilladsmateriale. Yderligere, dens parringsegenskaber er forudsigelige og konsistente, (i modsætning til RNA, som anses for promiskuøst, på grund af baseparringer, der kan være uventede).
Teknikken har vist sig vildt vellykket til at skabe utallige former i 2- og 3-dimensioner, som bekvemt samler sig selv, når de designede DNA-sekvenser blandes sammen. Den vanskelige del er at forberede den korrekte DNA-sekvens og routing-design til stilladser og hæftestrenge for at opnå den ønskede målstruktur. Typisk, dette er omhyggeligt arbejde, der skal udføres manuelt.
Med den nye teknik, målstrukturen er først beskrevet i form af et trådnet, der består af polyedre. Fra dette, en spændingstræ-algoritme genereres. Dette er dybest set et kort, der automatisk vil lede ruten af DNA-stilladsstrengen gennem hele origamistrukturen, berøring af hvert toppunkt i den geometriske form, enkelt gang. Derefter tildeles komplementære hæftestrenge, og den endelige form samles selv.
For at teste metoden, enklere former kendt som platoniske faste stoffer blev først fremstillet, efterfulgt af stadig mere komplekse strukturer. Disse omfattede objekter med ikke-sfæriske topologier og usædvanlige interne detaljer, som aldrig var blevet eksperimentelt realiseret før.
De færdige designs demonstrerede top-down-teknikkens evne til automatisk at generere stillads- og hæftningsruter til en ekspansiv række af nanoformer, udelukkende baseret på overfladegeometri. Cryo-EM blev brugt til at bekræfte strukturel troskab og stabilitet af de samlede origami-strukturer.
Yderligere eksperimenter bekræftede, at de producerede DNA-strukturer potentielt var egnede til biologiske anvendelser, da de udviste langtidsstabilitet i serum- og lav-saltforhold.
Forskningen baner vej for udviklingen af designede nanoskala -systemer, der efterligner virussens egenskaber, fotosyntetiske organismer og andre sofistikerede produkter af naturlig evolution.
Ud over sin ansættelse ved Biodesign Instituttet, Hao Yan er Milton D. Glick Distinguished Professor, College of Liberal Arts and Sciences, Skolen for Molekylær Videnskab ved ASU.
 Varme artikler
Varme artikler
-
 Bedre, dristigere print med silicium nanostrukturerKredit:American Chemical Society Fra lærebøger til kunst til aviser, tryksager er en del af vores hverdag. Men det blæk, der bruges i nutidens printere, er begrænset i farver og opløsning. Nu i en
Bedre, dristigere print med silicium nanostrukturerKredit:American Chemical Society Fra lærebøger til kunst til aviser, tryksager er en del af vores hverdag. Men det blæk, der bruges i nutidens printere, er begrænset i farver og opløsning. Nu i en -
 Kigger på atomare strukturer med ikke mere end blyant og papirKrystalstruktur af CoTiSb. (A) Uafslappet (001) plade af CoTiSb med TiSb-terminering. Den konventionelle bulk enhedscelle er markeret med stiplede linjer (kantlængde a), og (1 × 1) overfladeenhedscell
Kigger på atomare strukturer med ikke mere end blyant og papirKrystalstruktur af CoTiSb. (A) Uafslappet (001) plade af CoTiSb med TiSb-terminering. Den konventionelle bulk enhedscelle er markeret med stiplede linjer (kantlængde a), og (1 × 1) overfladeenhedscell -
 Flyt dig, silicium, der er et nyt kredsløb i byenHybride CNT/IGZO kredsløb fremstilles på en polyimidfilm lamineret på et polydimethylsiloxan (PDMS) substrat. Kredit:USC Viterbi, Chongwu Zhou (Phys.org) - Når det kommer til elektronik, silicium
Flyt dig, silicium, der er et nyt kredsløb i byenHybride CNT/IGZO kredsløb fremstilles på en polyimidfilm lamineret på et polydimethylsiloxan (PDMS) substrat. Kredit:USC Viterbi, Chongwu Zhou (Phys.org) - Når det kommer til elektronik, silicium -
 Atomisk uoverensstemmelse skaber nano-håndvægteDette billede kombinerer et transmissionselektronmikroskopbillede af en nanohåndvægt med et gulddomæne orienteret i retning. Frø- og gulddomænerne i håndvægten på billedet til højre identificeres ved
Atomisk uoverensstemmelse skaber nano-håndvægteDette billede kombinerer et transmissionselektronmikroskopbillede af en nanohåndvægt med et gulddomæne orienteret i retning. Frø- og gulddomænerne i håndvægten på billedet til højre identificeres ved
- Nitrogenforurenings vej til vandløb væver sig gennem flere skove (og hurtigere) end antaget
- Ring tilbyder sit billigste videodørklokkekamera til dato
- Bill henvender sig til Facebook, Google duopol over online annonceindtægter
- Laserpulser afslører fremtidens superledere
- Atomfejl skaber overraskende, højeffektive UV LED-materialer
- Supercomputerende indsats for at modellere de komplekse interaktioner, der påvirker klimaændringer…


