Cellulær mikroRNA-detektion med mirakler

Uddrag fra time-lapse-filmen af miRNA-detektion på 1 time ved hjælp af miRacles-assay. Kredit:Science Advances, doi:10.1126/sciadv.aau9443
MikroRNA'er (miRNA'er) er korte ikke-kodende regulatoriske RNA'er, der kan undertrykke genekspression post-transkriptionelt og bruges derfor i stigende grad som biomarkører for sygdom. Påvisning af miRNA'er kan være besværligt og dyrt, da de kræver amplifikation, mærkning og radioaktive sonder. I en nylig rapport offentliggjort den Videnskabens fremskridt , Arun Richard Chandrasekaran og medarbejdere ved RNA Instituttet og Institut for Biologiske Videnskaber, ved University of Albany, State University of New York, rapporteret på et enkelt trin, ikke-enzym mikroRNA detektionsassay ved hjælp af konformationelt ikke-responsive DNA nanoswitches.
Forskerne kaldte analysen 'mirakler, ' for at forkorte 'mikro-RNA aktiveret betinget looping af konstruerede switches'. Assayet demonstrerede subattomol- og enkeltnukleotidspecificitet ved anvendelse af en agarosegelelektroforeseaflæsning. I forsøgene, de påviste cellulære mikroRNA'er fra RNA-ekstraktioner opnået fra differentierende muskler på nanogram-skalaen. Forskerne præsenterede et omkostningseffektivt eksperimentelt setup til at detektere miRNA'er inden for en times tidsramme for at give et overbevisende alternativ til de eksisterende metoder til kvantitativ polymerasekædereaktion (qPCR) og Northern blotting for at kvantificere de regulatoriske genetiske materialer.
miRNA'er kan regulere mange biologiske processer under normal fysiologisk udvikling og sygdom ved at påvirke celleproliferation, differentiering og apoptose in vivo. Ekspressionen af miRNA'er kan kvantificeres i væv, celler og kropsvæsker som stabile biomarkører for cellulære hændelser og sygdomsdiagnose, fremhæve vigtigheden af deres opdagelse. Alligevel, miRNA-detektion er udfordrende på grund af lav overflod, lille størrelse og ligheder i rækkefølge. Biomolekylerne udgør cirka 0,01 procent af det totale RNA-indhold, og individuelle mRNA-kopier pr. celleområde kan variere meget. Derudover miRNA'er i en familie kan variere med et enkelt nukleotid, mens specifikke miRNA'er kan reguleres under sygdom og regelmæssig cellulær funktion. Som resultat, påvisningsstrategierne for miRNA kræver høj specificitet og evnen til korrekt at identificere nogle få molekyler fra en prøve, der er rigeligt med dominerende RNA-molekyler.
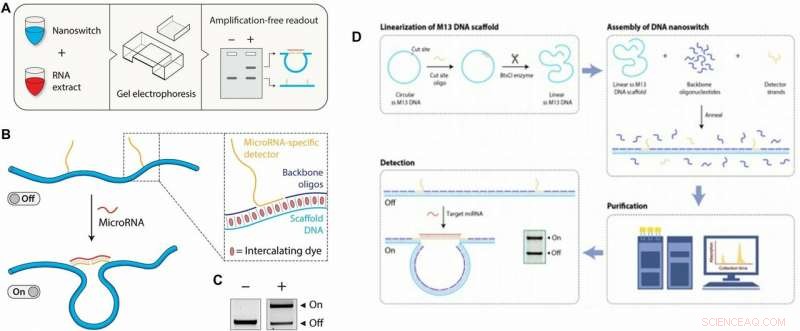
Til venstre:Koncept og arbejdsgang for miracles-analysen. (A) Workflow for miRacles-analysen:Tilpassede DNA-nanoswitches blandes med mål-miRNA-prøve, inkuberet, og kør på en agarosegel til påvisning. (B) DNA nanoswitches gennemgår en konformationel ændring fra en lineær "slukket" tilstand til en sløjfet "on" tilstand, når de er bundet til et mål miRNA. Indsat:Nanoswitchen er sammensat af et enkeltstrenget M13 stillads, oligonukleotider i rygraden, og enkeltstrengede forlængelser (detektorer) komplementære til target miRNA'et. Interkalerende farvestoffer, der er iboende til elektroforeseprocessen, giver signalet til at visualisere nanoswitchene. (C) De to konformationer er opløselige i en standard agarosegel. Til højre:Dybdegående undersøgelse af DNA-nanoswitch-konstruktionsprocessen. (D) Linearisering:Fremstilling af et lineært enkeltstrenget M13-stillads fortsætter ved at anneale et oligonukleotid for at muliggøre spaltning på det dobbeltstrengede sted med et restriktionsenzym (BtsCI i dette tilfælde). Samling:DNA nanoswitch-samling udføres ved at blande et 10x molært overskud af rygradoligonukleotider og detektorstrenge (specifikke for målet) til den enkeltstrengede M13 og påføre en kølerampe fra 90˚C til 25˚C ved 1˚C/min. . Oprensning:For at fjerne overskydende oligonukleotider, der anvendes en væskekromatografimetode. Registrering:For at bruge nanoswitchene, de blandes med en opløsning indeholdende mikroRNA-mål og køres på en gel for at løse tænd- og sluktilstandene som vist i det indsatte. Kredit:Science Advances, doi:10.1126/sciadv.aau9443
Traditionelle metoder til påvisning af miRNA omfatter Northern blotting, kvantitativ revers-transkriptionspolymerasekædereaktion (qRT-PCR), næste generations sekventering og mikroarray-baseret hybridisering for at adskille miRNA-signalet fra støj. Af de angivne metoder, Northern blotting kan direkte identificere native miRNA'er, mens andre er afhængige af yderligere mærkningsmetoder eller trinvis amplifikation, øger omkostningerne, kompleksitet og ydelse af detektion. For eksempel, innovative DNA-nanostrukturer kan bruges til miRNA-detektion, hvor forskellige forskningsgrupper tidligere har kombineret nanostrukturer med nanopartikler, hybridiseringskædereaktioner og overgangsmetal dichalcogenid nanoplader for at muliggøre processen.
I nærværende arbejde, Chandrasekaran et al. udviklet et relativt simpelt DNA-baseret apparat til at løse en kompleks biomedicinsk udfordring. I miracles assay, forskerne brugte et 'smart reagens' sammensat af rationelt designede DNA-nanoswitches til enkel og omkostningseffektiv native miRNA-detektion uden brug af specialudstyr i laboratoriet. DNA nanoswitches blev oprindeligt designet som værktøjer til enkeltmolekyle biofysiske eksperimenter og senere anerkendt for deres evne til at detektere og kvantificere biomolekylære interaktioner ved hjælp af gelelektroforese. Tidligere forskningssamarbejde fra det samme forskerhold fokuserede på molekylær detektion for at kvantificere proteinniveauer og detektere syntetiske DNA-sekvenser som et proof-of-concept.
Det nuværende arbejde udvidede på foreløbige undersøgelser og koncepter for at producere brugerklar multiplekset miRNA-detektion og kvantificering. Forskerne analyserede nanogram af cellulære RNA-ekstrakter i en kort tidsramme ved hjælp af en eksperimentel opsætning bygget ved hjælp af almindelige laboratorieforsyninger. De designede DNA-nanoswitchen som en lineær dupleks, der dannede en løkke i nærvær af mål-miRNA-molekylet. For at konstruere nanoswitchen, Chandrasekaran et al. brugte DNA-origami-tilgangen ved at hybridisere korte oligonukleotider komplementære til et enkeltstrenget DNA-stillads.
De designede to fjerne "detektor"-strenge med udhæng, der var komplementære til forskellige segmenter af mål-miRNA'et. Når miRNA'et genkendte og bandt til konstruktionen, kontakten rekonfigureret fra den lineære "slukket"-tilstand til den sløjfede "til"-tilstand. De kvantificerede de to tilstande ved hjælp af standard agarosegelelektroforese for at detektere signalet, der stammer fra den sløjfede nanoswitch. Signalet blev kun forstærket af et enkelt miRNA af interesse, resultaterne sammenlignet positivt med fluorescensresonansenergioverførselsteknikken (FRET).

Validering af miRacles-analysen. (A) Specificitet af DNA nanoswitches med detektorer designet til let-7b. Så lavt som 1-nt mismatch mellem detektorerne og målet miRNA eliminerer signalet. (B) Grænse for detektion af assayet. NC, negativ kontrol. AU, vilkårlige enheder. (C) Tidsforløb for analysen for et lavkoncentrationsmål. (D) Dynamisk område af analysen ved forskellige reaktionstider. Kredit:Science Advances, doi:10.1126/sciadv.aau9443.
For at validere konceptet, holdet af forskere valgte et let-7b-mål miRNA på grund af dets meget konserverede familie på mere end et dusin-relaterede miRNA'er, der varierede med et eller flere nukleotider. Disse miRNA'er var velegnede på grund af deres kritiske rolle i biologiske funktioner og dysregulering i flere menneskelige sygdomme. For at eliminere krydstale og støj mellem nanoswitchen og målet - hvilket førte til reduceret signalintensitet sammenlignet med et perfekt match, forskerne redesignede rationelt nanoswitchene. For at opnå perfekt specificitet, de destabiliserede interaktionen på den side, der indeholdt uoverensstemmelsen. Resultaterne af undersøgelsen illustrerede høj specificitet af det således udviklede assay, giver et svar på en nøgleudfordring i miRNA-detektion, der kulminerede fra et højt signal/støj-forhold.
Den lave overflod af miRNA'er krævede også høj påvisningsfølsomhed, hvilket forskerne opnåede ved at optimere protokollen. De udførte derefter lignende eksperimenter for to andre varianter af miRNA'er (miR-15 a og miR-206), resulterer i subattomol til enkelt-attomol niveauer af påvisning. For eksempel, for en lav koncentration af en målprøve (6 pM), signalet steg i 4 timer, med få ændringer ud over denne tidsramme.
miRNA-detektion fra differentierende myoblastceller. (A) Skematisk, der viser myoblastceller, høstet under dyrkning i GM og på differentieringsdag 1 og 4, behandlet til at give totale og små RNA-fraktioner. En tidlig myogen differentieringsmarkør, Myog, og en sen myogen differentieringsmarkør, MHC, blev målt ved (B) Western blotting og (C) ved immuncytokemi for at bekræfte differentiering. Både Myog og MHC blev opreguleret i DM1 og DM4. GAPDH (glyceraldehyd-3-phosphatdehydrogenase) tjente som kontrol i (B). (D) Chandrasekaran et al. detekterede miR-206 i de differentierede prøver med 50 ng små RNA'er og (E) med 500 ng totale RNA'er. Kvantificering af gelintensiteter viser en skarp progressiv opregulering under differentiering, ens i både (F) små RNA og (G) total RNA prøver. Fra DM4 prøver, vi bemærker påvisning fra så lidt som (H) 200 pg små RNA'er og (I) 500 pg totale RNA'er. Kredit:Science Advances, doi:10.1126/sciadv.aau9443. 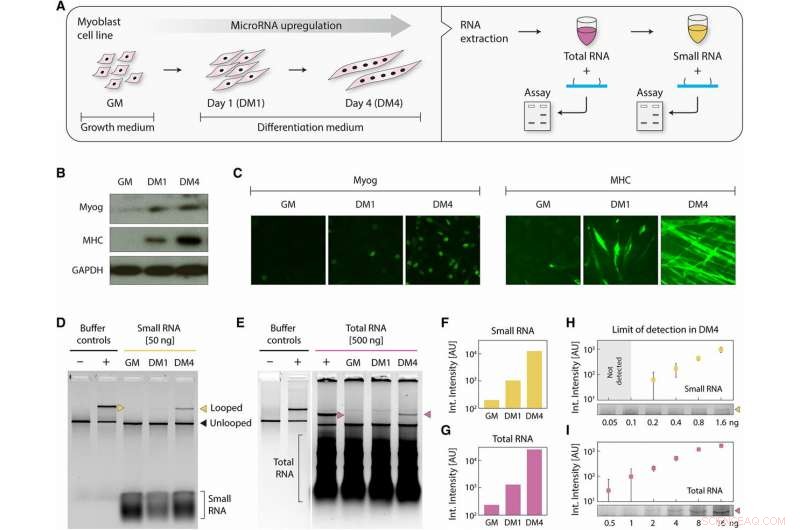
Da flere mRNA'er også kan ændre deres ekspression under forskellige cellulære eller sygdomsstadier, fænomenet krævede yderligere detektionsmuligheder. For at opnå dette i den eksperimentelle opsætning, Chandrasekaran et al. brugte programmerbarheden af nanoswitches og udviklede et multipleksingssystem, der er i stand til at detektere flere miRNA'er fra den samme prøve. De placerede detektorstrengene på de ønskede steder i DNA-stilladset, hvilket resulterer i loops af forskellig størrelse, når de er bundet til target miRNA'et. Sløjfestørrelsen af nanoswitchen bestemte således gelmigreringen, resulterer i et unikt bånd på gelen til nøjagtig detektion. Til eksperimentet, forskerne valgte fire miRNA'er til stede i muskelceller; miR-206, miR-125b, miR-24 og miR-133-3p og en negativ kontrol miRNA; miR-39 specifikt for arten Caenorhabditis elegans.
Inden for 50 ng af små RNA'er, forskerne opdagede de fire miRNA'er på forskellige ekspressionsniveauer, mens den bekræfter ingen detektering med den negative kontrol. Multiplexing-strategien gjorde det muligt for forskerne direkte at sammenligne miRNA-niveauer i en enkelt prøve, uden mærkning eller amplifikation. I alt, arbejdet gav et yderligere skridt i retning af at udvide gennemstrømningen af miRacles-analysen. Muligheden kan udvides til også at rumme flere miRNA'er pr. switch.
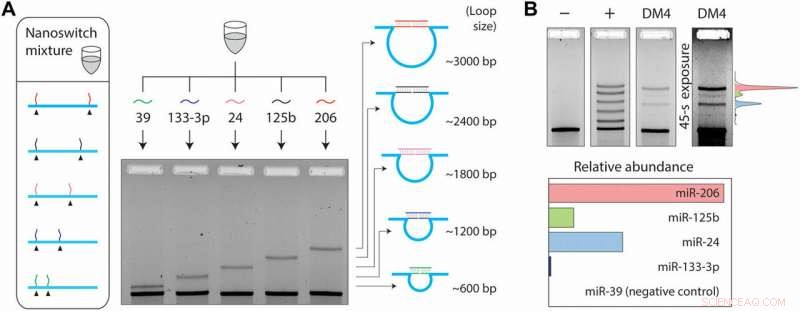
Fem-kanals multipleksing opsætning. (A) Multipleksing muliggør påvisning af forskellige miRNA'er med forskellige løkkestørrelser. (B) En multiplekset nanoswitch-blanding viser fem bånd med lignende intensitet i en positiv kontrol bestående af alle fem mål miRNA'er. I 50 ng DM4 små RNA'er, fire forskellige miRNA'er detekteres ved forskellige ekspressionsniveauer, med miR-39 (et C. elegans-specifikt miRNA) ikke påvist.
På denne måde Chandrasekaran et al. væsentligt bevæget sig fremad fra deres foreløbige proof-of-concept-detektion af syntetiske DNA-sekvenser; at etablere, karakterisere og optimere et brugsklart miRNA-detektionsassay med biologiske ekstrakter. Det demonstrerede arbejde var et første eksempel på at bruge DNA nanoswitches til at detektere miRNA'er fra en ægte biologisk prøve. Mens ydeevnen af miRNA-analysen var konkurrencedygtig sammenlignet med andre almindeligt anvendte teknikker, selektiviteten af 1 nukleotid set i det foreliggende arbejde var vanskelig at opnå med eksisterende metoder. Følsomheden af mirakler overgik også Northern blotting og mikroarrays. Analysen kunne måle miRNA'er uden behov for amplifikation, med enklere protokoller og uden den tilføjede fejl ved ekstra prøvebehandling. Protokollen blandede simpelthen nanoswitchene med prøvevæsken til gelelektroforese, at producere resultater af høj kvalitet i laboratoriet. Forskningsarbejdet kan potentielt overføres fra biologiske til kliniske prøver for at diagnosticere og overvåge sygdom.
Vigtigere, nærværende arbejde er på linje med det bredere begreb sparsommelig videnskab; en lovende vision om omkostningseffektiv videnskab, der allerede har produceret billige løsninger til blodcentrifugering og vandrensningsteknikker inden for biomedicinsk teknik. Chandrasekaran et al. sigter mod fortsat at bidrage til den nye tendens inden for videnskab, ved at forstyrre det eksisterende omkostnings-/ydelsesforhold for at give bred skala adgang til simple miRNA-detektionsmetoder med bland-og-læs smarte reagenser.
© 2019 Science X Network
Sidste artikelNanomaterialer giver planter superevner
Næste artikelPolyU udvikler elektrostatisk ladet nanofiber til luftbåren filtrering
 Varme artikler
Varme artikler
-
 Observerer elektroner, der surfer på lysbølger på grafenElektroner og lys bevæger sig sammen langs grafenarket. Kredit:ICFO/ F. Vialla Forskere har undersøgt, hvordan lys kan bruges til at observere et elektronisk materiales kvantenatur. De fangede lys
Observerer elektroner, der surfer på lysbølger på grafenElektroner og lys bevæger sig sammen langs grafenarket. Kredit:ICFO/ F. Vialla Forskere har undersøgt, hvordan lys kan bruges til at observere et elektronisk materiales kvantenatur. De fangede lys -
 Fremstilling af fejlfri grafenbelægningerKredit:AlexanderAlUS/Wikipedia/CC BY-SA 3.0 grafen, det ultratynde vidundermateriale kun et enkelt kulstofatom i tykkelse, har løftet om så imponerende applikationer som slidstærke, friktionsfri b
Fremstilling af fejlfri grafenbelægningerKredit:AlexanderAlUS/Wikipedia/CC BY-SA 3.0 grafen, det ultratynde vidundermateriale kun et enkelt kulstofatom i tykkelse, har løftet om så imponerende applikationer som slidstærke, friktionsfri b -
 Nano-indkapsling til effektiv levering i Parkinsons behandlingKredit:Autonomous University of Barcelona Parkinsons sygdom (PD) er en almindelig neurodegenerativ lidelse forårsaget af dopaminerge neuroners død i en del af hjernen (kendt som substantia nigra p
Nano-indkapsling til effektiv levering i Parkinsons behandlingKredit:Autonomous University of Barcelona Parkinsons sygdom (PD) er en almindelig neurodegenerativ lidelse forårsaget af dopaminerge neuroners død i en del af hjernen (kendt som substantia nigra p -
 Forskere bygger en nanocage med antiaromatiske vægge(a) konstruktion af antiaromatisk-væggede nanospace. (b) røntgenkrystalstruktur med et 3D NICS-gitter, viser magnetisk afskærmning oplevet i nanorummet. Antiaromatiske virkninger bliver stærkere i stø
Forskere bygger en nanocage med antiaromatiske vægge(a) konstruktion af antiaromatisk-væggede nanospace. (b) røntgenkrystalstruktur med et 3D NICS-gitter, viser magnetisk afskærmning oplevet i nanorummet. Antiaromatiske virkninger bliver stærkere i stø
- Byer som Lagos har brug for bygningsdesign, der ikke bare kopierer globale stilarter
- Forskere introducerer en kosmokemisk model for Pluto-dannelse
- Akademiker siger, at han bliver syndebuk i Facebook-datasag (opdatering)
- Sådan finder du X & Y-skæringer på en grafregner
- Unikke syntetiske antistoffer viser løfte om forbedret opdagelse af sygdomme og toksiner
- En simpel mekanisme kunne have været afgørende for livets udvikling


