Ny teknik forbedrer proteoform-billeddannelse i humant væv
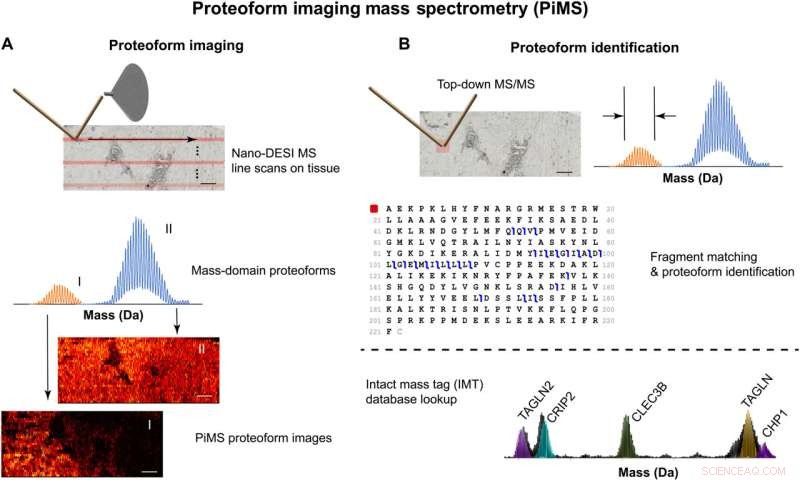
Illustration af PiMS workflow til proteoform billeddannelse og identifikation. (A) Scanningstilgang (øverst), påvisning af proteoformer i massedomænet (midten) og billedrekonstruktion (nederst). (B) To tilgange til at identificere proteoformer ved hjælp af enten direkte fragmentering af proteoformioner og spektral udlæsning ved individuelle ion MS/MS (øverst) eller databaseopslag af nøjagtige masseværdier (IMT, nederst). Skala barer, 1 mm. Kredit:Science Advances (2022). DOI:10.1126/sciadv.abp9929
Efterforskere ledet af Neil Kelleher, Ph.D., professor i medicin i afdelingen for hæmatologi og onkologi og for biokemi og molekylær genetik, har udviklet en ny billedbehandlingsteknik, der firedobler påvisningen af intakte proteoformer sammenlignet med nuværende proteinbilleddannelsesmetoder .
Billedteknikken, beskrevet i et nyligt papir offentliggjort i Science Advances , giver høj opløsning, high-throughput billeddannelse af proteoformer eller alle modificerede versioner af proteiner. Det er vigtigt, at teknikken er "mærkefri", kræver ikke antistoffer og kan identificere hele proteoformer direkte fra ethvert ufikseret væv. Teknikken kan i øjeblikket detektere omkring 1.000 proteoformer og lokaliserer proteoformer med en rumlig opløsning på 40 til 70 mikron.
Adskillige teknikker bruges almindeligvis til at afbilde proteiner i humant væv, men meget få er i stand til at afbilde proteoformer. Dem, der kan afbilde hele proteoformer, gør det ved at adskille proteoformen fra væv og ionisere dem til massespektrometri. Disse teknikker tilbyder imidlertid lav molekylær specificitet.
For at løse dette problem udviklede Kellehers team proteoform billeddannende massespektrometri (PiMS). Teknikken fungerer ved at udtrække proteoformer fra vævet med nanodråber, "veje" de ekstraherede proteoformer for at identificere dem og derefter bruge disse data til at konstruere proteoformbilleder af det scannede væv.
"Den virkelige innovation med PiMS er, at den kobler en robust eksisterende teknik til at udvinde og ionisere proteoformer, nanoDESI, med en banebrydende teknologi til individuel ionmassespektrometri, der blev opfundet i fællesskab af Thermo Fisher Scientific og Northwestern Proteomics. Sammenlignet med almindelige detektionsteknikker, individuel ionmassespektrometri tilbyder op til 500 gange mere følsomhed og 20 gange mere opløsningsevne. Det øger teknikkens kraft betydeligt, og PiMS registrerer større, sjældnere proteoformer og udvider i høj grad grænserne for proteomdækning," sagde Kelleher, der også er direktør for Northwesterns Proteomics Center of Excellence, Robert H. Lurie Comprehensive Cancer Centers Proteomics Core Facility og Northwesterns Chemistry of Life Processes Institute.
For at demonstrere PiMS' evner brugte Kellehers team teknikken til at afbilde proteoformer fra funktionelle enheder af den menneskelige nyre. Disse billeder afslørede distinkte rumlige lokaliseringer af proteoformer fra forskellige anatomiske regioner og funktionelle vævsenheder som nyrebarken versus medulla.
PiMS' øgede proteomdækning åbner også døren for bredere anvendelser inden for kortlægning af molekylært væv, identifikation af nye biomarkører og forbedring af sygdomsdiagnose, ifølge Kelleher.
"For nylig har der været et stort fremstød inden for genomik og proteomik for enkeltcellebiologi:for bedre at fange sygdommes heterogenitet ved at bruge rumlige eller enkeltcellede tilgange, der bevarer de mange forskellige signaler i stedet for bulktilgange, der blander alle celletyper og Den rumlige tilgang i særdeleshed tilføjer en langt større præcision til protein-billeddannelse, og vi skubber i øjeblikket på det for at identificere tusindvis af proteoformer med enkeltcellet opløsning," sagde Kelleher. + Udforsk yderligere
Human proteoform projekt til kortlægning af proteiner i menneskekroppen
 Varme artikler
Varme artikler
-
 Hvad fossiler afslører om hybridisering af tidlige menneskerTidlig moderne menneske (venstre) og neandertaler. Kredit:Kunstnerisk illustration:Gleiver Prieto; Copyright:K. Harvati Mange mennesker, der lever i dag, har en lille komponent af neandertaler-DNA
Hvad fossiler afslører om hybridisering af tidlige menneskerTidlig moderne menneske (venstre) og neandertaler. Kredit:Kunstnerisk illustration:Gleiver Prieto; Copyright:K. Harvati Mange mennesker, der lever i dag, har en lille komponent af neandertaler-DNA -
 5 sande historier om tvillinger adskilt ved fødslenHej vent, hvor skal du hen? Alex Linghorn/Photodisc/Getty Images Tvillinger, af deres meget to-til-en karakter, er et historisk og videnskabeligt fascinerende fænomen. Start med, sige, Kain og Abel,
5 sande historier om tvillinger adskilt ved fødslenHej vent, hvor skal du hen? Alex Linghorn/Photodisc/Getty Images Tvillinger, af deres meget to-til-en karakter, er et historisk og videnskabeligt fascinerende fænomen. Start med, sige, Kain og Abel, -
 Unicorn Root genopliver sig selv efter 130 årEnhjørning rod, kendt for de hvide blomsterstilke, der formodentlig giver den sit navn, dukkede op igen i Maine i sommeren 2018 efter et fravær på 130 år. Maine Natural Areas Program Enhjørning rod (
Unicorn Root genopliver sig selv efter 130 årEnhjørning rod, kendt for de hvide blomsterstilke, der formodentlig giver den sit navn, dukkede op igen i Maine i sommeren 2018 efter et fravær på 130 år. Maine Natural Areas Program Enhjørning rod ( -
 Genomisk undersøgelse afslører spor til druernes vilde fortidDataene indikerer, at mennesker har samlet druer i naturen i århundreder, før de dyrkede dem, siger studieleder Brandon Gaut, UCI professor i økologi og evolutionær biologi. Forskningen afslørede også
Genomisk undersøgelse afslører spor til druernes vilde fortidDataene indikerer, at mennesker har samlet druer i naturen i århundreder, før de dyrkede dem, siger studieleder Brandon Gaut, UCI professor i økologi og evolutionær biologi. Forskningen afslørede også
- Discovery kunne reducere nukleart affald med en forbedret metode til kemisk konstruktion af molekyle…
- Egenskaber ved et lineært programmeringsproblem
- Lyse områder på Ceres tyder på geologisk aktivitet
- Nyt værktøj måler atomare skala defekter, identificerer transistorbegrænsninger
- Spin-polariseret overfladetilstande i superledere
- Kuldioxidniveauer højest i 3 millioner år, viser en hidtil uset computersimulering


