Forskere lokker proteiner til at danne syntetiske strukturer med en metode, der efterligner naturen
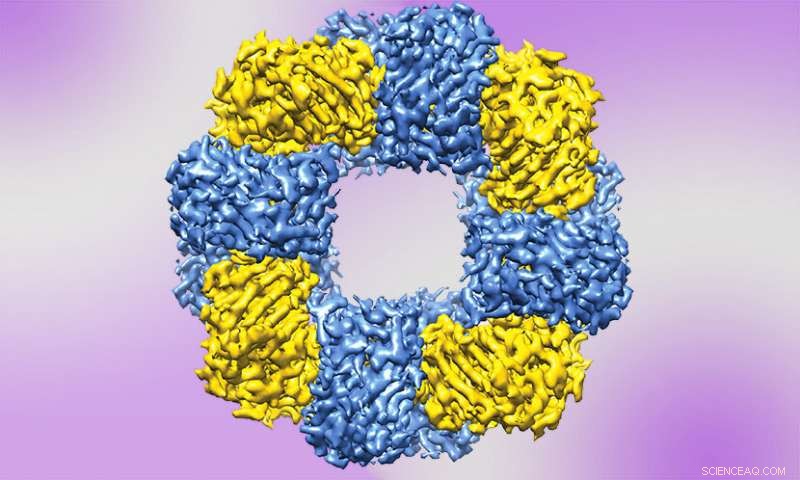
Som et bevis på konceptet, et team af forskere ved University of Texas i Austin byggede bittesmå strukturer, der ligner to donuts stablet oven på hinanden ved at påføre elektriske ladninger på specifikke pletter på naturligt forekommende proteiner. Kredit:University of Texas i Austin
Forskere har længe drømt om at skabe syntetiske strukturer ud af det samme råmateriale, som naturen bruger i levende systemer - proteiner - og troede, at et sådant fremskridt ville give mulighed for udvikling af transformative nanomaskiner, for eksempel, molekylære bure, der præcist leverer kemoterapimedicin til tumorer eller fotosyntetiske systemer til at høste energi fra lys. Nu har et team af biologer fra University of Texas i Austin og University of Michigan opfundet en måde at bygge syntetiske strukturer ud fra proteiner, og ligesom i naturen, metoden er enkel og kan bruges til mange forskellige formål.
"Vi tror, vi kan bruge disse strukturer ligesom Legos til at bygge større ting, " sagde David Taylor, assisterende professor i molekylær biovidenskab ved UT Austin og co-korresponderende forfatter på et nyt papir offentliggjort i dag i tidsskriftet Naturkemi . "Vi kender også nogle af reglerne for at ændre grundopskriften til at lave forskellige typer byggeklodser."
Som et bevis på konceptet, holdet byggede små strukturer, der ligner to donuts stablet oven på hinanden ved at påføre elektriske ladninger på specifikke pletter på naturligt forekommende proteiner. Tidligere forskere har formået at skabe syntetiske strukturer fra proteiner, men først efter omhyggeligt at have påført noget på proteiner eller skabt nye proteiner fra bunden, gør tidligere metoder komplicerede, tidskrævende og begrænsende. Derimod den nye metode, kaldet "SUpercharged PROtein Assembly (SuPrA), " efterligner den måde, proteiner i levende organismer fungerer på, når de laver de molekylære maskiner, der udfører livets forskellige funktioner:Strukturerne i den nye metode er selvsamlende og fleksible.
"Vores tilgang tager et protein, der normalt ikke samles, og giver det mange potentielle websteder, hvor det måske er i stand til, tillader den at 'vælge', som passer bedst til resten af dens geometri og kemi, sagde Anna Simon, en postdoktor i UT Austins Institut for Molekylær Biovidenskab og medforfatter af papiret. "Dette er vigtigt, fordi det giver os en måde at semi-direkte proteiner til at organisere sig i større strukturer uden på forhånd at skulle forstå, præcis hvordan de vil passe sammen."
Det originale koncept for denne nye metode blev udviklet af Andy Ellington, associeret direktør for UT Austins Center for Systemer og Syntetisk Biologi, også en professor i molekylær biovidenskab og den medforfatter på undersøgelsen.
For at demonstrere deres koncept, forskerne startede med grønt fluorescerende protein, et standardprotein, der bruges som et glødende mærke i alle mulige biologiske eksperimenter. De lavede to lidt forskellige versioner, ved at bruge en aldrig-før-forsøgt metode:tilføjelse af elektriske ladninger for at lokke proteinet til at danne diskrete, symmetriske strukturer. En version havde positive ladninger tilføjet på visse steder, og det blev blandet i en opløsning med en anden version, der havde negative ladninger på bestemte steder. Holdet fandt hver version selvsamlet i utallige små strukturer, eller makromolekylære komplekser, hver med det samme antal og arrangement af proteiner.
Fordi denne metode gør det muligt at bygge strukturer ud fra naturligt forekommende proteiner, forskerne siger, at det tilbyder videnskaben et nyt værktøj, der er skalerbart, overkommelig og bæredygtig.
"Det er ligesom, hvordan folk bruger 3-D-printere til at lave ting ud af materialer, de ikke ville have brugt tidligere, "Taylor sagde." Denne nye metode giver os en anden mulighed for materialer. Disse materialer er let tilgængelige, billig og ikke skadelig for miljøet."
Sidste artikelForskere får kontrol over syntesen af bløde molekyler
Næste artikelGå på pisterne med biobaserede ski og snowboards
 Varme artikler
Varme artikler
-
 PoreDesigner forbedrer proteinkanaldesign til vandbehandling, bioseparationerFigur, der skitserer det grundlæggende i PoreDesigners arbejdsgang til ændring af porestørrelsen af et bakteriekanalprotein. Kredit:Manish Kumar / Penn State PoreDesigner, en fuldt automatiseret
PoreDesigner forbedrer proteinkanaldesign til vandbehandling, bioseparationerFigur, der skitserer det grundlæggende i PoreDesigners arbejdsgang til ændring af porestørrelsen af et bakteriekanalprotein. Kredit:Manish Kumar / Penn State PoreDesigner, en fuldt automatiseret -
 Kraftigt tidligt lys af LED'erForskere bruger supercomputere til at få indsigt i nye materialer, der kan gøre LED-belysning endnu lysere og mere overkommelig. Der er fundet nye egenskaber i kubiske III-nitrider LED-materialer, der
Kraftigt tidligt lys af LED'erForskere bruger supercomputere til at få indsigt i nye materialer, der kan gøre LED-belysning endnu lysere og mere overkommelig. Der er fundet nye egenskaber i kubiske III-nitrider LED-materialer, der -
 Mikrober kunne gøre medicinproduktionen mere effektivAlkaloidbaserede lægemidler afledt af planter kan være potente behandlinger for en række forskellige sygdomme. Men at få disse kraftfulde terapeutiske midler fra planter kan tage lang tid og koste man
Mikrober kunne gøre medicinproduktionen mere effektivAlkaloidbaserede lægemidler afledt af planter kan være potente behandlinger for en række forskellige sygdomme. Men at få disse kraftfulde terapeutiske midler fra planter kan tage lang tid og koste man -
 Sådan beregnes den første ioniseringsenergi af hydrogenatomet relateret til Balmer SeriesBalmer serien er betegnelsen for de spektrale emissioner fra brintatom. Disse spektrale linier (som er fotoner udsendt i det synlige lysspektrum) er produceret fra den energi, der kræves for at fjerne
Sådan beregnes den første ioniseringsenergi af hydrogenatomet relateret til Balmer SeriesBalmer serien er betegnelsen for de spektrale emissioner fra brintatom. Disse spektrale linier (som er fotoner udsendt i det synlige lysspektrum) er produceret fra den energi, der kræves for at fjerne
- Hvordan begrænser begrænset uddannelse unge?
- SpaceX opsender kommunikationssatellit, grøfter gammel booster
- Egypten afslører gamle kister, statuer fundet i Saqqara
- Særlig levering:Makromolekyler via edderkopper bider
- Storbritannien bøder Facebook på grund af skandale om databeskyttelse, EU søger revision
- Både Sn- og Zn-enkeltatomer på CuO-katalysator fremmer synergistisk dimethyldichlorsilansyntese


