Nyt stofbibliotek for at fremskynde søgningen efter aktive stoffer
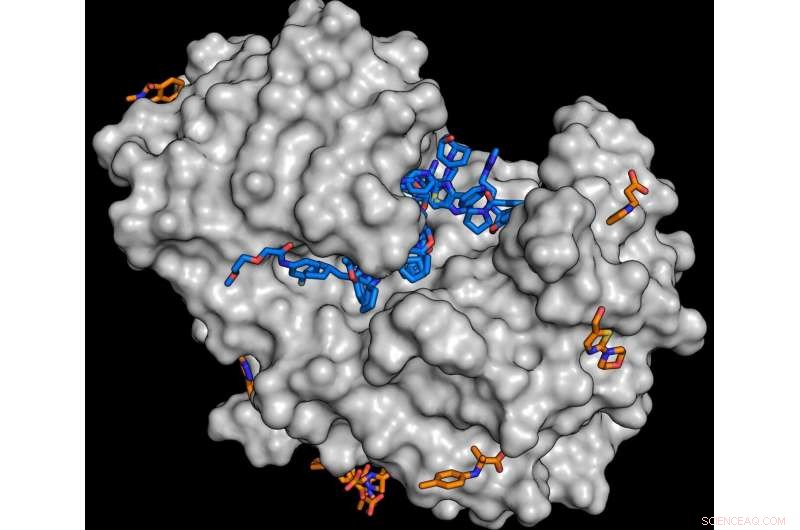
Til studiet, enzymet endothiapepsin (grå) blev kombineret med molekyler fra fragmentbiblioteket. Analysen viser, at adskillige stoffer er i stand til at koble sig til enzymet (blå og orange molekyler). Hvert stof fundet er et potentielt udgangspunkt for udviklingen af større molekyler. Kredit:J. Wollenhaupt/HZB
For at fremskynde den systematiske udvikling af lægemidler, MX-teamet på Helmholtz-Zentrum Berlin (HZB) og Drug Design Group på University of Marburg har etableret et nyt stofbibliotek. Det består af 1103 organiske molekyler, der kunne bruges som byggesten til nye lægemidler. MX-teamet har nu valideret dette bibliotek i samarbejde med FragMAX-gruppen på MAX IV. HZB's stofbibliotek er tilgængeligt for forskning over hele verden og spiller også en rolle i søgningen efter stoffer, der er aktive mod SARS-CoV-19.
For at lægemidler skal være effektive, de er normalt nødt til at koble til proteiner i en organisme. Som en nøgle i en lås, en del af lægemiddelmolekylet skal passe ind i fordybninger eller hulrum i målproteinet. I flere år nu, teamet fra den makromolekylære krystallografiafdeling (MX) ved HZB ledet af Dr. Manfred Weiss sammen med Drug Design Group ledet af prof. Gerhard Klebe (University of Marburg) har derfor arbejdet på at opbygge såkaldte fragmentbiblioteker. Disse består af små organiske molekyler (fragmenter), med hvilke de funktionelt vigtige hulrum på overfladen af proteiner kan sonderes og kortlægges. Proteinkrystaller mættes med fragmenterne og analyseres derefter ved hjælp af kraftigt røntgenlys. Dette gør det muligt at opnå tredimensionel strukturel information ved niveauer af atomare opløsning. Blandt andet, det er muligt at finde ud af, hvor godt et specifikt molekylefragment kobler sig til målproteinet. Udviklingen af disse stofbiblioteker fandt sted som en del af det fælles Frag4Lead-forskningsprojekt og blev finansieret af det tyske forbundsministerium for uddannelse og forskning (BMBF).
MX-teamet (MX står for Macromolecular Crystallography) har nu offentliggjort designet af et kemisk mangfoldigt fragmentbibliotek kaldet "F2X-Universal" biblioteket, som består af 1, 103 forbindelser. Et repræsentativt udvalg af 96 forbindelser er blevet ekstraheret fra dette bibliotek, som omtales som F2X Entry Screen. I forbindelse med udgivelsen af biblioteket, dette valg er nu blevet testet og valideret af MX-teamet fra HZB ved MAX IV røntgenkilden i Lund, Sverige og på BESSY II.
I undersøgelsen, HZB- og MAX IV-holdene verificerede effektiviteten af F2X Entry-biblioteket ved at screene endothiapepsin og Aar2/RnaseH-proteinkomplekset som målenzymer. I næste trin, MX-teamet vil bruge hele det universelle bibliotek.
"For den nuværende undersøgelse, eksperterne i fragmentscreening hos HZB—BESSY II arbejdede meget tæt sammen med FragMAX-projektteamet på MAX IV", sagde Dr. Uwe Müller fra MX-teamet hos HZB, som hjalp med at opsætte de tre MX-strålelinjer ved BESSY II samt BioMAX-strålelinien ved MAX IV. "Dette gjorde det muligt for begge partnere at videreudvikle deres egne teknologiplatforme og bruge dem til billeddannelse af de funktionelle overflader af forskellige proteiner. Dette vil være et glimrende grundlag for fremtidigt samarbejde mellem MAX IV og HZB."
 Varme artikler
Varme artikler
-
 Ny tilgang bestemmer optimale materialedesign med minimale dataKredit:Northwestern University Northwestern University-forskere har udviklet en ny beregningsmæssig tilgang til at accelerere designet af materialer, der udviser metal-isolator overgange (MIT), en
Ny tilgang bestemmer optimale materialedesign med minimale dataKredit:Northwestern University Northwestern University-forskere har udviklet en ny beregningsmæssig tilgang til at accelerere designet af materialer, der udviser metal-isolator overgange (MIT), en -
 Mikrobølgeovn plast øger lithium-svovl batteriets levetidForskere har opdaget, at iblødsætning af plast med lav densitet i et svovlholdigt opløsningsmiddel, at sætte det i en mikrobølgeovn og omdanne det til et kulstofstillads gør, at lithium-svovl-batterie
Mikrobølgeovn plast øger lithium-svovl batteriets levetidForskere har opdaget, at iblødsætning af plast med lav densitet i et svovlholdigt opløsningsmiddel, at sætte det i en mikrobølgeovn og omdanne det til et kulstofstillads gør, at lithium-svovl-batterie -
 Elektrokatalysator udviser fremragende vandspaltningsaktivitetVist ovenfor demonstrerer den teoretisk forudsagte struktur af Fe 3 Co(PO 4 ) 4 @rGO. Jern (Fe) er angivet som gul, kobolt (Co) i blåt, fosforsyre (P) i lilla, oxygen (O) i rødt, og hydrogen (H)
Elektrokatalysator udviser fremragende vandspaltningsaktivitetVist ovenfor demonstrerer den teoretisk forudsagte struktur af Fe 3 Co(PO 4 ) 4 @rGO. Jern (Fe) er angivet som gul, kobolt (Co) i blåt, fosforsyre (P) i lilla, oxygen (O) i rødt, og hydrogen (H) -
 Alsidige byggesten laver strukturer med overraskende mekaniske egenskaberFire typer af diskret sammensatte mekaniske metamaterialer. Venstre til højre:Stiv, i overensstemmelse, auxetic, og chiral. (A) Som støbte ansigtsdele. (B) Single voxel, forfra. (C) En 2 × 2 × 2 terni
Alsidige byggesten laver strukturer med overraskende mekaniske egenskaberFire typer af diskret sammensatte mekaniske metamaterialer. Venstre til højre:Stiv, i overensstemmelse, auxetic, og chiral. (A) Som støbte ansigtsdele. (B) Single voxel, forfra. (C) En 2 × 2 × 2 terni
- Worst-case CO2-emissionsscenarie er bedst til at vurdere klimarisiko og påvirkninger frem til 2050
- Sådan bruges en Nikon Digital SLR på et teleskop
- Mørk materie orkan giver mulighed for at opdage aksioner
- Solceller, der kombinerer silicium med perovskit, har opnået rekordeffektivitet på 25,2 procent
- Digitalt landbrug baner vejen til landbrugets bæredygtighed
- Hvordan er celler, væv og organer relateret?


