Ny kortlægningsmetode belyser medicinbare steder på proteiner
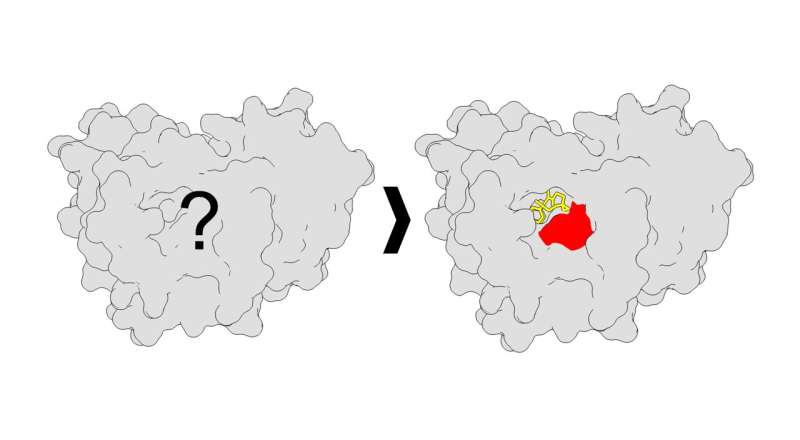
At identificere nye måder at målrette proteiner involveret i menneskelige sygdomme på er en prioritet for mange forskere rundt om i verden. Men det kan være svært at opdage, hvordan man ændrer funktionen af disse proteiner, især i levende celler. Nu har forskere fra Scripps Research udviklet en ny metode til at undersøge, hvordan proteiner interagerer med lægemiddellignende små molekyler i menneskelige celler – og afslører kritisk information om, hvordan man potentielt målretter dem terapeutisk.
Strategien, offentliggjort i Nature Chemical Biology den 2. januar 2024, bruger en kombination af kemi og analytiske teknikker til at afsløre de specifikke steder, hvor proteiner og små molekyler binder sammen. I sidste ende kan denne metode føre til udvikling af mere målrettede og effektive terapier.
"Vores nye teknologi kan bruges til at finde nye lægelige steder på proteiner til enhver menneskelig sygdom, fra kræft til Alzheimers sygdom," siger lektor ved Institut for Kemi Christopher Parker, Ph.D., seniorforfatter af undersøgelsen. "Vi er ubegrænsede i, hvordan dette kan bruges. Vores arbejde har potentialet til at indlede en helt ny måde at opdage lægemidler på."
Parker-laboratoriet har til formål at opdage, hvordan proteiner fungerer i enhver menneskelig celletype for at udvikle effektive terapeutiske midler til en bred vifte af menneskelige sygdomme. I denne undersøgelse byggede Parker og hans team sit indledende arbejde i Scripps Research professor Benjamin Cravatts laboratorium for at skabe en ny metode til at undersøge, hvordan proteiner interagerer med små molekyler i levende celler.
De udviklede en analytisk strategi for bedre at forstå, hvordan disse proteiner interagerer med små molekyler med meget højere opløsning end nogensinde før. For at gøre dette brugte de kemiske prober kaldet fotoaffinitetsprober, som er molekyler, der kan aktiveres af lys for at tillade proberne at fange et bundet protein.
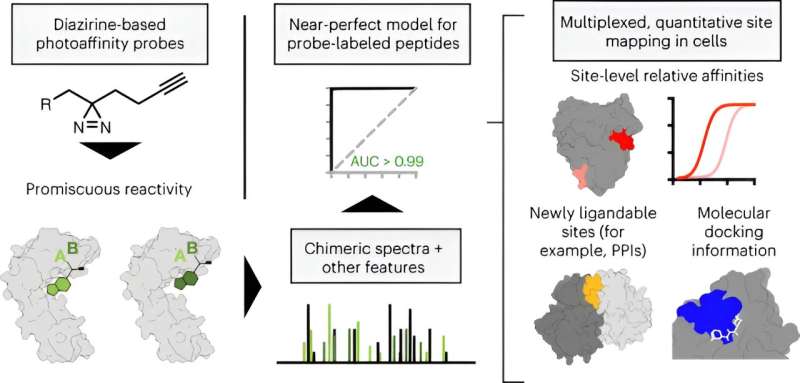
Ved at indsamle data fra interaktioner mellem proteiner og fotoaffinitetsprober identificerede Parker-teamet steder på proteiner, hvor små molekyler kunne forbinde og binde. I det væsentlige fandt holdet over tusind nye låse (bindingssteder på proteinerne) og tilsvarende nøgler (små molekyler), hvoraf langt størstedelen var nye steder med små molekylebindinger, som ikke var blevet rapporteret før. Derudover fandt de nye funktioner ved bindingsstederne – såsom nye former.
"At identificere disse specifikke bindingssteder vil hjælpe videnskabsmænd med at designe nye molekyler, der passer endnu bedre til disse lommer, hvilket potentielt kan føre til mere effektive terapier," siger Jacob M. Wozniak, medforfatter og tidligere postdoktor i Parker-laboratoriet. Den anden medførsteforfatter af artiklen var Weichao Li, Ph.D., en forskningsmedarbejder også i Parker-laboratoriet.
Ved at bruge rigdommen af data i denne undersøgelse og samarbejde med medforfatter Stefano Forli, Ph.D., lektor i Institut for Integrativ Strukturel og Beregningsbiologi, modellerede forfatterne derefter, hvordan visse molekyler kunne binde til disse proteiner. Dette informationsbibliotek kunne bruges til at designe terapier, der interagerer med proteiner på en mere målrettet måde.
"Vores nye proces afslører yderligere muligheder for terapeutisk intervention og opdagelse i menneskelige celler," siger Parker. "Dernæst planlægger vi at bruge denne teknologi til at målrette proteiner, der er relevante for autoimmune sygdomme og kræft."
Flere oplysninger: Jacob M. Wozniak et al., Enhanced mapping of small-molecule binding sites in cells, Nature Chemical Biology (2024). DOI:10.1038/s41589-023-01514-z
Journaloplysninger: Kemisk naturbiologi
Leveret af The Scripps Research Institute
 Varme artikler
Varme artikler
-
 Laboratoriedyrket mejeri:Den næste fødevaregrænseKan mejeriprodukter snart produceres i laboratorier? Kredit:Shutterstock Lab-dyrket kød får stor opmærksomhed sammen med plantebaserede køderstatninger. Teknologi driver industrien i retning af at
Laboratoriedyrket mejeri:Den næste fødevaregrænseKan mejeriprodukter snart produceres i laboratorier? Kredit:Shutterstock Lab-dyrket kød får stor opmærksomhed sammen med plantebaserede køderstatninger. Teknologi driver industrien i retning af at -
 Tændt for IR-aktive organiske pigmenterKredit:Wiley I fotosyntese og organisk solcelle, pigmentmolekyler omdanner lys til elektrisk ladning. Et team af kemikere har nu produceret et usædvanligt organisk pigment, som tændes af en elektr
Tændt for IR-aktive organiske pigmenterKredit:Wiley I fotosyntese og organisk solcelle, pigmentmolekyler omdanner lys til elektrisk ladning. Et team af kemikere har nu produceret et usædvanligt organisk pigment, som tændes af en elektr -
 Tænd og sluk for en porøs materialefarve med syreBillede af den skabte hydrogenbinding organiske ramme CPHATN- (TCB) (til venstre), og dens aktiverede version, CPHATN-1a (højre). Kredit:Hisaki I. et al., Syreresponsive, hydrogenbundne organiske ramm
Tænd og sluk for en porøs materialefarve med syreBillede af den skabte hydrogenbinding organiske ramme CPHATN- (TCB) (til venstre), og dens aktiverede version, CPHATN-1a (højre). Kredit:Hisaki I. et al., Syreresponsive, hydrogenbundne organiske ramm -
 Carbonopsamling tager en svampelignende form med ny omkostningseffektiv metodeBall-and-stick model af kuldioxid. Kredit:Wikipedia Et internationalt hold af videnskabsmænd bruger porøse, svampelignende materialer, der kan fange kuldioxid i deres hulrum, samtidig med at andre
Carbonopsamling tager en svampelignende form med ny omkostningseffektiv metodeBall-and-stick model af kuldioxid. Kredit:Wikipedia Et internationalt hold af videnskabsmænd bruger porøse, svampelignende materialer, der kan fange kuldioxid i deres hulrum, samtidig med at andre
- Inside job:En ny teknik til afkøling af en fusionsreaktor
- Billede:European Service Module-komponent af Orion-rumfartøjet
- Teamopfindelse kan hjælpe med at beskytte astronauter mod stråling i rummet
- Er alder forbundet med billedet af den perfekte partner?
- Lab-fremstillede sekskantede diamanter stivere end naturlige diamanter
- Ny indsigt i, hvordan antiarytmika virker


