DNA-origami:Foldet DNA som byggemateriale til molekylære enheder
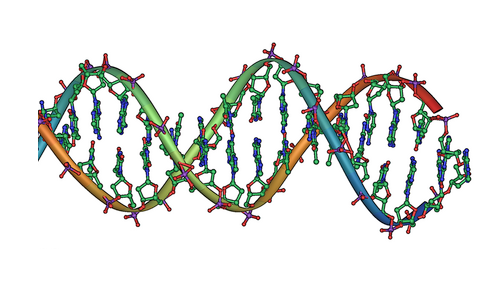
DNA dobbelt helix. Kredit:offentlig ejendom
Levende ting bruger DNA til at lagre den genetiske information, der gør hver plante, bakterie, og mennesket unikt. Gengivelsen af denne information er gjort mulig, fordi DNA's nukleotider - A'er og T'er, G'er og C'er - passer perfekt sammen, som matchende puslespilsbrikker. Ingeniører kan drage fordel af matchningen mellem lange strenge af DNA-nukleotider til at bruge DNA som en slags molekylær origami, folde det ind i alt fra smiley-ansigtskunst i nanoskala til seriøse midler til udlevering af stoffer.
Paul Rothemund diskuterer potentialet i sådanne teknikker. Rothemund er forskningsprofessor i bioteknik, edb og matematiske videnskaber, og beregnings- og neurale systemer i Division of Engineering and Applied Science på Caltech.
Hvad laver du?
Jeg bruger DNA og RNA som byggematerialer til at skabe former og mønstre med en opløsning på kun få nanometer. De mindste træk i de DNA-strukturer, vi laver, er omkring 20, 000 gange mindre end pixels på de smarteste computerskærme, som hver er omkring 80 mikron på tværs. En stor del af vores arbejde gennem de sidste 20 år har netop været at finde ud af, hvordan man får DNA- eller RNA-strenge til at folde sig selv til en ønsket computerdesignet form. Da vi har mestret evnen til at lave den form eller det mønster, vi ønsker, vi er gået videre til at bruge disse former som "pegboards" til at arrangere andre objekter i nanostørrelse, såsom proteinenzymer, kulstof-nanorør transistorer, og fluorescerende molekyler.
Hvorfor er dette vigtigt?
Hver opgave i din krop, fra at fordøje mad til at bevæge dine muskler til at føle lys, er drevet af bittesmå biologiske maskiner i nanometerskala, alt sammen bygget fra "nedefra" via selvfoldning af molekyler som proteiner og RNA'er. De milliarder af transistorer, der udgør chipsene i vores mobiltelefoner og computere, er titusinder af nanometer store, men de er bygget på en "top-down" måde ved hjælp af smarte trykprocesser i milliardfabrikker. Vores mål er at lære at bygge komplekse kunstige enheder, som biologi bygger naturlige - dvs. startende fra selvfoldende molekyler, der samles til større mere komplekse strukturer. Ud over langt billigere enheder, dette vil muliggøre helt nye applikationer, såsom menneskeskabte molekylære maskiner, der kan træffe komplekse terapeutiske beslutninger og kun anvende lægemidler, hvor det er nødvendigt.
Hvordan kom du ind i denne branche?
Som bachelor på Caltech, Jeg havde meget svært ved at beslutte mig for, hvordan jeg skulle kombinere mine forskellige interesser inden for datalogi, kemi, og biologi. Heldigvis, afdøde Jan L. A. van de Snepscheut introducerede sin datalogi-klasse til den hypotetiske idé om at bygge en DNA Turing-maskine - en meget simpel maskine, som ikke desto mindre kan køre alle mulige computerprogrammer. Han udfordrede os, tyder på, at nogen, der kendte til både biokemi og datalogi, kunne finde på en konkret måde at bygge sådan en DNA-computer på. Til en projektklasse i informationsteori med Yaser Abu-Mostafa, professor i elektroteknik og datalogi, Jeg fandt på en ret ineffektiv, endnu muligt, måde at gøre dette på. På det tidspunkt, Jeg kunne ikke interessere nogen Caltech-professorer i at bygge min DNA-computer, men kort efter, USC-professor Len Adleman udgav et papir om en mere praktisk DNA-computer i Videnskab . Jeg kom til Adlemans laboratorium på USC som kandidatstuderende, og jeg har prøvet at bruge DNA til at bygge computere eller andre komplekse enheder lige siden. Jeg vendte tilbage til Caltech som postdoc i 2001 og blev forskningsprofessor i 2008.
 Varme artikler
Varme artikler
-
 Bøjning - men ikke brud - på jagt efter nyt materialeMXene-polymer nanokompositmateriale, skabt af Drexel -forskere, viser enestående fleksibilitet, styrke og ledningsevne. Kredit:Drexel University At lave et papirfly i skolen plejede at betyde prob
Bøjning - men ikke brud - på jagt efter nyt materialeMXene-polymer nanokompositmateriale, skabt af Drexel -forskere, viser enestående fleksibilitet, styrke og ledningsevne. Kredit:Drexel University At lave et papirfly i skolen plejede at betyde prob -
 Robust ny proces danner 3D-former fra flade ark af grafenDenne undersøgelse viser grafenintegration til en række forskellige mikrostrukturerede geometrier, herunder pyramider, søjler, kupler, og omvendte pyramider. Kredit:Nam Research Group, University of I
Robust ny proces danner 3D-former fra flade ark af grafenDenne undersøgelse viser grafenintegration til en række forskellige mikrostrukturerede geometrier, herunder pyramider, søjler, kupler, og omvendte pyramider. Kredit:Nam Research Group, University of I -
 Nano-klitter med ionstrålen:Ny metode til selvorganiserede nanostrukturerMed ionstrålen som værktøj, forskerne ved Helmholtz-Zentrum Dresden-Rossendorf, Tyskland, er det lykkedes at skabe selvorganiserede krystallinske strukturer på nanoskala. Kredit:SIMIT, det kinesiske v
Nano-klitter med ionstrålen:Ny metode til selvorganiserede nanostrukturerMed ionstrålen som værktøj, forskerne ved Helmholtz-Zentrum Dresden-Rossendorf, Tyskland, er det lykkedes at skabe selvorganiserede krystallinske strukturer på nanoskala. Kredit:SIMIT, det kinesiske v -
 Snoede van der Waals-materialer som en ny platform til at realisere eksotisk stofTwisted van der Waals materialer tilbyder et enormt potentiale for grundforskning, materialevidenskab og kvanteteknologier. Kredit:Jörg Harms / MPSD Forskere fra MPSD, RWTH Aachen University og Fl
Snoede van der Waals-materialer som en ny platform til at realisere eksotisk stofTwisted van der Waals materialer tilbyder et enormt potentiale for grundforskning, materialevidenskab og kvanteteknologier. Kredit:Jörg Harms / MPSD Forskere fra MPSD, RWTH Aachen University og Fl
- Flyt dig, Laurel eller Yanny:Undersøgelse ser på, hvorfor vi hører tale som sang efter mange gent…
- Hvad sker der i geologi?
- Suomi NPP-satellit får nat og infrarød udsigt over orkanen Hector
- Hvilken type kropsbeklædninger har amfibier?
- Biofysikstudier gør spændende fremskridt for fremtiden for DNA-sekventering
- Billede:400 hektar stor naturbrand er den største nogensinde i den tyske delstat


