En klar vej til bedre indsigt i biomolekyler

Illustration af 3D-diffraktionsmønsteret af oktaedriske nanopartikler, opnået ved at kombinere mange snapshots efter strukturel udvælgelse. Kredit:Kartik Ayyer / Jörg Harms, MPSD
Et internationalt hold af forskere ledet af Kartik Ayyer fra MPSD har opnået nogle af de skarpest mulige 3-D-billeder af guld-nanopartikler. Resultaterne danner grundlaget for at opnå billeder i høj opløsning af makromolekyler. Undersøgelsen blev udført på det europæiske XFEL's Single Particles, Klynger, og Biomolecules &Serial Femtosecond Crystallography (SPB/SFX) instrument, og resultaterne er blevet offentliggjort i Optica .
Kulhydrater, lipider, proteiner og nukleinsyrer er mikromolekyler, der befolker celler og er livsvigtige. Nøglen til at forstå, hvordan disse makromolekyler virker, ligger i at forstå deres struktur. Brug af guld nanopartikler som erstatning for biomolekyler, holdet målte 10 millioner diffraktionsmønstre og brugte dem til at generere 3D-billeder med rekordstor opløsning. Guldpartikler spreder langt flere røntgenstråler end bio-prøver og er dermed gode testprøver. De giver mange flere data, som gør dem meget nyttige til finjustering af metoder, der derefter kan bruges på biomolekyler.
"Teknikkerne, der bruges til at opnå billeder i høj opløsning af biomolekyler, omfatter røntgenkrystallografi, som kræver, at biomolekylerne krystalliseres, " siger Kartik Ayyer, lederen af Computational Nanoscale Imaging-gruppen ved MPSD. "Dette er ikke en nem proces. Alternativt kryo-elektronmikroskopi arbejder med frosne molekyler, " tilføjer han. Men fremkomsten af røntgenfri elektronlasere åbnede dørene til enkeltpartikelbilleddannelse (SPI), en teknik, der har potentialet til at levere højopløselige billeder af biomolekyler ved stuetemperatur og uden krystallisering. Derfor kan biomolekylerne studeres tættere på deres oprindelige tilstand. Dette giver igen bedre indsigt i deres struktur og funktion i vores kroppe.
Men to forhindringer forblev i SPI:Indsamling af nok højkvalitetsdiffraktionsmønstre og korrekt klassificering af biomolekylernes strukturelle variabilitet. Teamets arbejde viser, at begge disse barrierer kan overvindes, siger Kartik Ayyer:"Tidligere SPI-eksperimenter producerede kun omkring titusindvis af diffraktionsmønstre, selv i bedste tilfælde. Imidlertid, at få resolutioner, der er relevante for strukturel biologi, forskere har brug for 10 til 100 gange flere diffraktionsmønstre." forklarer Ayyer. "Takket være de unikke muligheder i den europæiske XFEL-facilitet, nemlig det høje antal røntgenlaserimpulser pr. sekund og høj pulsenergi, holdet var i stand til at indsamle 10 millioner diffraktionsmønstre i et enkelt 5-dages eksperiment. Denne mængde data er uden fortilfælde, og vi tror, at vores eksperiment vil tjene som en skabelon for fremtiden for dette forskningsfelt, " han siger.
For at overvinde problemet med strukturel variabilitet af biomolekyler, det er, beskæftiger sig med et øjebliksbillede fra hver partikel, der er lidt forskellige fra hinanden, forskerne udviklede en særlig algoritme. Diffraktionsmønstrene opsamles af en todimensionel detektor - meget ligesom et hurtigt røntgenkamera. En algoritme sorterer derefter dataene og giver forskerne mulighed for at rekonstruere billedet af biomolekylet. "Vi brugte funktionerne i Adaptive Gain Integrating Pixel Detector (AGIPD), hvilket gjorde det muligt for os at fange mønstre i den høje hastighed. Vi indsamlede og analyserede derefter dataene med tilpassede algoritmer for at opnå billeder med rekordstor opløsning, " siger Ayyer.
"Denne undersøgelse udnyttede virkelig den unikke egenskab ved den høje gentagelsesrate på vores anlæg, hurtig-framing detektor og effektiv prøvelevering, " siger Adrian Mancuso, førende videnskabsmand i SPB/SFX-gruppen. "Det viser, at i fremtiden European XFEL er godt placeret til at udforske grænserne for 'syn' for ukrystalliseret, stuetemperatur biomolekyler."
Sidste artikelKraftfuldt grafenhybridmateriale til højeffektive superkondensatorer
Næste artikelSådan løser du magnetiske nano-knuder
 Varme artikler
Varme artikler
-
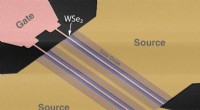 Skalerbare todimensionelle materialer fremmer fremtidens elektronikFalskfarvet scanningselektronmikroskop (SEM) billede af en radiofrekvent felt-effekt-transistor (RF-FET) sammensat af en 2-3 lags tyk epitaxialt vokset wolframdiselenid (WSe2) aktiv kanal. Kredit:Bria
Skalerbare todimensionelle materialer fremmer fremtidens elektronikFalskfarvet scanningselektronmikroskop (SEM) billede af en radiofrekvent felt-effekt-transistor (RF-FET) sammensat af en 2-3 lags tyk epitaxialt vokset wolframdiselenid (WSe2) aktiv kanal. Kredit:Bria -
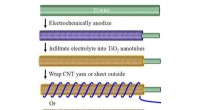 Forskere skræddersyr strømkilde til bærbar elektronikRadialt justerede titaniumoxidnanorør øger overfladearealet, at pakke mere kraft i tøjets rammer. Kredit:Udlånt af tidsskriftet Energy Storage Materials Bærbare strømkilder til bærbar elektronik e
Forskere skræddersyr strømkilde til bærbar elektronikRadialt justerede titaniumoxidnanorør øger overfladearealet, at pakke mere kraft i tøjets rammer. Kredit:Udlånt af tidsskriftet Energy Storage Materials Bærbare strømkilder til bærbar elektronik e -
 Forskere udvikler en ny proces til strukturering af kvantematerialer(a) Scanningelektronmikrofotografi taget under Jülich-processen:Vist er en matrice under fremstilling. Den topologiske isolator (angivet med rødt) er allerede deponeret selektivt. I et næste fremstill
Forskere udvikler en ny proces til strukturering af kvantematerialer(a) Scanningelektronmikrofotografi taget under Jülich-processen:Vist er en matrice under fremstilling. Den topologiske isolator (angivet med rødt) er allerede deponeret selektivt. I et næste fremstill -
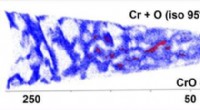 Hjælper materialer med at håndtere ekstrem stressVigtige trykvands-atomreaktorkomponenter fremstilles af en nikkel-baseret legering, der indeholder dobbelt så meget krom som det tidligere anvendte materiale. Den nye legering, kaldet legering 690, pr
Hjælper materialer med at håndtere ekstrem stressVigtige trykvands-atomreaktorkomponenter fremstilles af en nikkel-baseret legering, der indeholder dobbelt så meget krom som det tidligere anvendte materiale. Den nye legering, kaldet legering 690, pr
- Ved prøveløsladelse, kriminalforsorgen vælger empati, vender tilbage til fængslet afvisning
- Fremstilling af komplekse 3D metalliske strukturer i nanoskala muliggjort
- Voldslegitimerende vers i religiøse tekster øger støtten til dødelig vold
- Sådan læses en Sling Psychrometer
- Kan jeg blande disse kemikalier? Der er en app til det!
- Schumer foreslår 462 milliarder dollars bilbytte - gas til el


