Nanoteknologi muliggør visualisering af RNA-strukturer ved atomær opløsning
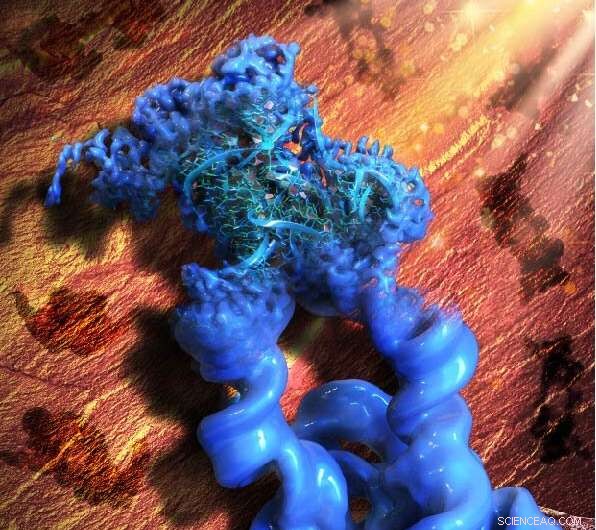
Denne illustration er inspireret af det palæolitiske klippemaleri i Lascaux-hulen, hvilket betyder akronymet for vores metode, ROCK. Billedligt er mønstrene af klippekunsten i baggrunden (brun) 2D-projektionerne af den konstruerede dimeriske konstruktion af Tetrahymena-gruppe I-intronen, mens hovedobjektet foran (blåt) er det rekonstruerede 3D-kryo-EM-kort af dimer, med én monomer i fokus og raffineret til den høje opløsning, der gjorde det muligt for samarbejdspartnerne at bygge en atommodel af RNA'et. Kredit:Wyss Institute ved Harvard University
Vi lever i en verden lavet og drevet af RNA, den lige så vigtige søskende til det genetiske molekyle DNA. Faktisk antager evolutionsbiologer, at RNA eksisterede og selv-replikerede selv før fremkomsten af DNA og proteinerne kodet af det. Spol frem til moderne mennesker:Videnskaben har afsløret, at mindre end 3 % af det menneskelige genom transskriberes til messenger RNA (mRNA) molekyler, der igen oversættes til proteiner. I modsætning hertil er 82% af det transskriberet til RNA-molekyler med andre funktioner, hvoraf mange stadig er gådefulde.
For at forstå, hvad et individuelt RNA-molekyle gør, skal dets 3D-struktur dechifreres på niveau med dets konstituerende atomer og molekylære bindinger. Forskere har rutinemæssigt studeret DNA- og proteinmolekyler ved at omdanne dem til regelmæssigt pakkede krystaller, der kan undersøges med en røntgenstråle (røntgenkrystallografi) eller radiobølger (kernemagnetisk resonans). Imidlertid kan disse teknikker ikke anvendes på RNA-molekyler med næsten samme effektivitet, fordi deres molekylære sammensætning og strukturelle fleksibilitet forhindrer dem i let at danne krystaller.
Nu er et forskningssamarbejde ledet af Wyss Core Faculty-medlem Peng Yin, Ph.D. ved Wyss Institute for Biologically Inspired Engineering ved Harvard University, og Maofu Liao, Ph.D. på Harvard Medical School (HMS), har rapporteret om en fundamentalt ny tilgang til strukturel undersøgelse af RNA-molekyler. ROCK, som det kaldes, bruger en RNA-nanoteknologisk teknik, der gør det muligt at samle flere identiske RNA-molekyler til en meget organiseret struktur, hvilket reducerer fleksibiliteten af individuelle RNA-molekyler betydeligt og multiplicerer deres molekylvægt. Anvendt på velkendte model-RNA'er med forskellige størrelser og funktioner som benchmarks viste holdet, at deres metode muliggør strukturel analyse af de indeholdte RNA-underenheder med en teknik kendt som kryo-elektronmikroskopi (cryo-EM). Deres fremskridt er rapporteret i Naturmetoder .
"ROCK bryder de nuværende grænser for RNA-strukturundersøgelser og gør det muligt at låse op for 3D-strukturer af RNA-molekyler, som er svære eller umulige at få adgang til med eksisterende metoder og ved nær-atomisk opløsning," sagde Yin, der sammen med Liao ledede undersøgelsen . "Vi forventer, at dette fremskridt vil styrke mange områder inden for grundlæggende forskning og lægemiddeludvikling, herunder det spirende felt af RNA-terapi." Yin er også leder af Wyss Institutes Molecular Robotics Initiative og professor ved Institut for Systembiologi ved HMS.
Få kontrol over RNA
Yins team på Wyss Institute har været banebrydende for forskellige tilgange, der gør det muligt for DNA- og RNA-molekyler at samle sig selv til store strukturer baseret på forskellige principper og krav, herunder DNA-klodser og DNA-origami. De antog, at sådanne strategier også kunne bruges til at samle naturligt forekommende RNA-molekyler til højt ordnede cirkulære komplekser, hvor deres frihed til at bøje og bevæge sig er stærkt begrænset ved specifikt at forbinde dem sammen. Mange RNA'er folder på komplekse, men forudsigelige måder, hvor små segmenter baseparrer sig med hinanden. Resultatet er ofte en stabiliseret "kerne" og "stammeløkker", der buler ud i periferien.
"I vores tilgang installerer vi 'kysseløkker', der forbinder forskellige perifere stamløkker, der tilhører to kopier af et identisk RNA på en måde, der gør det muligt at danne en samlet stabiliseret ring, der indeholder flere kopier af RNA'et af interesse," sagde Di. Liu, Ph.D., en af to førsteforfattere og en postdoktor i Yins gruppe. "Vi spekulerede i, at disse ringe af højere orden kunne analyseres med høj opløsning ved hjælp af cryo-EM, som var blevet anvendt på RNA-molekyler med første succes."
Afbildning af stabiliseret RNA
I cryo-EM bliver mange enkeltpartikler lynfrosset ved kryogene temperaturer for at forhindre yderligere bevægelser og visualiseres derefter med et elektronmikroskop og ved hjælp af beregningsalgoritmer, der sammenligner de forskellige aspekter af en partikels 2D-overfladeprojektioner og rekonstruerer dens 3D-arkitektur. . Peng og Liu slog sig sammen med Liao og hans tidligere kandidatstuderende François Thélot, Ph.D., den anden medførsteforfatter af undersøgelsen. Liao har med sin gruppe ydet vigtige bidrag til det hastigt fremadskridende kryo-EM-felt og den eksperimentelle og beregningsmæssige analyse af enkelte partikler dannet af specifikke proteiner.
"Cryo-EM har store fordele i forhold til traditionelle metoder til at se detaljer i høj opløsning af biologiske molekyler, herunder proteiner, DNA'er og RNA'er, men den lille størrelse og bevægelige tendens af de fleste RNA'er forhindrer vellykket bestemmelse af RNA-strukturer. Vores nye metode til at samle RNA-multimerer løser disse to problemer på samme tid ved at øge størrelsen af RNA og reducere dets bevægelse," sagde Liao, der også er lektor i cellebiologi ved HMS. "Vores tilgang har åbnet døren til hurtig strukturbestemmelse af mange RNA'er ved cryo-EM." Integrationen af RNA-nanoteknologi og cryo-EM-tilgange fik holdet til at navngive deres metode "RNA-oligomeriseringsaktiveret cryo-EM via installation af kyssløjfer" (ROCK).
For at levere proof-of-princippet for ROCK fokuserede holdet på et stort intron-RNA fra Tetrahymena, en encellet organisme, og et lille intron-RNA fra Azoarcus, en nitrogenfikserende bakterie, samt den såkaldte FMN-riboswitch . Intron-RNA'er er ikke-kodende RNA-sekvenser spredt ud over sekvenserne af frisk-transskriberede RNA'er og skal "splejses" ud for at det modne RNA kan dannes. FMN-riboswitchen findes i bakterielle RNA'er involveret i biosyntesen af flavinmetabolitter afledt af vitamin B2. Ved binding af et af dem, flavinmononukleotid (FMN), skifter det sin 3D-konformation og undertrykker syntesen af dets moder-RNA.
"Samlingen af Tetrahymena gruppe I-intron til en ringlignende struktur gjorde prøverne mere homogene og muliggjorde brugen af beregningsværktøjer, der udnyttede symmetrien i den samlede struktur. Selvom vores datasæt er relativt beskedent i størrelse, tillod ROCKs medfødte fordele os at løse strukturen med en hidtil uset opløsning," sagde Thélot. "RNA's kerne er opløst ved 2,85 Å [en Ångström er en ti-milliarder (US) af en meter og den foretrukne metrisk brugt af strukturelle biologer], hvilket afslører detaljerede træk ved nukleotidbaserne og sukkerrygraden. Jeg tror ikke, vi kunne være kommet dertil uden ROCK - eller i det mindste ikke uden betydeligt flere ressourcer."
Cryo-EM er også i stand til at fange molekyler i forskellige tilstande, hvis de for eksempel ændrer deres 3D-konformation som en del af deres funktion. Ved at anvende ROCK på Azoarcus intron RNA og FMN riboswitch, lykkedes det teamet at identificere de forskellige konformationer, som Azoarcus intronen går igennem under sin selvsplejsningsproces, og at afsløre den relative konformationelle stivhed af FMN riboswitchens ligandbindingssted .
"Denne undersøgelse af Peng Yin og hans samarbejdspartnere viser elegant, hvordan RNA-nanoteknologi kan fungere som en accelerator til at fremme andre discipliner. At være i stand til at visualisere og forstå strukturerne af mange naturligt forekommende RNA-molekyler kan have en enorm indflydelse på vores forståelse af mange biologiske og patologiske processer på tværs af forskellige celletyper, væv og organismer og muliggør endda nye tilgange til lægemiddeludvikling," sagde Wyss-grundlægger, Donald Ingber, M.D., Ph.D. + Udforsk yderligere
Ny indsigt i strukturerne og mekanismerne af nøgleproteiner involveret i mikrobiel fotosyntese
 Varme artikler
Varme artikler
-
 GraphON:Ledende belægninger og materialers gennembrudEn GraphON belægning på kvarts. Kredit:CSIRO Australiens nationale videnskabsagentur, CSIRO, har skabt en banebrydende ny form for grafitisk materiale, der er ledende, nem at anvende og giver stør
GraphON:Ledende belægninger og materialers gennembrudEn GraphON belægning på kvarts. Kredit:CSIRO Australiens nationale videnskabsagentur, CSIRO, har skabt en banebrydende ny form for grafitisk materiale, der er ledende, nem at anvende og giver stør -
 Nanotråde til fremtidens elektronik og optoelektronikUnder transmissionselektronmikroskopet (TEM), flere kobolt phthalocyanin nanotråde vokser ud af en jern phthalocyanin nanotråd, der er dekoreret med sølvpartikler. Kredit:Empa Organiske halvledere
Nanotråde til fremtidens elektronik og optoelektronikUnder transmissionselektronmikroskopet (TEM), flere kobolt phthalocyanin nanotråde vokser ud af en jern phthalocyanin nanotråd, der er dekoreret med sølvpartikler. Kredit:Empa Organiske halvledere -
 Hvordan man deler et vandmolekyleFigur 1:STM -billede af ultratynd MgO -film på Ag (100) overflade. (PhysOrg.com) - Et forskerhold på RIKEN, Japans flagskibs forskningsorganisation er for første gang lykkedes selektivt at kontrol
Hvordan man deler et vandmolekyleFigur 1:STM -billede af ultratynd MgO -film på Ag (100) overflade. (PhysOrg.com) - Et forskerhold på RIKEN, Japans flagskibs forskningsorganisation er for første gang lykkedes selektivt at kontrol -
 Går svømmende:Biotemplates gennembrud baner vej for billigere nanobotterBane for en skabeloniseret spiralformet silica nanosvømmer manuelt styret til at bevæge sig i et omtrentligt figur-otte mønster; skala bar er 5 μm. Kredit:Jamel Ali Et træk ved science fiction -hi
Går svømmende:Biotemplates gennembrud baner vej for billigere nanobotterBane for en skabeloniseret spiralformet silica nanosvømmer manuelt styret til at bevæge sig i et omtrentligt figur-otte mønster; skala bar er 5 μm. Kredit:Jamel Ali Et træk ved science fiction -hi
- Kaos er ikke tilfældighed:En kompleks systemforsker forklarer
- Mexico øger beskyttelsen på præ-spansktalende ceremonielle sted
- Fleksibel bioelektronisk enhed baseret på levende materiale til overvågning af laktat- og tumorcel…
- Undersøgelser undersøger marine hedebølger, skiftende havstrømme
- Flyvende biler kan reducere emissionerne, udskifte fly, og frigør veje – men ikke hurtigt nok
- Et nyt magnetisk materiale og en optagelsesproces for at øge datakapaciteten markant


