Ny metode til at generere potente, specifikke bindingsproteiner til nye lægemidler
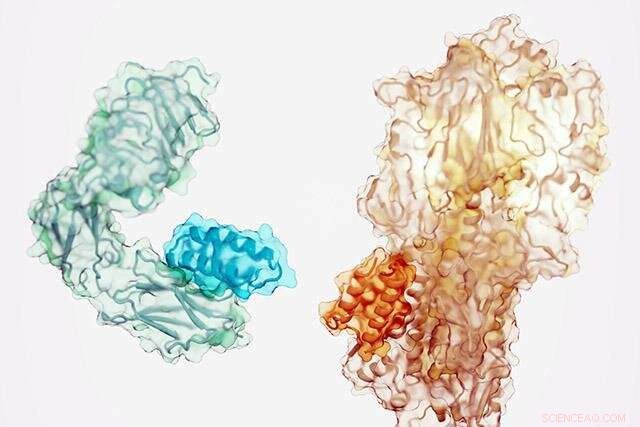
Små proteiner (mørkere nuance) designet til at binde sig til insulinreceptoren (venstre) og en komponent af influenzavirussen (højre). Kredit:Ian C. Haydon/Institute for Protein Design
Et team af videnskabsmænd har skabt en kraftfuld ny metode til at generere proteinlægemidler. Ved hjælp af computere designede de molekyler, der kan målrette mod vigtige proteiner i kroppen, såsom insulinreceptoren, samt sårbare proteiner på overfladen af vira. Dette løser en langvarig udfordring inden for udvikling af lægemidler og kan føre til nye behandlinger for kræft, diabetes, infektion, inflammation og mere.
Forskningen vises i dag i tidsskriftet Nature , blev ledet af forskere i laboratoriet af David Baker, professor i biokemi ved University of Washington School of Medicine og modtager af 2021 Breakthrough Prize in Life Sciences.
"Evnen til at generere nye proteiner, der binder tæt og specifikt til ethvert molekylært mål, du ønsker, er et paradigmeskifte i lægemiddeludvikling og molekylærbiologi mere bredt," sagde Baker.
Antistoffer er nutidens mest almindelige proteinbaserede lægemidler. De fungerer typisk ved at binde sig til et specifikt molekylært mål, som derefter enten bliver aktiveret eller deaktiveret. Antistoffer kan behandle en lang række sundhedssygdomme, herunder COVID-19 og kræft, men det er en udfordring at generere nye. Antistoffer kan også være dyre at fremstille.
Et team ledet af to postdoktorale forskere i Baker-laboratoriet - Longxing Cao og Brian Coventry - kombinerede de seneste fremskridt inden for beregningsproteindesign for at nå frem til en strategi til at skabe nye proteiner, der binder molekylære mål på en måde, der ligner antistoffer. De udviklede software, der kan scanne et målmolekyle, identificere potentielle bindingssteder, generere proteiner rettet mod disse steder og derefter screene fra millioner af kandidatbindingsproteiner for at identificere dem, der med størst sandsynlighed vil fungere.
Holdet brugte den nye software til at generere højaffinitetsbindende proteiner mod 12 forskellige molekylære mål. Disse mål omfatter vigtige cellulære receptorer såsom TrkA, EGFR, Tie2 og insulinreceptoren samt proteiner på overfladen af influenzavirus og SARS-CoV-2 (virussen, der forårsager COVID-19).
"Når det kommer til at skabe nye lægemidler, er der lette mål, og der er hårde mål," sagde Cao, som nu er assisterende professor ved Westlake University. "I dette papir viser vi, at selv meget hårde mål er modtagelige for denne tilgang. Vi var i stand til at lave bindende proteiner til nogle mål, der ikke havde nogen kendte bindingspartnere eller antistoffer,"
I alt producerede holdet over en halv million kandidatbindende proteiner til de 12 udvalgte molekylære mål. Data indsamlet om denne store pulje af kandidatbindende proteiner blev brugt til at forbedre den overordnede metode.
"Vi ser frem til at se, hvordan disse molekyler kan bruges i en klinisk sammenhæng, og endnu vigtigere, hvordan denne nye metode til at designe proteinlægemidler kan føre til endnu mere lovende forbindelser i fremtiden," sagde Coventry. + Udforsk yderligere
Mod ét lægemiddel til behandling af alle coronavirus
 Varme artikler
Varme artikler
-
 Fluorescerende alkometer gør optimering af katalysatorer meget nemmereFluorescensmikroskopibilleder viser virkningerne af forskellige former på katalysatoraktivitet. Kredit:Utrecht University En ny test for industrielle katalysatorer udviklet af kemikere ved Utrecht
Fluorescerende alkometer gør optimering af katalysatorer meget nemmereFluorescensmikroskopibilleder viser virkningerne af forskellige former på katalysatoraktivitet. Kredit:Utrecht University En ny test for industrielle katalysatorer udviklet af kemikere ved Utrecht -
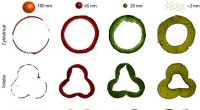
 Ny maskinlæringstilgang letter molekylær konformersøgning i komplekse molekylerKredit:Aalto Universitet CEST-forskere udviklede en ny maskinlæringstilgang baseret på et lav-energi latent rum (LOLS) og densitets funktionel teori (DFT) til at søge efter molekylære konformere.
Ny maskinlæringstilgang letter molekylær konformersøgning i komplekse molekylerKredit:Aalto Universitet CEST-forskere udviklede en ny maskinlæringstilgang baseret på et lav-energi latent rum (LOLS) og densitets funktionel teori (DFT) til at søge efter molekylære konformere. -
 Hvor stort er et fragment?Til venstre:et Prince Ruperts drop. Til højre:når dråbens hale klemmes, den bryder i utallige bittesmå fragmenter - alle nogenlunde samme størrelse. Kredit:S. Kooij et al., Naturkommunikation Når
Hvor stort er et fragment?Til venstre:et Prince Ruperts drop. Til højre:når dråbens hale klemmes, den bryder i utallige bittesmå fragmenter - alle nogenlunde samme størrelse. Kredit:S. Kooij et al., Naturkommunikation Når -
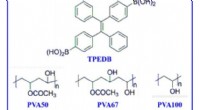 Storskala fremstilling af polymerbaseret stuetemperatur phosphorescens via klikkemiMolekylformler for fosfor og polymermatricer. De molekylære formler for TPEDB, PVA med forskellig alkohollysegrad (PVA50, PVA67, PVA100), og kontrollerede polymerer (PDDA, PSS, og PVDF). Kredit:Scienc
Storskala fremstilling af polymerbaseret stuetemperatur phosphorescens via klikkemiMolekylformler for fosfor og polymermatricer. De molekylære formler for TPEDB, PVA med forskellig alkohollysegrad (PVA50, PVA67, PVA100), og kontrollerede polymerer (PDDA, PSS, og PVDF). Kredit:Scienc
- Undersøgelsesprojekter med brug af kokosnød Husk
- Synkronsvømning:Biologi på mikroskala
- Hvordan gør Pandas Mate?
- 13, 000-årige menneskelige fodaftryk fundet ud for Canadas Stillehavskyst
- Den ene ring – for at spore dine fingres placering
- Undersøgelse finder chloroformemissioner, i fremmarch i Østasien, kan forsinke ozonindvindingen me…


