Virkelig tilfældige netværk

Farverne repræsenterer nodernes identiteter. Den øverste række viser, hvor mange forbindelser hver node har. I cirklen kan du se alle mulige måder at lave en sammenhængende graf ud af disse noder. Den nye algoritme kan vælge en af disse tilfældigt og gentagne gange. Kredit:Szabolcs Horvat et al. 2020/MPI-CBG/CSBD
Mange naturlige og menneskeskabte netværk, såsom computer, biologiske eller sociale netværk har en forbindelsesstruktur, der kritisk former deres adfærd. Det akademiske felt af netværksvidenskab beskæftiger sig med at analysere sådanne komplekse netværk i den virkelige verden og forstå, hvordan deres struktur påvirker deres funktion eller adfærd. Eksempler er det vaskulære netværk af vores kroppe, netværket af neuroner i vores hjerne, eller netværket af, hvordan en epidemi spreder sig gennem et samfund.
Behovet for pålidelige null-modeller
Analysen af sådanne netværk fokuserer ofte på at finde interessante egenskaber og funktioner. For eksempel, hjælper strukturen af et bestemt kontaktnetværk sygdomme med at sprede sig særligt hurtigt? For at finde ud af, vi har brug for en baseline - et sæt tilfældige netværk, en såkaldt 'nul-model' — at sammenligne med. Desuden, da flere forbindelser åbenbart skaber flere muligheder for infektion, antallet af forbindelser for hver node i basislinjen skal matches til det netværk, vi analyserer. Så hvis vores netværk ser ud til at lette spredningen mere end basislinjen, vi ved, at det må skyldes dens specifikke netværksstruktur. Imidlertid, skaber virkelig tilfældige, upartisk, null-modeller, der matches i nogle ejendomme, er vanskelige - og kræver normalt en anden tilgang for hver ejendom af interesse. Eksisterende algoritme, der skaber forbundne netværk med et specifikt antal forbindelser for hver knude, lider alle af ukontrolleret bias, hvilket betyder, at nogle netværk genereres mere end andre, potentielt kompromittere undersøgelsens konklusioner.
En ny metode, der eliminerer bias
Szabolcs Horvát og Carl Modes ved Center for Systembiologi Dresden (CSBD) og Max Planck Institute of Molecular Cell Biology and Genetics (MPI-CBG) udviklede en sådan model, som gør det muligt at eliminere bias, og nå frem til solide konklusioner. Szabolcs Horvát siger, "Vi udviklede en nul-model for tilsluttede netværk, hvor skævheden er under kontrol og kan udregnes. vi skabte en algoritme, der kan generere tilfældigt forbundne netværk med et foreskrevet antal forbindelser for hver node. Med vores metode, vi viste, at mere naive, men almindeligt anvendte tilgange kan føre til ugyldige konklusioner." Den koordinerende forfatter af undersøgelsen, Carl Modes konkluderer:"Dette fund illustrerer behovet for matematisk velfunderede metoder. Vi håber, at vores arbejde vil være nyttigt for det bredere netværksvidenskabelige samfund. For at gøre det så nemt som muligt for andre forskere at bruge det, vi udviklede også en software og gjorde den offentlig tilgængelig."
 Varme artikler
Varme artikler
-
 Forskere opdager en ny måde at kontrollere infrarødt lys påDenne 8-tommer wafer indeholder faseændringspixels, der kan styres til at modulere lys. Forskere studerer egenskaberne og adfærden af pixels for at informere om skabelsen af fremtidige enheder, de
Forskere opdager en ny måde at kontrollere infrarødt lys påDenne 8-tommer wafer indeholder faseændringspixels, der kan styres til at modulere lys. Forskere studerer egenskaberne og adfærden af pixels for at informere om skabelsen af fremtidige enheder, de -
 Virkelige anvendelser til ellipserFormen på en ellips dannes, når en kegle skæres i en vinkel. Hvis du vipper et glas vand, er den resulterende form på vandoverfladen også en ellipse. Du kan også se ellipser, når en hula hoop eller
Virkelige anvendelser til ellipserFormen på en ellips dannes, når en kegle skæres i en vinkel. Hvis du vipper et glas vand, er den resulterende form på vandoverfladen også en ellipse. Du kan også se ellipser, når en hula hoop eller -
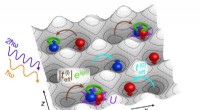 En metode til at simulere stærkt korrelerede faser af kvantemålingsteorierGitterpotentiale i x -z -planet bestående af individuelle dimerer med en energiskævhed Δ0. Gitterpositionen er sinusformet moduleret i x -retningen ved to frekvenser ω/(2π) og 2ω/(2π) ved hjælp af en
En metode til at simulere stærkt korrelerede faser af kvantemålingsteorierGitterpotentiale i x -z -planet bestående af individuelle dimerer med en energiskævhed Δ0. Gitterpositionen er sinusformet moduleret i x -retningen ved to frekvenser ω/(2π) og 2ω/(2π) ved hjælp af en -
 AlphaZero lærer at styre kvanteverdenenKredit:CC0 Public Domain Skakverdenen var forbløffet, da computeralgoritmen AlphaZero lærte, efter bare fire timer alene at slå de bedste skakprogrammer bygget på menneskelig ekspertise. Nu har en
AlphaZero lærer at styre kvanteverdenenKredit:CC0 Public Domain Skakverdenen var forbløffet, da computeralgoritmen AlphaZero lærte, efter bare fire timer alene at slå de bedste skakprogrammer bygget på menneskelig ekspertise. Nu har en
- Fysikere forudsiger ikke-metallisk halvmetallicitet
- Døgn -uret sætter tempoet for plantevækst
- Gamle klipper giver spor til Jordens tidlige historie
- Vil vi nyde vores arbejde mere, når rutineopgaver er automatiseret? Ikke nødvendigvis, foreslår e…
- Pilotskalaanlæg til udvinding af sjældne jordartselementer i hjertet af kullandet
- Spotify bliver offentliggjort den 3. april


