Forskere udvikler værktøjer til at skærpe 3D-visning af store RNA-molekyler
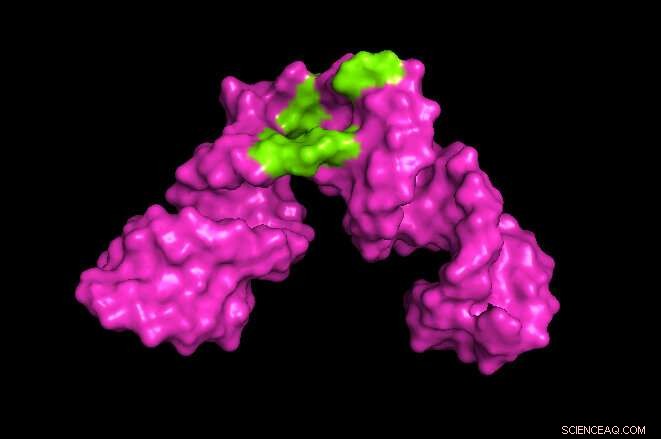
University of Maryland forskere udviklede en metode til at udvide omfanget af nuklear magnetisk resonans (NMR) spektroskopi. I eksemplet ovenfor, forskerne var i stand til at skabe et 3D-billede, der afslørede stedet på et stykke hepatitis-RNA, hvor små molekyler, såsom et lægemiddel, kunne binde (vist med grønt). Kredit:Kwaku Dayie/University of Maryland
Forskere fra University of Maryland har udviklet en metode til at bestemme strukturerne af store RNA-molekyler ved høj opløsning. Metoden overvinder en udfordring, der har begrænset 3D-analyse og billeddannelse af RNA til kun små molekyler og stykker af RNA i de sidste 50 år.
Den nye metode, som udvider omfanget af nuklear magnetisk resonans (NMR) spektroskopi, vil gøre det muligt for forskere at forstå formen og strukturen af RNA-molekyler og lære, hvordan de interagerer med andre molekyler. Den indsigt, som denne teknologi giver, kan føre til målrettede RNA-terapeutiske behandlinger for sygdom. Forskningspapiret om dette arbejde blev offentliggjort i tidsskriftet Videnskabens fremskridt den 7. oktober, 2020.
"Kernemagnetisk resonansspektroskopi har siddet fast og kigget på ting, der er små, sige 35 RNA-byggesten eller nukleotider. Men de fleste af de interessante ting, der er biologisk og medicinsk relevante, er meget større, 100 nukleotider eller mere, " sagde Kwaku Dayie, en professor i kemi og biokemi ved UMD og seniorforfatter af papiret. "Så, at kunne nedbryde træstammen og se på ting, der er store, er meget spændende. Det vil give os mulighed for at kigge ind i disse molekyler og se, hvad der foregår på en måde, vi ikke har været i stand til før."
I NMR-spektroskopi, videnskabsmænd retter radiobølger mod et molekyle, spændende atomerne og "lyser op" molekylet. Ved at måle ændringer i magnetfeltet omkring de exciterede atomer - den nukleare magnetiske resonans - kan videnskabsmænd rekonstruere karakteristika såsom formen, molekylets struktur og bevægelse. De data, dette producerer, kan derefter bruges til at generere billeder, meget som MR-billeder set i medicin.
Normalt, NMR-signaler fra de mange atomer i et biologisk molekyle såsom RNA overlapper hinanden, gør analysen meget vanskelig. Imidlertid, i 1970'erne, forskere lærte at biokemisk konstruere RNA-molekyler til at fungere bedre med NMR ved at erstatte brintatomerne med magnetisk aktive fluoratomer. I relativt små RNA-molekyler bestående af 35 eller færre nukleotider, fluoratomerne lyser let op, når de rammes af radiobølger og forbliver ophidsede længe nok til analyse i høj opløsning. Men efterhånden som RNA-molekyler bliver større, fluoratomerne lyser kun kortvarigt, mister derefter hurtigt deres signal. Dette har forhindret højopløsnings 3D-analyse af større RNA-molekyler.
Tidligere arbejde fra andre havde vist, at fluor fortsatte med at producere et stærkt signal, når det var ved siden af et kulstofatom indeholdende seks protoner og syv neutroner (C-13). Så, Dayie og hans team udviklede en relativt nem metode til at ændre det naturligt forekommende C-12 i RNA (som har 6 protoner og 6 neutroner) til C-13 og installere et fluoratom (F-19) direkte ved siden af det.
Dayie og hans team demonstrerede først, at deres metode kunne producere data og billeder svarende til nuværende metoder ved at anvende det på stykker af RNA fra HIV indeholdende 30 nukleotider, som tidligere var blevet afbildet. De anvendte derefter deres metode på stykker af hepatitis B-RNA indeholdende 61 nukleotider - næsten dobbelt så stor som tidligere mulig NMR-spektroskopi for RNA.
Deres metode gjorde det muligt for forskerne at identificere steder på hepatitis B-RNA'et, hvor små molekyler binder og interagerer med RNA'et. Det kunne være nyttigt for at forstå effekten af potentielle terapeutiske lægemidler. Det næste skridt for forskerne er at analysere endnu større RNA-molekyler.
"Dette arbejde giver os mulighed for at udvide det, der kan bringes i fokus, " sagde Dayie. "Vores beregninger fortæller os, at i teorien, vi kan se på virkelig store ting, som en del af ribosomet, som er den molekylære maskine, der syntetiserer proteiner inde i celler."
Ved at forstå formen og strukturen af et molekyle, forskere kan bedre forstå dens funktion, og hvordan den interagerer med sit miljø. Hvad mere er, denne teknologi vil gøre det muligt for forskere at se 3D-strukturen, når den ændrer sig, fordi især RNA-molekyler skifter form hyppigt. Denne viden er nøglen til at udvikle terapeutika, der snævert målretter mod sygdomsspecifikke molekyler uden at påvirke sunde cellefunktioner.
"Håbet er, at hvis forskerne kender afkrogene i et molekyle, der er dysfunktionelt, så kan de designe stoffer, der fylder afkroge og kan tage det ud af drift, " sagde Dayie. "Og hvis vi kan følge disse molekyler, når de ændrer form og struktur, så vil deres reaktion på potentielle stoffer være en smule mere forudsigelig, og det kan være mere effektivt at designe lægemidler, der er effektive."
Sidste artikelKatalysator for bæredygtig gassyntese
Næste artikel2 forskere vinder Nobels kemipris for genredigeringsværktøj
 Varme artikler
Varme artikler
-
 Varme elektroner sender kuldioxid tilbage til fremtidenKatalysatornanopartiklerne udviklet af KAUST-forskere bruger lysenergi til at omdanne kuldioxid og brint til metan. Kredit:KAUST; Anastasia Serin Atmosfærisk kuldioxid (CO 2 ) er en væsentlig dr
Varme elektroner sender kuldioxid tilbage til fremtidenKatalysatornanopartiklerne udviklet af KAUST-forskere bruger lysenergi til at omdanne kuldioxid og brint til metan. Kredit:KAUST; Anastasia Serin Atmosfærisk kuldioxid (CO 2 ) er en væsentlig dr -
 Lagring af data som blandinger af fluorescerende farvestofferBlandinger af fluorescerende farvestofmolekyler, placeret små steder på en epoxyoverflade med en inkjetprinter, kode data. Kredit:American Chemical Society. Efterhånden som verdens behov for datal
Lagring af data som blandinger af fluorescerende farvestofferBlandinger af fluorescerende farvestofmolekyler, placeret små steder på en epoxyoverflade med en inkjetprinter, kode data. Kredit:American Chemical Society. Efterhånden som verdens behov for datal -
 Forskere ser på olie og vand interaktion for at forhindre vandforureningKredit:Jim Young, UIC Olieudslip i havet har en ødelæggende indvirkning på miljøet. Mere end 400, 000 liter olie spildes i havene hvert år, påvirker hundredvis af kvadratkilometer og dræber mere e
Forskere ser på olie og vand interaktion for at forhindre vandforureningKredit:Jim Young, UIC Olieudslip i havet har en ødelæggende indvirkning på miljøet. Mere end 400, 000 liter olie spildes i havene hvert år, påvirker hundredvis af kvadratkilometer og dræber mere e -
 Polyproline beskytter cellemonolag mod fryseskaderKredit:Wiley Naturen har udviklet sukkerarter, aminosyrer, og specielle frostvæskeproteiner som kryobeskyttelsesmidler. Folk bruger organiske opløsningsmidler og syntetiske polymerer som tilsætnin
Polyproline beskytter cellemonolag mod fryseskaderKredit:Wiley Naturen har udviklet sukkerarter, aminosyrer, og specielle frostvæskeproteiner som kryobeskyttelsesmidler. Folk bruger organiske opløsningsmidler og syntetiske polymerer som tilsætnin
- Ny undersøgelse afslører sammenhæng mellem klima, livet og kontinenternes bevægelse
- Hvordan man laver skummetmælkagarplader
- Guldlok og de tre kvanteprikker:Lige tilpas til maksimal solpanelydelse
- Bump på et plot fra Chandra X-ray observatory afslører overskydende røntgenstråler, antyder mør…
- Otte trin til en stærkere cybersikkerhedsstrategi
- Hvad er den sikreste energikilde?


