Forskere skitserer metode til DNA -beregning i ny bog
Forskere ved New York University's Courant Institute of Mathematical Sciences har skitseret en metode til lagring af programmer inde i DNA, der forenkler nanocomputing - beregning på molekylært niveau. Medforfatter af Jessie Chang og Dennis Shasha, Lagrede programmer i DNA:En forenklet ramme for nanocomputing (Morgan og Claypool) beskriver, hvordan man bygger millioner af DNA -programmer, hvorfra instruktioner kan skrælles væk ad gangen fra hvert program i synkronisering.
Motivationen for dette arbejde ligner den for lagrede programmer inde i din bærbare computer. Inden computere, der var mekaniske lommeregnere, hvor enkeltpersoner ville slå nøgler efter en procedure, og et nummer til sidst ville blive vist. Når lommeregnere blev hurtigere, det blev klart, at det, der trængte til forbedring, var stansningsprocessen, ikke beregningsgraden. At gøre dette, de første computerdesignere lagrede programmerne indeholdende "stanse" instruktioner inde i maskiner, så de kunne køre på egen hånd. Når disse instruktioner var gemt, hele beregningen kunne køre med maskinens hastighed.
Lagrede programmer med ur inde i DNA tilbyder en vej til at gøre det samme for DNA -computing. Mens computere er afhængige af data gemt i strenge på 0'er og 1'er, DNA - livets byggesten - gemmer information i molekylerne ("baser") repræsenteret af A, T, C, og G. To enkelt DNA -strenge vil binde, hvis hvert A i en streng er på linje med hver T i den anden og tilsvarende for Cs og Gs. Hvis kun nogle af baserne i streng s1 er tilpasset deres yndlingspartnere i s2, så vil en anden streng s3 med bedre justering skubbe s1 af vejen. Dette fænomen "forskydning" gør det muligt for forskere at oprette DNA -skulpturer og nanorobotter. Imidlertid, som håndholdte lommeregnere, DNA -computing er i øjeblikket afhængig af at hælde reagensglas af DNA i et større reagensglas med DNA, hindrer dens hastighed og gør dets brug sart.
I deres bog, Shasha og Chang tilbyder en metode til at gemme DNA -instruktioner inde i en kemisk løsning på en måde, der tillader beregningsprocessen at køre i henhold til et globalt ur bestående af specielle DNA -strenge kaldet "kryds" og "tak". Hver gang et "kryds" og "tak" kommer ind i et DNA -rør, frigives en instruktionsstreng fra en instruktionsstak. Dette ligner den måde, hvorpå en urcyklus i en elektronisk computer får en ny instruktion til at komme ind i en behandlingsenhed. Så længe der forbliver tråde på stakken, den næste cyklus frigiver en ny instruktionsstreng. Uanset den faktiske streng eller komponent, der skal frigives ved et bestemt urtrin, "kryds" og "tak" tråde forbliver de samme - i virkeligheden, fungerer som en automatisk inputenhed og gør op med manuel dataindtastning.
Aidan Daly, en Harvard -bachelor på en sommerpraktik på NYU, arbejdede sammen med Shasha og Chang for at teste deres byggeproces i laboratoriet hos NYU Chemistry Professor Nadrian Seeman, der grundlagde og udviklede området DNA -nanoteknologi. Seemans kreationer-lige fra tredimensionelle DNA-strukturer til en DNA-samlebånd-giver ham mulighed for at arrangere stykker og danne specifikke molekyler på en nanoskala med en vis præcision, ligner den måde, hvorpå en robotbilfabrik kan få at vide, hvilken slags bil der skal laves.
 Varme artikler
Varme artikler
-
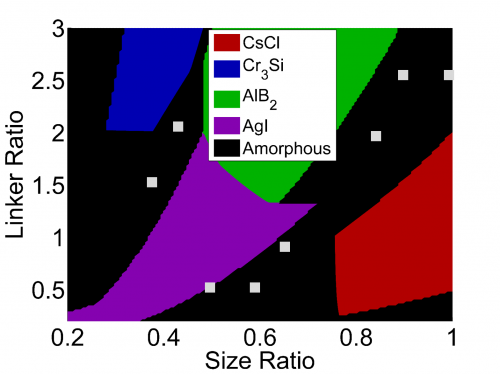 Brug af genetiske algoritmer til at opdage nye nanostrukturerede materialerDette er et fasediagram, der viser klyngeformationerne forudsagt af GA og deres validering (kvadrater). Kredit:Columbia Engineering Forskere ved Columbia Engineering, ledet af kemiingeniørprofesso
Brug af genetiske algoritmer til at opdage nye nanostrukturerede materialerDette er et fasediagram, der viser klyngeformationerne forudsagt af GA og deres validering (kvadrater). Kredit:Columbia Engineering Forskere ved Columbia Engineering, ledet af kemiingeniørprofesso -
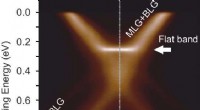 Grafen på vej mod superledningBåndstrukturen i et dobbeltlag af grafen blev scannet af ARPES ved synkrotronlyskilden BESSY II. Kredit:HZB Forskere ved HZB har fundet beviser for, at dobbeltlag af grafen har en egenskab, der ka
Grafen på vej mod superledningBåndstrukturen i et dobbeltlag af grafen blev scannet af ARPES ved synkrotronlyskilden BESSY II. Kredit:HZB Forskere ved HZB har fundet beviser for, at dobbeltlag af grafen har en egenskab, der ka -
 Åndbar, bærbar elektronik på huden til langsigtet sundhedsovervågningDiagrammet øverst illustrerer strukturen af guld nanomesh-ledere lamineret på hudoverfladen. Nanomesh, konstrueret af polyvinylalkohol (PVA) nanofibre og et guld (Au) lag, klæber til huden, når den
Åndbar, bærbar elektronik på huden til langsigtet sundhedsovervågningDiagrammet øverst illustrerer strukturen af guld nanomesh-ledere lamineret på hudoverfladen. Nanomesh, konstrueret af polyvinylalkohol (PVA) nanofibre og et guld (Au) lag, klæber til huden, når den -
 Intense lasere tilbereder komplekse, selvmonterede nanomaterialerBrookhaven Lab -videnskabsmand Kevin Yager (tv) og postdoktor Pawel Majewski med det nye Laser Zone Annealing -instrument på Center for Functional Nanomaterials. Nanoskala materialer har ekstraord
Intense lasere tilbereder komplekse, selvmonterede nanomaterialerBrookhaven Lab -videnskabsmand Kevin Yager (tv) og postdoktor Pawel Majewski med det nye Laser Zone Annealing -instrument på Center for Functional Nanomaterials. Nanoskala materialer har ekstraord
- Singapore-forskere udvikler billigere, men effektive tyndfilmsolceller
- Hvor i alverden er Salishhavet?
- Sådan fanges en sand Crab
- Hvorfor er der så få kvindelige administrerende direktører?
- NASA opsuger Tropical Storm Leslies vanddampkoncentration
- Nanopartikler kan forårsage DNA-skade på tværs af en cellulær barriere


