Unikt 4D -mikroskop fanger bevægelse af DNA -strukturer i rum og tid
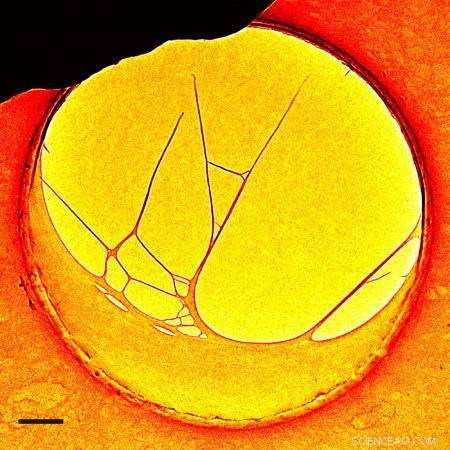
En DNA-struktur set gennem 4D-elektronmikroskopet opfundet hos Caltech. Kredit:Zewail &Lorenz/Caltech
(Phys.org) - Hver stor struktur, fra Empire State Building til Golden Gate Bridge, afhænger af specifikke mekaniske egenskaber for at forblive stærke og pålidelige. Stivhed - et materiales stivhed - er af særlig betydning for at opretholde den robuste funktionalitet for alt fra kolossale bygninger til de mindste af nanoskala strukturer. I biologiske nanostrukturer, som DNA-netværk, det har været svært at måle denne stivhed, som er afgørende for deres egenskaber og funktioner. Men forskere ved California Institute of Technology (Caltech) har for nylig udviklet teknikker til at visualisere adfærden af biologiske nanostrukturer i både rum og tid, giver dem mulighed for direkte at måle stivhed og kortlægge dens variation i hele netværket.
Den nye metode er beskrevet i den tidlige udgave af 4. februar Proceedings of the National Academy of Sciences ( PNAS ).
"Denne type visualisering tager os ind på domæner inden for de biologiske videnskaber, som vi ikke har udforsket før, siger nobelpristageren Ahmed Zewail, Linus Pauling professor i kemi og professor i fysik ved Caltech, der medforfatter avisen med Ulrich Lorenz, en postdoktor i Zewails laboratorium. "Vi leverer metoden til at finde ud af - direkte - stivheden af et biologisk netværk, der har egenskaber i nanoskala."
At kende de mekaniske egenskaber ved DNA -strukturer er afgørende for at bygge robuste biologiske netværk, blandt andre applikationer. Ifølge Zewail, denne type visualisering af biomekanik i rum og tid bør være anvendelig til undersøgelse af andre biologiske nanomaterialer, herunder de unormale proteinsamlinger, der ligger til grund for sygdomme som Alzheimers og Parkinsons.
Zewail og Lorenz var i stand til at se, for første gang, bevægelsen af DNA-nanostrukturer i både rum og tid ved hjælp af det firedimensionelle (4D) elektronmikroskop udviklet på Caltechs Physical Biology Center for Ultrafast Science and Technology. Centret ledes af Zewail, der skabte det i 2005 for at fremme forståelsen af den kemiske og biologiske adfærds grundlæggende fysik.
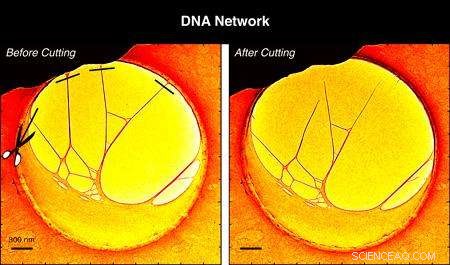
Ved at bruge elektronerne i mikroskopet, adskillige DNA-filamenter blev skåret væk fra carbonfilmen (til venstre), så en tredimensionel, fritstående struktur blev opnået under 4D mikroskop (højre).
"I naturen, materiens opførsel bestemmes af dets struktur - arrangementerne af dets atomer i rummets tre dimensioner - og af hvordan strukturen ændrer sig med tiden, den fjerde dimension, " forklarer Zewail. "Hvis du ser en hest galoppere i slowmotion, du kan følge galoppernes tid, og du kan se detaljeret hvad, for eksempel, hvert ben klarer sig over tid. Når vi kommer til nanometerskalaen, det er en anden historie - vi skal forbedre den rumlige opløsning til en milliard gange hestens for at visualisere, hvad der sker. "
Zewail blev tildelt Nobelprisen i kemi i 1999 for sin udvikling af femtokemi, som anvender ultrakorte laserblink til at observere grundlæggende kemiske reaktioner, der forekommer på femtosekundets tid (en milliontedel af en milliarddel af et sekund). Although femtochemistry can capture atoms and molecules in motion, giving the time dimension, it cannot concurrently show the dimensions of space, and thus the structure of the material. This is because it utilizes laser light with wavelengths that far exceed the dimension of a nanostructure, making it impossible to resolve and image nanoscale details in tiny physical structures such as DNA .
To overcome this major hurdle, the 4D electron microscope employs a stream of individual electrons that scatter off objects to produce an image. The electrons are accelerated to wavelengths of picometers, or trillionths of a meter, providing the capability for visualizing the structure in space with a resolution a thousand times higher than that of a nanostructure, and with a time resolution of femtoseconds or longer.
The experiments reported in PNAS began with a structure created by stretching DNA over a hole embedded in a thin carbon film. Using the electrons in the microscope, several DNA filaments were cut away from the carbon film so that a three-dimensional, free-standing structure was achieved under the 4D microscope.
Næste, the scientists employed laser heat to excite oscillations in the DNA structure, which were imaged using the electron pulses as a function of time—the fourth dimension. By observing the frequency and amplitude of these oscillations, a direct measure of stiffness was made.
"It was surprising that we could do this with a complex network, " says Zewail. "And yet by cutting and probing, we could go into a selective area of the network and find out about its behavior and properties."
Using 4D electron microscopy, Zewail's group has begun to visualize protein assemblies called amyloids, which are believed to play a role in many neurodegenerative diseases, and they are continuing their investigation of the biomechanical properties of these networks. He says that this technique has the potential for broad applications not only to biological assemblies, but also in the materials science of nanostructures.
 Varme artikler
Varme artikler
-
 Ny undersøgelse viser lovende i at bruge RNA-nanoteknologi til at behandle kræftformer og virusinf…University of Kentuckys Peixuan Guo betragtes som en af de tre bedste nanobioteknologiske eksperter i verden. Kredit:UK HealthCare En ny undersøgelse fra University of Kentucky forskere viser lø
Ny undersøgelse viser lovende i at bruge RNA-nanoteknologi til at behandle kræftformer og virusinf…University of Kentuckys Peixuan Guo betragtes som en af de tre bedste nanobioteknologiske eksperter i verden. Kredit:UK HealthCare En ny undersøgelse fra University of Kentucky forskere viser lø -
 Forskere tuner nanotrådsegenskaber med peptiddekorationerKredit:CC0 Public Domain I det seneste papir fra Geobacter Lab ledet af mikrobiolog Derek Lovley ved University of Massachusetts Amherst, han og kolleger rapporterer om et stort fremskridt i søgen
Forskere tuner nanotrådsegenskaber med peptiddekorationerKredit:CC0 Public Domain I det seneste papir fra Geobacter Lab ledet af mikrobiolog Derek Lovley ved University of Massachusetts Amherst, han og kolleger rapporterer om et stort fremskridt i søgen -
 Bakterier dræbes af ny lysaktiveret belægningDette fotografi afbilder Clostridium difficile-kolonier efter 48 timers vækst på en blodagarplade; Forstørret 4,8X. C. difficile, en anaerob gram-positiv stang, er den hyppigst identificerede årsag ti
Bakterier dræbes af ny lysaktiveret belægningDette fotografi afbilder Clostridium difficile-kolonier efter 48 timers vækst på en blodagarplade; Forstørret 4,8X. C. difficile, en anaerob gram-positiv stang, er den hyppigst identificerede årsag ti -
 Bandagemateriale hjælper med at stoppe blødningen uden at klæbe til såretFremmer helbredelse og kan efterfølgende let fjernes:en ny form for bandage belagt med silikone og carbon nanofibre. Kredit:Li Z et al. Naturkommunikation 2019 Forskere fra ETH Zürich og National
Bandagemateriale hjælper med at stoppe blødningen uden at klæbe til såretFremmer helbredelse og kan efterfølgende let fjernes:en ny form for bandage belagt med silikone og carbon nanofibre. Kredit:Li Z et al. Naturkommunikation 2019 Forskere fra ETH Zürich og National
- Måder at forudsige en storm med lufttryk
- Storbritannien skal bøde Facebook for brud på data (opdatering)
- WeWork sagsøger Japans SoftBank for at bakke ud af aftalen
- Sådan konverteres pund til Ounces
- Zoom udruller nye foranstaltninger, efterhånden som sikkerhedsangsten stiger
- Love tilbyder begrænset beskyttelse mod prisdifferentiering ved hjælp af algoritmer


