Sådan ser du levende maskiner
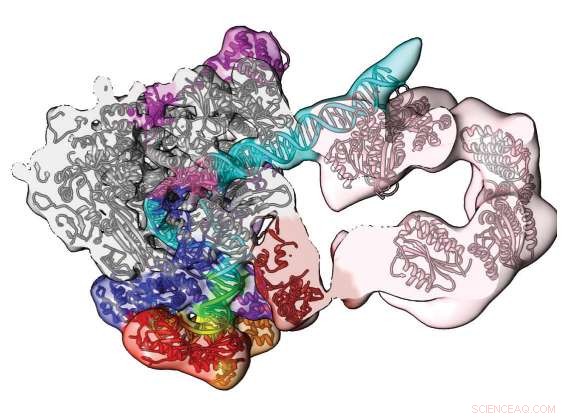
Supercomputing og kryo-elektronmikroskopi afslører denne del af det menneskelige præ-initieringskompleks. Det åbne konformationsdensitetskort og -model viser DNA'ets vej (blå/grøn) og dets engagement af transkriptionsfaktorkomponenten TFIIH (pink). Genoptrykt med tilladelse fra Macmillan Publishers Ltd:He, Y. et al. Nær-atomisk opløsningsvisualisering af human transkriptionspromotoråbning. Natur 533, 359-365 (2016).
Det lyder som noget ud af Borg i Star Trek. Robotter i nanostørrelse samler sig selv for at danne biologiske maskiner, der udfører det arbejde, der holder en i live. Og alligevel fortsætter sådan noget virkelig.
Hver celle i vores krop - det være sig kød og blod, hjerne og alt derimellem - har identisk DNA, den snoede trappe af nukleinsyrer kodet unikt for hver organisme. Komplekse samlinger, der ligner molekylære maskiner, tager stykker af DNA kaldet gener og laver en hjernecelle, når det er nødvendigt, i stedet for, sige, en knoglecelle. Disse molekylære maskiner er så komplekse, dog så lille, at videnskabsmænd i dag lige er begyndt at forstå deres struktur og funktion ved hjælp af de nyeste mikroskoper og supercomputere. Biologiske molekylære maskiner kunne lægge grundlaget for at udvikle kur mod sygdomme som kræft. Hvor lille kan man se, og hvad vil man finde?
Kryo-elektronmikroskopi kombineret med supercomputersimuleringer har skabt den bedste model til dato, med detaljer på næsten atomniveau, af en vital molekylær maskine, det menneskelige præ-initieringskompleks (PIC). Et videnskabshold fra Northwestern University, Berkeley National Laboratory, Georgia State University, og UC Berkeley offentliggjorde deres resultater på PIC maj 2016 i tidsskriftet Natur .
"For første gang, strukturer er blevet detaljeret af de komplekse grupper af molekyler, der åbner menneskets DNA, " sagde studie medforfatter Ivaylo Ivanov, lektor i kemi ved Georgia State University. Ivanov ledede det beregningsmæssige arbejde, der modellerede atomerne i de forskellige proteiner, der fungerer som tandhjul i PIC-molekylmaskinen.
PIC finder gener forbundet med fremstilling af et specifikt protein, såsom et antistof eller et enzym. Der trækker PIC'en de to DNA-strenge fra hinanden og føder den kodende streng til arbejdshestenzymet RNA-polymerase II. Dette starter transskription, hvor DNA-bits kopieres af RNA-polymerase II til en enkelt streng af messenger-RNA. RNA'et finder vej til 'proteinfabrikker' i cellen kaldet ribosomer, der tager dem som ordrer for, hvilket protein der skal fremstilles. Hvis DNA er som tegningen af et nyt hus, RNA'er er instruktioner til 'entreprenørerne' på ribosomarbejdsstationen. De fremstillede proteiner er som neglene, træ, gips, og næsten alt andet i huset.
Eksperimentet begyndte med billeder omhyggeligt taget af PIC. De blev lavet af en gruppe ledet af undersøgelsens medforfatter Eva Nogales, en professor i Institut for Molekylær og Cellulær Biologi ved UC Berkeley og også Senior Faculty Scientist ved Lawrence Berkeley National Laboratory og Howard Hughes Medical Investigator.
Nogales' gruppe brugte kryo-elektronmikroskopi (cryo-EM), en stigende stjerne inden for laboratorieteknikker. De frøs kryogenisk human PIC bundet til DNA. Indfrysningen holdt det i en kemisk aktiv, nær-naturligt miljø. Dernæst zappede de det med elektronstråler. Takket være de seneste fremskridt inden for direkte elektrondetektorteknologi, cryo-EM kan nu i næsten atomar opløsning afbilde store og komplicerede biologiske strukturer, som har vist sig at være for svære at krystallisere. Gå til teknikken, røntgen krystallografi, kræver krystalliserede prøver, og cryo-EM undgår dette hårde skridt.
Over 1,4 millioner cryo-EM 'fryse rammer' af PIC blev behandlet ved hjælp af supercomputere ved National Energy Research for Scientific Computing Center for at bortfiltrere baggrundsstøj og rekonstruerede tredimensionelle tæthedskort, der viser detaljer i form af molekylet, der aldrig havde været set før.
"Cryo-EM gennemgår en stor udvidelse, ligesom al den computersoftware, der bruges til at generere både tæthedskortene og også til at fortolke dem, som vi har gjort i denne undersøgelse, Nogales sagde. "Det giver os mulighed for at få højere opløsning af flere strukturer i forskellige stater, så vi ikke kun kan beskrive et billede af, hvordan de ser ud, men flere billeder, der viser, hvordan de bevæger sig. Vi ser ikke et kontinuum, men vi ser øjebliksbilleder gennem handlingsprocessen."
Undersøgelsesforskere byggede derefter en nøjagtig model, der gav fysisk mening med tæthedskortene for PIC ved hjælp af XSEDE, eXtream Science and Engineering Discovery Environment, finansieret af National Science Foundation. XSEDE giver forskere mulighed for interaktivt at dele computerressourcer, data og ekspertise via et enkelt virtuelt system. Ivaylo Ivanovs team har kørt over fire millioner kernetimers simuleringer på Stampede-supercomputeren ved Texas Advanced Computing Center for at modellere komplekse molekylære maskiner, inklusive dem til denne undersøgelse. Ivanovs bredere molekylære maskinarbejde inkluderer også en XSEDE-allokering på 1,7 millioner kernetimer på Comet-supercomputeren ved San Diego Supercomputing Center.
"Jeg har brugt XSEDE-ressourcer i mere end 12 år nu, " sagde Ivanov. "Uden tilgængeligheden af XSEDE-ressourcer, al vores forskning ville have været meget mere begrænset i forhold til de systemer, vi kan adressere. For os, XSEDE har været helt afgørende."
Målet med hele denne beregningsmæssige indsats er at producere atommodeller, der fortæller hele historien om strukturen og funktionen af proteinkomplekset af molekyler. For at komme dertil tog Ivanovs team de tolv komponenter i PIC-samlingen og skabte homologimodeller for hver komponent, der redegjorde for deres aminosyresekvenser og deres relation til lignende kendte protein 3-D strukturer.
Dernæst tilnærmede de de eksperimentelle tætheder, Nogales' hold fandt på et gitter. "Vi kan bruge en metode kaldet molekylær dynamik fleksibel tilpasning, " forklarede Ivanov, "hvor du i det væsentlige kører en molekylær dynamik-simulering. Og du bruger den eksperimentelle tæthed til at forspænde atomerne i den molekylære dynamik-simulering til at bevæge sig ind i de tættere områder af EM-kortet. Det er processen med fleksibel tilpasning til EM-kortet."
De forfinede modellen med Phoenix krystallografiske forfiningspakke. "Det er en gratis teknik, der giver os mulighed for at placere sidekæder og forbedre modellen, så vi kan fange alle de detaljer, der er til stede i tæthedskortet, " sagde Ivanov.
XSEDE var "absolut nødvendigt" for denne modellering, sagde Ivanov. "Når vi inkluderer vand og modioner ud over PIC-komplekset i en molekylær dynamik-simuleringsboks, vi får simuleringssystemets størrelse på over en million atomer. Man kan ikke køre det på en arbejdsstation eller endda på en beskeden klynge. For det skal vi virkelig gå til tusinde kerner. I dette tilfælde, vi gik op til to tusinde og otteogfyrre kerner. Og til det havde vi brug for adgang til Stampede, " sagde Ivanov.
En af indsigterne i undersøgelsen er en arbejdsmodel for, hvordan PIC åbner den ellers stabile DNA-dobbelthelix for transkription. Nogales forklarede, at man kunne forestille sig en snor lavet af to tråde snoet rundt om hinanden. Hold den ene ende meget stramt. Tag fat i den anden og drej den i den modsatte retning af tråden for at optrevle ledningen. Det er dybest set sådan, de levende maskiner, der holder os i live, gør det.
"DNA'et skal åbnes og flyttes ind i det aktive polymerasested for at kode for det første RNA-nukleotid, " forklarede Nogales. "Pre-initieringskomplekset holder de to strenge af DNA'et meget tæt sammen i den ene ende, så de ikke kan bevæge sig og de ikke kan åbne sig. På den anden side af PIC'en er der en maskine, der bruger energi til at skubbe DNA'et, vrid den i den modsatte retning, hvori de to tråde er trådet. Og når dette sker, mellem de to sider, trådene åbner sig, sagde Nogales.
Denne undersøgelse løste strukturen af den molekylære maskine, der fungerer som de snoede fingre, transkriptionsfaktorkomponenten TFIIH. "TFIIH har en translokase-underenhed, hvis rolle er samtidig at skubbe DNA'et mod polymerasens aktive sted og afvikle DNA'et. Ved det kombinerede skub og afvikling, effektivt adskiller du de to strenge af DNA'et, " sagde Ivanov.
Begge videnskabsmænd sagde, at de lige er begyndt at få en forståelse af transskription på atomniveau, afgørende for genekspression og i sidste ende sygdom. "Mange sygdomstilstande opstår, fordi der er fejl i, hvor meget et bestemt gen aflæses, og hvor meget et bestemt protein med en bestemt aktivitet i cellen er til stede, Nogales sagde. "Disse sygdomstilstande kan skyldes overdreven produktion af proteinet, eller omvendt ikke nok. Det er meget vigtigt at forstå den molekylære proces, der regulerer denne produktion, så vi kan forstå sygdomstilstanden."
"Dette arbejde illustrerer godt to generelle principper, der vil drive videnskaben i de næste par år, " kommenterede Peter Preusch, programmedarbejder hos National Institutes of Health (NIH). "Den ene er anvendelsen af hybridmetoder – kombinationer af biofysiske metoder, herunder røntgenkrystallografi og cryoEM sammen med beregningsmetoder i stor skala til at integrere information om større molekylære komplekser. To, der er et krav om, at teamvidenskab trækker ekspertise fra flere efterforskere til at løse problemer, der ikke kan løses af et enkelt laboratorium, der arbejder alene." Peter Preusch er biofysisk afdelingschef, Afdelingen for cellebiologi og biofysik, National Institute of General Medical Sciences, NIH.
Selvom dette grundlæggende arbejde ikke direkte producerer helbredelser, det lægger grundlaget for at hjælpe med at udvikle dem i fremtiden, sagde Ivanov. "For at forstå sygdom, vi er nødt til at forstå, hvordan disse komplekser fungerer i første omgang... Et samarbejde mellem beregningsmodeller og eksperimentelle strukturbiologer kan være meget frugtbart i fremtiden. "
Nature Articles-undersøgelsen fra maj 2016 (DOI:10.1038/nature17970), "Næratomisk opløsning visualisering af menneskelig transkriptionspromotoråbning, " blev skrevet af Yuan He, Lawrence Berkeley National Laboratory og nu ved Northwestern University; Chunli Yan og Ivaylo Ivanov, Georgia State University; Jie Fang, Carla Inouye, Robert Tjian, Eva Nogales, UC Berkeley. Finansiering kom fra National Institute of General Medical Sciences (NIH) og National Science Foundation.
 Varme artikler
Varme artikler
-
 Selvfoldende 3-D lysfølsomme grafenarkitekturerMekanisme og alsidighed af selvfoldende SU8-film. a) Selvfoldende SU8-dobbeltlag, hvor SU8-bundlaget er fuldt tværbundet, og det øverste SU8-lag er delvist tværbundet. SU8 dobbeltlagsfilmen foldes opa
Selvfoldende 3-D lysfølsomme grafenarkitekturerMekanisme og alsidighed af selvfoldende SU8-film. a) Selvfoldende SU8-dobbeltlag, hvor SU8-bundlaget er fuldt tværbundet, og det øverste SU8-lag er delvist tværbundet. SU8 dobbeltlagsfilmen foldes opa -
 Simulering af elektroniske nanokomponenter til udviklings- og produktionsprocessenSkematisk visning af det indre af en gate-all-around Si nanotrådtransistor med en atomistisk opløsning. Farven på hvert atom svarer til dets effektive temperatur, viser selvopvarmende effekter tæt på
Simulering af elektroniske nanokomponenter til udviklings- og produktionsprocessenSkematisk visning af det indre af en gate-all-around Si nanotrådtransistor med en atomistisk opløsning. Farven på hvert atom svarer til dets effektive temperatur, viser selvopvarmende effekter tæt på -
 Nanoteknologi tilbyder en ny tilgang til at øge lagringskapaciteten for dielektriske kondensatorerEt diagram over den dielektriske kondensatorforskning udviklet af et University of Delaware-ledet forskerhold. Kredit:Kathy F. Atkinson/University of Delaware 21. oktober kl. 2015, var den dag, hv
Nanoteknologi tilbyder en ny tilgang til at øge lagringskapaciteten for dielektriske kondensatorerEt diagram over den dielektriske kondensatorforskning udviklet af et University of Delaware-ledet forskerhold. Kredit:Kathy F. Atkinson/University of Delaware 21. oktober kl. 2015, var den dag, hv -
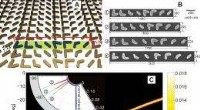 Plasmoniske nanoantenner viser løfte om optiske innovationerBilledet øverst til venstre viser en skematisk oversigt over en række guld plasmoniske nanoantenner, der er i stand til præcist at manipulere lys på nye måder, en teknologi, der kunne muliggøre en ræk
Plasmoniske nanoantenner viser løfte om optiske innovationerBilledet øverst til venstre viser en skematisk oversigt over en række guld plasmoniske nanoantenner, der er i stand til præcist at manipulere lys på nye måder, en teknologi, der kunne muliggøre en ræk
- Ny teknologi har til formål at forbedre smagen, holdbarhed, produktion af øl, mad
- Teamarbejde i et molekyle
- Fire NASA-sponsorerede eksperimenter skal lanceres på Virgin Galactic-rumfartøjet
- Undersøgelse viser, at GW190521 -begivenheden kunne forklares ved oprindelige sorte huller
- Hvad spiser kardinalfuglen?
- Forskere redigerer sommerfuglfløjpletter og -striber


