Fremtiden for datalagring er dobbeltspiralformet, viser forskning
Et dobbeltstrenget DNA-fragment. Kredit:Vcpmartin/Wikimedia/ CC BY-SA 4.0
Forestil dig Bachs "Cellosuite nr. 1" spillet på en DNA-streng.
Dette scenarie er ikke så umuligt, som det ser ud til. DNA er for lille til at modstå en rytmisk strygning eller glidende buestreng, og DNA er et kraftcenter til lagring af lydfiler og alle slags andre medier.
"DNA er naturens originale datalagringssystem. Vi kan bruge det til at gemme enhver form for data:billeder, video, musik - hvad som helst," sagde Kasra Tabatabaei, forsker ved Beckman Institute for Advanced Science and Technology og medforfatter på denne undersøgelse.
Udvidelse af DNA's molekylære sammensætning og udvikling af en præcis ny sekventeringsmetode gjorde det muligt for et multi-institutionelt team at transformere den dobbelte helix til en robust, bæredygtig datalagringsplatform.
Holdets papir udkom i Nano Letters i februar 2022.
I den digitale informations tidsalder føler enhver, der er modig nok til at navigere i de daglige nyheder, det globale arkiv bliver tungere dag for dag. Papirfiler bliver i stigende grad digitaliseret for at spare plads og beskytte information mod naturkatastrofer.
Lige fra videnskabsmænd til influentere på sociale medier, alle med information at opbevare står for at drage fordel af en sikker, bæredygtig datalåseboks – og den dobbelte helix passer til regningen.
"DNA er en af de bedste muligheder, hvis ikke den bedste mulighed, til især at gemme arkivdata," sagde Chao Pan, en kandidatstuderende ved University of Illinois Urbana-Champaign og medforfatter på denne undersøgelse.
Dens levetid konkurrerer kun med holdbarhed, DNA er designet til at klare Jordens hårdeste forhold - nogle gange i titusinder af år - og forblive en levedygtig datakilde. Forskere kan sekventere forstenede tråde for at afdække genetiske historier og puste liv i forsvundet landskab.
På trods af sin lille statur er DNA lidt ligesom Dr. Whos berygtede politiboks:større på indersiden, end det ser ud til.
"Hver dag bliver der genereret adskillige petabytes data på internettet. Kun et gram DNA ville være tilstrækkeligt til at gemme disse data. Så tæt er DNA som lagringsmedium," sagde Tabatabaei, der også er femteårs Ph. D. studerende.
Et andet vigtigt aspekt af DNA er dets naturlige overflod og næsten uendelige fornybarhed, en egenskab, der ikke deles af det mest avancerede datalagringssystem på markedet i dag:siliciummikrochips, som ofte cirkulerer i blot årtier før en uhøjtidelig begravelse i en bunke af deponeret e -affald.
"I en tid, hvor vi står over for hidtil usete klimaudfordringer, kan betydningen af bæredygtige lagringsteknologier ikke overvurderes. Nye grønne teknologier til DNA-optagelse dukker op, som vil gøre molekylær lagring endnu vigtigere i fremtiden," siger Olgica Milenkovic, Franklin W. Woeltge professor i elektro- og computerteknik og en co-PI på studiet.
Med en vision om fremtiden for datalagring undersøgte det tværfaglige team DNA's årtusind gamle MO. Derefter tilføjede forskerne deres eget twist fra det 21. århundrede.
I naturen indeholder hver DNA-streng fire kemikalier – adenin, guanin, cytosin og thymin – ofte refereret til med initialerne A, G, C og T. De arrangerer og omarrangerer sig selv langs den dobbelte helix i kombinationer, som videnskabsmænd kan afkode. , eller sekvens, for at skabe mening.
The researchers expanded DNA's already broad capacity for information storage by adding seven synthetic nucleobases to the existing four-letter lineup.
"Imagine the English alphabet. If you only had four letters to use, you could only create so many words. If you had the full alphabet, you could produce limitless word combinations. That's the same with DNA. Instead of converting zeroes and ones to A, G, C, and T, we can convert zeroes and ones to A, G, C, T, and the seven new letters in the storage alphabet," Tabatabaei said.
Because this team is the first to use chemically modified nucleotides for information storage in DNA, members innovated around a unique challenge:Not all current technology is capable of interpreting chemically modified DNA strands. To solve this problem, they combined machine learning and artificial intelligence to develop a first-of-its-kind DNA sequence readout processing method.
Their solution can discern modified chemicals from natural ones, and differentiate each of the seven new molecules from one another.
"We tried 77 different combinations of the 11 nucleotides, and our method was able to differentiate each of them perfectly," Pan said. "The deep learning framework as part of our method to identify different nucleotides is universal, which enables the generalizability of our approach to many other applications."
This letter-perfect translation comes courtesy of nanopores:proteins with an opening in the middle through which a DNA strand can easily pass. Remarkably, the team found that nanopores can detect and distinguish each individual monomer unit along the DNA strand—whether the units have natural or chemical origins.
"This work provides an exciting proof-of-principle demonstration of extending macromolecular data storage to non-natural chemistries, which hold the potential to drastically increase storage density in non-traditional storage media," said Charles Schroeder, the James Economy Professor of Materials Science and Engineering and a co-PI on this study.
DNA literally made history by storing genetic information. By the looks of this study, the future of data storage is just as double-helical. + Udforsk yderligere
Using DNA-like punch cards to store data
Sidste artikelEnginering af 2D-halvledere med indbyggede hukommelsesfunktioner
Næste artikelKontrollerer, hvor hurtigt grafen afkøles
 Varme artikler
Varme artikler
-
 Forskere skaber verdens første molekylære transistorIngeniører justerede spændingen påført via guldkontakter til et benzenmolekyle, giver dem mulighed for at hæve og sænke molekylets energitilstande og demonstrere, at det kunne bruges nøjagtigt som en
Forskere skaber verdens første molekylære transistorIngeniører justerede spændingen påført via guldkontakter til et benzenmolekyle, giver dem mulighed for at hæve og sænke molekylets energitilstande og demonstrere, at det kunne bruges nøjagtigt som en -
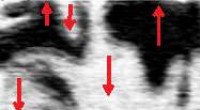 Undersøgelse af ferroelektriske domænevægge tilbyder en ny ledningsvej i nanoskalaSPM-billeder af (110) overfladen af spaltet h-HoMnO3. (øverst) PFM-billede, der viser ferroelektriske domæner i planet (orienteret lodret, røde pile). (nederst) cAFM-billede, der viser forbedret led
Undersøgelse af ferroelektriske domænevægge tilbyder en ny ledningsvej i nanoskalaSPM-billeder af (110) overfladen af spaltet h-HoMnO3. (øverst) PFM-billede, der viser ferroelektriske domæner i planet (orienteret lodret, røde pile). (nederst) cAFM-billede, der viser forbedret led -
 Nanorør fiberantenner lige så dygtige som kobberRice University kandidatstuderende Amram Bengio opsætter en nanorør fiberantenne til test. Forskere ved Rice og National Institute of Standards and Technology har fastslået, at nanorørfibre fremstille
Nanorør fiberantenner lige så dygtige som kobberRice University kandidatstuderende Amram Bengio opsætter en nanorør fiberantenne til test. Forskere ved Rice og National Institute of Standards and Technology har fastslået, at nanorørfibre fremstille -
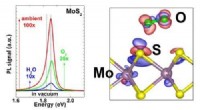 Forskere øger lysemissionen i 2D-halvledere med en faktor på 100(Venstre) Graf, der viser ændringen i fotoluminescens af MoS2 ved eksponering for H2O alene, O2 alene, og omgivende luft ved tryk på 7, 200, og 760 Torr, henholdsvis. (Højre) Figur, der viser ladnings
Forskere øger lysemissionen i 2D-halvledere med en faktor på 100(Venstre) Graf, der viser ændringen i fotoluminescens af MoS2 ved eksponering for H2O alene, O2 alene, og omgivende luft ved tryk på 7, 200, og 760 Torr, henholdsvis. (Højre) Figur, der viser ladnings
- NASA spioner vindskæring, der stadig påvirker tropiske stormnalger
- Fokuser på en forstærkende læringsalgoritme, der kan lære af fiasko
- Effektiv metode til fotokatalytiske fluoralkyleringer af (hetero)arener
- Jo rigere du er, jo mere sandsynligt er du social distance, undersøgelse finder
- USA sydder i sjælden efterårs hedebølge
- Abiotiske faktorer i Redwood Forest økosystem


