Højhastigheds atomkraftmikroskopi hjælper med at forklare den rolle, som visse biomolekyler spiller i DNA-indpakningsdynamik
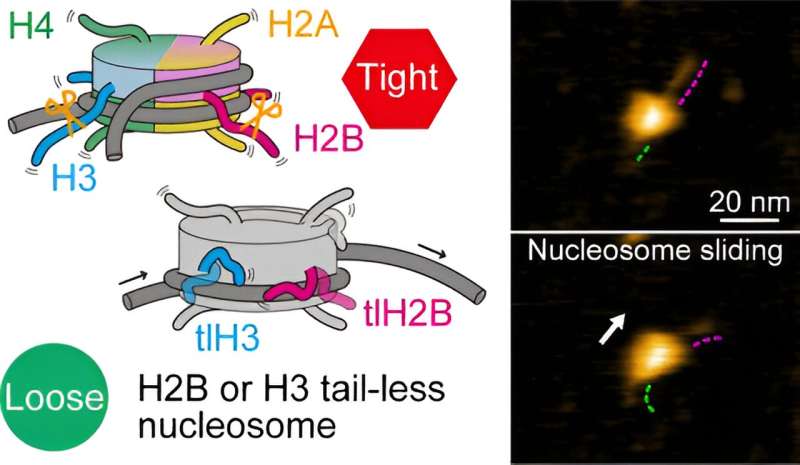
Hos planter og dyr er de grundlæggende pakkeenheder af DNA, som bærer genetisk information, de såkaldte nukleosomer. Et nukleosom består af et segment af DNA viklet omkring otte proteiner kendt som histoner.
Under genekspression (processen, der ligger til grund for proteinproduktion), er nukleosomer involveret i forskellige dynamiske strukturelle ændringer, såsom nukleosomglidning, DNA-udpakning og andre DNA-histon-interaktioner. Af særlig betydning i disse processer er endestrukturerne, eller haler, af histonerne.
Histonhaler gennemgår kemiske modifikationer, hvilket ændrer histonens funktionalitet efter behov. Detaljerede undersøgelser, og især visualiseringer, af nukleosomdynamik er afgørende for bedre at forstå histonhalernes rolle.
Mikihiro Shibata fra Kanazawa University og kolleger er nu lykkedes med at lave videooptagelser af haleløse nukleosomer, der viser, at fraværet af histonhaler markant øger et nukleosomes dynamiske aktivitet. Undersøgelsen er publiceret i Nano Letters .
Forskerne brugte højhastigheds atomkraftmikroskopi (HS-AFM), et kraftfuldt nanobilledværktøj til at visualisere molekylære strukturer og deres dynamik ved høj rumlig og tidsmæssig opløsning.
Til dette skulle nukleosomerne sættes på et substrat. Shibata og kolleger brugte en film af såkaldte søjle[5]arener (molekyler med en femkantet rørformet struktur) som substrat, hvilket danner en ideel overflade, da nukleosomerne let adsorberes til det, uden at dynamiske processer bliver undertrykt.
Forskerne så først på nukleosomer, hvor alle otte histoner manglede haler. Baseret på deres HS-AFM-observationer konkluderede de, at nukleosomglidning og DNA-udpakning/omvikling forekom oftere end for normale (kanoniske) nukleosomer. Dette tyder på, at uden haler svækkes histon-DNA-interaktionen, hvilket fører til en situation, hvor DNA lettere kan løsne sig fra histonerne.
For bedre at forstå rollerne af specifikke histonhaler, forberedte Shibata og kolleger nukleosomer, hvor en type histon var haleløs. Der er fire forskellige typer histoner, kaldet H2A, H2B, H3 og H4. HS-AFM eksperimenter på nukleosomerne afslørede, at H2B og H3 haleløse nukleosomer viste en øget frekvens af dynamik. Omvendt betyder det, at kanoniske H2B- og H3-histoner er afgørende for nukleosomstabilitet.
Forskerne påpeger, at de ikke kunne observere nogen faktisk bevægelse af histonhaler - højst sandsynligt var undersøgelsens tidsmæssige opløsning, 0,3 sekunder, meget langsommere end hastigheden af indpaknings-/udpakningsdynamikken i halerne. På trods af denne begrænsning beviser Shibatas og kollegers arbejde klart, at halerne af H2B- og H3-histoner er de vigtigste bidragydere til nukleosomdynamikken.
Med hensyn til fremtidigt arbejde, citerer forskerne, "en teknik til at mærke histonhalespidser kan gøre det muligt for HS-AFM at fange bevægelserne af histonhalerne selv."
Højhastigheds atomkraftmikroskopi
Det generelle princip for atomkraftmikroskopi (AFM) er at få en meget lille spids til at scanne overfladen af en prøve. Under denne vandrette (xy) scanning følger spidsen, som er fastgjort til en lille udkrager, prøvens lodrette (z) profil, hvilket inducerer en kraft på udkrageren, som kan måles.
Størrelsen af kraften ved xy-positionen kan relateres til z-værdien; de xyz-data, der genereres under en scanning, resulterer derefter i et højdekort, der giver strukturel information om den undersøgte prøve. I højhastigheds-AFM (HS-AFM) er arbejdsprincippet lidt mere involveret:cantileveren er lavet til at oscillere nær dens resonansfrekvens.
Når spidsen flyttes rundt på en overflade, registreres variationerne i amplituden (eller frekvensen) af cantileverens oscillation – som følge af spidsens interaktion med prøvens overflade – da disse giver et mål for den lokale z-værdi. AFM involverer ikke linser, så dens opløsning er ikke begrænset af den såkaldte diffraktionsgrænse som for eksempel ved røntgendiffraktion.
HS-AFM resulterer i en video, hvor tidsintervallet mellem billeder afhænger af den hastighed, hvormed et enkelt billede kan genereres (ved at xy-scanne prøven).
Forskere ved Nano Life Science Institute (WPI-NanoLSI), Kanazawa University har i de senere år udviklet HS-AFM yderligere, så det kan bruges til at studere biokemiske molekyler og biomolekylære processer i realtid. Mikihiro Shibata og kolleger har nu anvendt metoden til at studere nukleosomdynamikken i detaljer, og især rollen af de molekylære slutninger af histoner - proteiner, der spiller en afgørende rolle i DNA-tilgængelighed.
Flere oplysninger: Shin Morioka et al., High-Speed Atomic Force Microscopy afslører nukleosomets glidning og DNA-udpakning/-indpakningsdynamik af haleløse nukleosomer, nanobogstaver (2024). DOI:10.1021/acs.nanolett.4c00801
Journaloplysninger: Nanobreve
Leveret af Kanazawa University
 Varme artikler
Varme artikler
-
 Forskere udvikler metoder til at fremskynde opdagelse af infektionssygdomme, KræftUCLA-forskere var i stand til at bruge en molekylær kædereaktion til at påvise tilstedeværelsen af proteiner i blod og plasma på en måde, der er hurtigere og enklere. Kredit:Donghyuk Kim/UCLA Et
Forskere udvikler metoder til at fremskynde opdagelse af infektionssygdomme, KræftUCLA-forskere var i stand til at bruge en molekylær kædereaktion til at påvise tilstedeværelsen af proteiner i blod og plasma på en måde, der er hurtigere og enklere. Kredit:Donghyuk Kim/UCLA Et -
 Forskere opdager en lille drejning i tolagsgrafen, der kan løse et mysteriumDirac-spektret af dobbeltlagsgrafen, når de to lag er nøjagtigt justeret (venstre) skifter med en let mellemlagsdrejning, der bryder mellemlagskobling og potentiel symmetri, fører til et nyt spektrum
Forskere opdager en lille drejning i tolagsgrafen, der kan løse et mysteriumDirac-spektret af dobbeltlagsgrafen, når de to lag er nøjagtigt justeret (venstre) skifter med en let mellemlagsdrejning, der bryder mellemlagskobling og potentiel symmetri, fører til et nyt spektrum -
 Naturen inspirerer den første kunstige molekylære pumpeEn plan for en kunstig molekylær pumpe, der fungerer til at opdele ringe i en højenergitilstand på en polymethylenkæde. Kredit: Natur nanoteknologi (2015) doi:10.1038/nnano.2015.96 At bruge natur
Naturen inspirerer den første kunstige molekylære pumpeEn plan for en kunstig molekylær pumpe, der fungerer til at opdele ringe i en højenergitilstand på en polymethylenkæde. Kredit: Natur nanoteknologi (2015) doi:10.1038/nnano.2015.96 At bruge natur -
 Kulstof nanorør som transistormaterialeEt enkelt carbon nanorør (CNT) dyrkes mellem to kontakter. Enderne blev belagt ved dampaflejring af palladium for at forbinde til den suspenderede transistorkanal. (Foto:M. Muoth / ETH Zürich) Sch
Kulstof nanorør som transistormaterialeEt enkelt carbon nanorør (CNT) dyrkes mellem to kontakter. Enderne blev belagt ved dampaflejring af palladium for at forbinde til den suspenderede transistorkanal. (Foto:M. Muoth / ETH Zürich) Sch
- Hvordan brugen af forskellige former for titaniumoxid påvirker perovskit-solcellens ydeevne
- Hvordan man identificerer edderkopper fundet i Indiana
- Hunde kan skræmme (og endda dræbe) dyreliv. Sådan bliver du en ansvarlig ejer denne sommer
- En ny teknik til fremstilling af spændende metaloxidskeletter
- Spring frem i opdagelsen og udviklingen af nye antibiotika
- Ældste DNA fra domesticeret amerikansk hest giver troværdighed til skibbrudsfolklore


