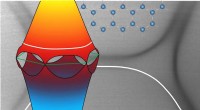
Mikroskopimetoden overvinder den traditionelle opløsningsgrænse for hurtig co-tracking af molekyler

Forskere ved Ludwig Maximilian University (LMU) har udviklet en innovativ metode til samtidig sporing af hurtige dynamiske processer af flere molekyler på molekylær skala.
Processer i vores kroppe er karakteriseret ved samspillet mellem forskellige biomolekyler såsom proteiner og DNA. Disse processer forekommer på en skala, ofte inden for et område på blot nogle få nanometer. De kan derfor ikke observeres med fluorescensmikroskopi, som har en opløsningsgrænse på omkring 200 nanometer på grund af diffraktion.
Når to farvestoffer, der markerer positioner af biomolekyler, er tættere på end denne optiske grænse, kan deres fluorescens ikke skelnes under mikroskopet. Da denne fluorescens bruges til at lokalisere dem, bliver nøjagtig bestemmelse af deres positioner umulig.
Denne opløsningsgrænse er traditionelt blevet overvundet i superopløsningsmikroskopimetoder ved at få farvestofferne til at blinke og tænde og slukke for deres fluorescens. Dette adskiller deres fluorescens midlertidigt, hvilket gør det muligt at skelne og muliggøre lokaliseringer under den klassiske opløsningsgrænse.
Men for applikationer, der involverer studiet af hurtige dynamiske processer, har dette trick en betydelig ulempe:blink forhindrer samtidig lokalisering af flere farvestoffer. Dette reducerer den tidsmæssige opløsning markant, når man undersøger dynamiske processer, der involverer flere biomolekyler.
Under ledelse af LMU-kemiker professor Philip Tinnefeld og i samarbejde med professor Fernando Stefani (Buenos Aires) har forskere ved LMU nu udviklet pMINFLUX multiplexing, en elegant tilgang til at løse dette problem.
Holdet har offentliggjort en artikel om deres metode i tidsskriftet Nature Photonics .
MINFLUX er en superopløsningsmikroskopimetode, der muliggør lokaliseringer med præcision på kun en nanometer. I modsætning til konventionel MINFLUX registrerer pMINFLUX tidsforskellen mellem excitationen af farvestoffer med en laserpuls og den efterfølgende fluorescens med sub-nanosekunders opløsning.
Ud over at lokalisere farvestofferne giver dette indsigt i en anden grundlæggende egenskab ved deres fluorescens:deres fluorescenslevetid. Dette beskriver, hvor lang tid det i gennemsnit tager for et farvestofmolekyle at fluorescere, efter at det er exciteret.
"Fluorescenslevetiden afhænger af det anvendte farvestof," forklarer Fiona Cole, medforfatter af publikationen. "Vi udnyttede forskelle i fluorescenslevetider, når vi brugte forskellige farvestoffer til at tildele de fluorescerende fotoner til farvestoffet, der udsendte uden behov for at blinke og den resulterende tidsmæssige adskillelse."
Til dette formål tilpassede forskerne lokaliseringsalgoritmen og inkluderede en multieksponentiel tilpasningsmodel for at opnå den nødvendige adskillelse.
"Dette gjorde det muligt for os at bestemme positionen af flere farvestoffer samtidigt og undersøge hurtige dynamiske processer mellem flere molekyler med nanometerpræcision," tilføjer Jonas Zähringer, også med-førsteforfatter.
Forskerne demonstrerede deres metode ved nøjagtigt at spore to DNA-strenge, mens de hoppede mellem forskellige positioner på en DNA-origami-nanostruktur, såvel som ved at adskille translationelle og roterende bevægelser af en DNA-origami-nanostruktur og ved at måle afstanden mellem antigenbindingssteder for antistoffer.
"Men det her er kun begyndelsen," siger Philip Tinnefeld. "Jeg er sikker på, at pMINFLUX multipleksing med sin høje tidsmæssige og rumlige opløsning vil give ny indsigt i proteininteraktioner og andre biologiske fænomener i fremtiden."
Flere oplysninger: Fiona Cole et al., Superopløst FRET og co-tracking i pMINFLUX, Nature Photonics (2024). DOI:10.1038/s41566-024-01384-4
Journaloplysninger: Naturfotonik
Leveret af Ludwig Maximilian University of München
 Varme artikler
Varme artikler
-
 Forskere øger lægemidlets effektivitet ved at bruge nanopartikler til at målrette cellernes kraft…Shanta Dhar, ret, en assisterende professor i kemi ved UGA Franklin College of Arts and Sciences, og ph.d.-studerende Sean Marrache har fremstillet nanopartikler, der øger effektiviteten af lægemidl
Forskere øger lægemidlets effektivitet ved at bruge nanopartikler til at målrette cellernes kraft…Shanta Dhar, ret, en assisterende professor i kemi ved UGA Franklin College of Arts and Sciences, og ph.d.-studerende Sean Marrache har fremstillet nanopartikler, der øger effektiviteten af lægemidl -
 En teknik til at overdope grafen ud over van Hove-singularitetenFor første gang, grafen er elektron-doteret forbi sin van Hove-singularitet. Til venstre:modellerede energiniveauer af grafen med eksemplarisk Fermi-niveau (hvid kurve) ud over singulariteten. Baggrun
En teknik til at overdope grafen ud over van Hove-singularitetenFor første gang, grafen er elektron-doteret forbi sin van Hove-singularitet. Til venstre:modellerede energiniveauer af grafen med eksemplarisk Fermi-niveau (hvid kurve) ud over singulariteten. Baggrun -
 Fysikere kaster lys over nanoskala -dynamikken ved centrifugering af centrifugeringBilledet repræsenterer et system med nukleare spins, hvis interaktioner medieres af elektronspins. Kredit:City University of New York I fysikken, termalisering, eller tendensen med delsystemer ind
Fysikere kaster lys over nanoskala -dynamikken ved centrifugering af centrifugeringBilledet repræsenterer et system med nukleare spins, hvis interaktioner medieres af elektronspins. Kredit:City University of New York I fysikken, termalisering, eller tendensen med delsystemer ind -
 Forskere har skabt en matematisk model for dynamikken af nanopartikler og vira i cellerDensitetsfordelingen i stationær tilstand fungerer ns(x) ved forskellige I(x) og ν (tal ved kurverne). Kredit:Krystaller (2022). DOI:10.3390/cryst12081159 Fysikere og matematikere fra Ural Federal
Forskere har skabt en matematisk model for dynamikken af nanopartikler og vira i cellerDensitetsfordelingen i stationær tilstand fungerer ns(x) ved forskellige I(x) og ν (tal ved kurverne). Kredit:Krystaller (2022). DOI:10.3390/cryst12081159 Fysikere og matematikere fra Ural Federal
- Mikroskop på en chip kunne bringe medicinsk ekspertise til fjerne patienter
- Undersøgelse afslører sammenhænge mellem unges ofre, overbevisning om regeringen, og politisk del…
- Er atomkraft sikker?
- Facebook gør reality-tv til sit nye våben for weboverherredømme
- Ny forskning kan bidrage til en bedre diagnosticering af kræft
- Nanoteknologi lover sikrere brystimplantater


