In-situ måling af 3-D proteinstruktur inde i levende eukaryote celler
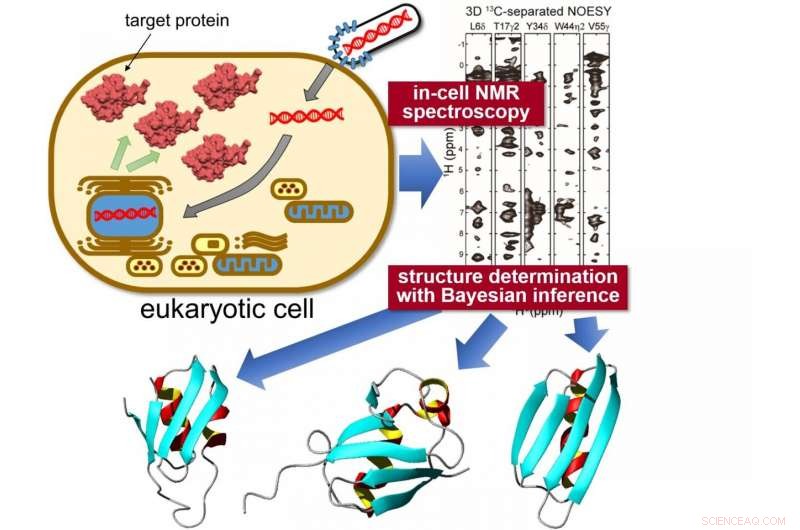
Målproteiner udtrykkes indeni sf9 celler, derefter målt under anvendelse af "in-cell" NMR-spektroskopi. Unik statistisk analyse ved hjælp af Bayesiansk slutning anvendes til at beregne den nøjagtige 3D-struktur af proteinerne med hidtil uset præcision. Kredit:Tokyo Metropolitan University
Forskere fra Tokyo Metropolitan University har med succes bestemt den høje opløsning, tredimensionel struktur af proteiner inde i levende eukaryote celler. De kombinerede "in-cell" kernemagnetisk resonans (NMR) spektroskopi, et bioreaktorsystem og banebrydende beregningsalgoritmer til at bestemme proteinstrukturer i overfyldte intracellulære miljøer for første gang. Teknikken lover indsigt i den intracellulære adfærd af sygdomsfremkaldende proteiner og nye lægemiddelscreeningsapplikationer, tillader in-situ visualisering af, hvordan proteiner reagerer på biokemiske stimuli.
Eukaryote celler er byggestenene i en lang række organismer, inklusive alle svampe, planter og dyr. Deres indre struktur er ekstremt kompleks og varieret, med et indviklet strukturelt hierarki og en bred vifte af biomakromolekyler fordelt rundt om et cytoskeletnetværk. Dette har gjort det svært at se, hvad hvert protein inde i cellerne gør i dets naturlige miljø, på trods af de åbenlyse biomedicinske fordele ved at vide, f.eks. hvordan et bestemt protein reagerer, når celler udsættes for kemiske stimuli, ligesom farmaceutiske lægemidler.
For at tackle denne udfordring, et hold fra Tokyo Metropolitan University ledet af adjunkt Teppei Ikeya og professor Yutaka Ito anvendte nuklear magnetisk resonans (NMR) spektroskopimålinger på specifikke proteiner udtrykt indeni sf9 dyrkede insektceller, en cellestamme, der oprindeligt stammer fra en type møllarve, der er meget brugt til proteinproduktion. Holdets banebrydende NMR-arbejde havde allerede haft held med at belyse højopløselige proteinstrukturer inde i bakterier (ikke-eukaryoter). Problemet med blot at anvende de samme teknikker til proteiner i sf9 celler var den signifikant lavere koncentration af målproteiner og korte levetid for celler, gør det vanskeligt at indsamle højkvalitets multidimensionelle NMR-spektre til nuklear Overhauser-effektspektroskopi (NOESY), som ville give præcis information om, hvordan forskellige atomer er fordelt inde i individuelle molekyler. Dermed, de kombinerede et sparsomt prøvetagningsbaseret hurtigt NMR-måleskema med avancerede beregningsmetoder, der anvender statistiske teknikker som Bayesiansk inferens, metoder skræddersyet til at belyse proteinstrukturer effektivt baseret på en begrænset mængde strukturel information fra in-celle NMR-spektre med iboende lav følsomhed. Et bioreaktorsystem var også udstyret inde i NMR-apparatet, som holdt cellerne i en sund tilstand under målingerne.
Med disse nye data, holdet var i stand til at belyse 3-D-strukturen af tre modelproteiner med hidtil uset høj opløsning, med en præcision på 0,5 Ångstrom (0,05 nanometer) for positionen af proteinets hovedkædeatomer. I særdeleshed, de identificerede en signifikant anderledes konformation i en lokaliseret region af et af proteinerne sammenlignet med dets referencestruktur i fortyndet opløsning. Konformationsforskellen mellem proteiner "i celler" og "i reagensglas" var formodentlig forårsaget af ikke-specifikke interaktioner med andre molekyler inde i cellerne. Det er ved at blive klart, at disse interaktioner bidrager til proteinernes biologiske funktioner:evnen til at lokalisere og kvantificere strukturelle ændringer af proteiner i et intracellulært miljø forventes at have en væsentlig indflydelse på biomedicinsk forskning, gør det muligt at se, hvordan forskellige forhold f.eks. neurodegenerative sygdomme påvirker proteinkonformationer in situ, og kvantitativt måle, hvordan behandlinger påvirker strukturelle anomalier.
 Varme artikler
Varme artikler
-
 En ny guldstandardforbindelse til generering af elektricitet fra varmeKredit:Pixabay/CC0 Public Domain Termoelektriske kraftgeneratorer, der laver elektrisk strøm fra spildvarme, ville være et nyttigt værktøj til at reducere drivhusgasemissioner, hvis det ikke var f
En ny guldstandardforbindelse til generering af elektricitet fra varmeKredit:Pixabay/CC0 Public Domain Termoelektriske kraftgeneratorer, der laver elektrisk strøm fra spildvarme, ville være et nyttigt værktøj til at reducere drivhusgasemissioner, hvis det ikke var f -
 Frem eller tilbage? Nye veje for protoner i vand eller methanolKredit:FV Berlin En kollaborativ ultrahurtig spektroskopi og ab initio molekylær dynamik simuleringsundersøgelse viser, at ledige protoner i form af hydroxid/methoxidioner er lige så relevante for
Frem eller tilbage? Nye veje for protoner i vand eller methanolKredit:FV Berlin En kollaborativ ultrahurtig spektroskopi og ab initio molekylær dynamik simuleringsundersøgelse viser, at ledige protoner i form af hydroxid/methoxidioner er lige så relevante for -
 Biologisk inspireret, højtydende polyurethan udviklet til strækbar elektronikKredit:CC0 Public Domain En forskergruppe ledet af prof. Zhu Jin ved Ningbo Institute of Materials Technology and Engineering (NIMTE) under det kinesiske videnskabsakademi (CAS) udviklede en polyu
Biologisk inspireret, højtydende polyurethan udviklet til strækbar elektronikKredit:CC0 Public Domain En forskergruppe ledet af prof. Zhu Jin ved Ningbo Institute of Materials Technology and Engineering (NIMTE) under det kinesiske videnskabsakademi (CAS) udviklede en polyu -
 En kemisk omfavnelse fra den perfekte værtGengwu Zhang udfører adskillelseseksperimentet ved hjælp af to ekstraktionstårne. Kredit:© 2020 KAUST En industriel proces, der i øjeblikket bruger store mængder energi i petrokemiske anlæg rundt
En kemisk omfavnelse fra den perfekte værtGengwu Zhang udfører adskillelseseksperimentet ved hjælp af to ekstraktionstårne. Kredit:© 2020 KAUST En industriel proces, der i øjeblikket bruger store mængder energi i petrokemiske anlæg rundt
- Forskere tager et skridt i retning af kvantemekanisk analyse af plantemetabolisme
- Eliminering af små ustabilitet i tokamaks, før de bliver til forstyrrelser
- Forskere sorterer gennem tolagsgrafen
- Sådan måles kulstofniveauer
- Nanopartikler bruges til at øge transformerolies termiske egenskaber
- Hvad gør mennesker til mennesker?


