Højhastigheds atomkraftmikroskopi tager på iboende uordnede proteiner
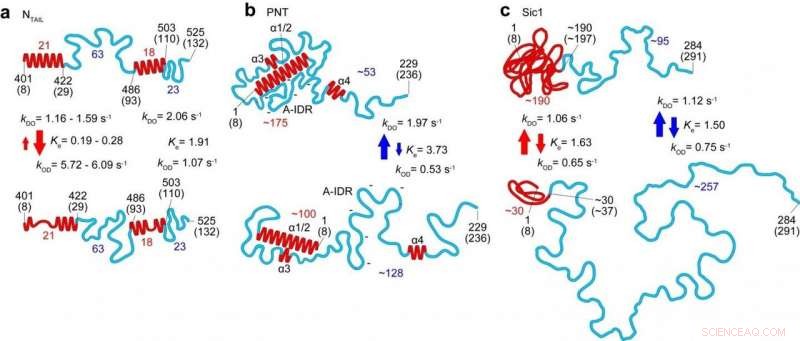
Strukturelle og dynamiske træk ved tre IDP'er (N HALE , PNT og Sic1) afsløret ved HS-AFM-billeddannelse. De øverste og nederste paneler svarer til de mere ordnede og mindre ordnede tilstande, henholdsvis. Tallene i rødt og blåt repræsenterer antallet af aminosyrer indeholdt i de respektive foldede områder og fuldstændigt uordnede områder, henholdsvis. De røde og blå pile angiver den kinetiske karakter af ændringer i højden af foldede områder og ende-til-ende afstand af fuldstændigt uordnede områder, henholdsvis. Kredit:Kanazawa University
Vores forståelse af biologiske proteiner hænger ikke altid sammen med, hvor almindelige eller vigtige de er. halvdelen af alle proteiner, molekyler, der spiller en integreret rolle i celleprocesser, er iboende uorden, hvilket betyder, at mange af standardteknikkerne til at sondere biomolekyler ikke virker på dem. Nu har forskere ved Kanazawa University i Japan vist, at deres hjemmedyrkede højhastigheds atomkraftmikroskopiteknologi kan give information ikke kun om strukturerne af disse proteiner, men også deres dynamik.
At forstå, hvordan et protein er sat sammen, giver værdifulde ledetråde til dets funktioner. Udviklingen af proteinkrystallografi i 1930'erne og 1950'erne bragte adskillige proteinstrukturer til syne for første gang, men det blev efterhånden klart, at en stor del af proteiner mangler en enkelt sæt struktur, hvilket gør dem vanskelige at behandle med røntgenkrystallografi. Da de er for tynde til elektronmikroskopi, de eneste levedygtige alternativer for mange af disse iboende uordnede proteiner (IDP'er) er nuklear magnetisk resonansbilleddannelse og lille vinkel røntgenspredning. Data indsamlet fra disse teknikker er gennemsnittet over ensembler og giver derfor ingen klar indikation af individuelle proteinkonformationer eller hvor ofte de forekommer. Atomkraftmikroskopi er på den anden side i stand til biologisk billeddannelse i nanoskala opløsning ved høj hastighed, så det kan fange dynamik såvel som proteinstrukturer.
I dette seneste arbejde forskere ved Kanazawa University sammen med samarbejdspartnere i Japan, Frankrig og Italien anvendte teknikken til at studere adskillige IDP'er og identificerede parametre, der definerer formen, størrelse og kædelængde af proteinregioner, samt en magtlov, der relaterer proteinstørrelsen til proteinlængden, og en kvantitativ beskrivelse af virkningen af glimmeroverfladen på proteindimensioner. Dynamikken i proteinkonformationerne, der er fanget takket være teknikkens højhastighedsegenskaber, afslørede kugler, der dukker op og forsvinder, og transformationer mellem fuldt ustrukturerede og løst foldede konformationer i segmenter op til 160 aminosyrer lange.
Undersøgelser af mæslingevirusnukleoproteinet hjalp især med at identificere ikke blot formen og dimensionerne, men også karakteristika ved ordens-forstyrrelser-overgangene i regionen, der er ansvarlig for molekylær genkendelse, som gør det muligt for vira at identificere værtsfaktorer, så de kan formere sig. De kunne også bestemme strukturer i større skala af virusets phosphoprotein, som ikke er tilgængelige for kernemagnetisk resonans (hvilket kun kan give en indikation af afstande mellem aminosyrer adskilt med mindre end 2 nm). Forskerne foreslår, at dannelsen af visse kompakte former, der observeres, kan forklare modstanden mod proteolyse - proteinnedbrydning.
I deres beretning om arbejdet, forskerne fremhæver, at såvel som et kraftfuldt værktøj i sig selv, "Når alle molekylære træk afsløret af HS-AFM kombineres med den foldede lokale struktur givet af NMR, den kombinerede information tillader en kvantitativ afgrænsning af de strukturelle og dynamiske karakterer af internt fordrevne, på en mere realistisk måde sammenlignet med billederne afbildet individuelt, som vist for PNT [mæslingevirus-phosphoprotein]."
Højhastigheds atomkraftmikroskopi
Atomkraftmikroskopi blev udviklet i 1980'erne og bragte opløsningen i atomskala opnået ved scanning tunneling mikroskopi (som vandt 1986 Nobelprisen i fysik) til ikke-ledende prøver. Det fungerer ved hjælp af en lille udhæng med en nanoskala spids for enden, som enten føles overfladen meget som en vinylpladenål eller banker på den. Whether by adjusting the tip height or the resonant frequency of the tapping, the interactions between tip and surface provide a signal that can be used to generate an image.
While AFM images brought huge benefits to biological research, these studies were able to move up a gear again when Toshio Ando and his team at Kanazawa University reported an atomic force microscope that operated at high speed. Atomic scale resolution images became movies bringing not just structures but also dynamics within grasp. Previous work on ordered proteins, which are reasonably well understood, as well as the IDP facilitates chromatin transcription (FACT) protein, has established that the technique can be used to image these biomolecules without effects from contact between tip and sample distorting the data.
Intrinsically disordered proteins
The arrival of xray crystallography gave researchers a clear view of vast numbers of biomolecule structures for the first time. But with the hundreds of thousands of biomolecule structures analyzed using protein crystallography since the technique first came into use in the 1930s and 1950s, a mounting body of evidence began to build that not all proteins have a single set structure. The observations ran counter to the prevailing paradigm of protein function determined by a fixed structure.
Over the past ten to twenty years the ubiquity of these intrinsically disorderd proteins and their importance in cell functions from signaling to the regulation of transcription and subsequent translation has become widely recognized. In the current work the researchers study IDPs including polyglutamine tract binding protein-1 (PQBP-1, involved in different processes, such as pre-mRNA splicing, transcription regulation, innate immunity and neuron development), autophagy proteins (which are invovolved in removing dysfunctional cell components) containing intrinsically disordered regions (IDRs) and the measles virus nucleoprotein.
 Varme artikler
Varme artikler
-
 Ny computermodel designer en lægemiddelleveringsstrategi til bekæmpelse af kræftForskere bekræftede, at længe, tynd, såkaldte endimensionelle partikler krydser typisk tumorernes porer bedst. Kredit:Kevin Craft Stanford -forskere har skabt en computersimulering, valideret af e
Ny computermodel designer en lægemiddelleveringsstrategi til bekæmpelse af kræftForskere bekræftede, at længe, tynd, såkaldte endimensionelle partikler krydser typisk tumorernes porer bedst. Kredit:Kevin Craft Stanford -forskere har skabt en computersimulering, valideret af e -
 Små silicapartikler:Kraftige midler, der kan udslette knoglesygdommeKredit:Lightspring, Shutterstock Nanopartikler udgør hovedsøjlen i nanomedicin:De bliver nu løbende udforsket for deres brug til målrettet medicinafgivelse eller reparation af beskadiget væv såsom
Små silicapartikler:Kraftige midler, der kan udslette knoglesygdommeKredit:Lightspring, Shutterstock Nanopartikler udgør hovedsøjlen i nanomedicin:De bliver nu løbende udforsket for deres brug til målrettet medicinafgivelse eller reparation af beskadiget væv såsom -
 Tilsætning af hydrogen til grafenHydrogenering (i rødt) af tolagsgrafen via Birch-type reaktion begynder fra kanterne. Billederne viser en grafenflage før (a), to minutter (b), og otte minutter (c), efter udsættelse for en opløsning
Tilsætning af hydrogen til grafenHydrogenering (i rødt) af tolagsgrafen via Birch-type reaktion begynder fra kanterne. Billederne viser en grafenflage før (a), to minutter (b), og otte minutter (c), efter udsættelse for en opløsning -
 Teknik bag verdens letteste grafen-urDenne visualisering viser lag af grafen, der bruges til membraner. Kredit:University of Manchester I januar 2017 blev verdens letteste mekaniske kronografur afsløret i Genève, Schweiz, fremvisning
Teknik bag verdens letteste grafen-urDenne visualisering viser lag af grafen, der bruges til membraner. Kredit:University of Manchester I januar 2017 blev verdens letteste mekaniske kronografur afsløret i Genève, Schweiz, fremvisning
- En elastisk fiber fyldt med elektroder, der skal revolutionere smart tøj
- Amerikanske astronauter foretager rumvandring for at udføre ISS -reparationer (opdatering)
- Krøllede nanotråde fanger mere lys for at drive elektroniske kredsløb i nanoskala
- 35-års datarekord kortlægger havtemperaturændringer
- Klimaændringer truer unikke permafrostlandformer
- Kabellængde vs Power Drop


