Benchmarking af beregningsmetoder for metagenomer
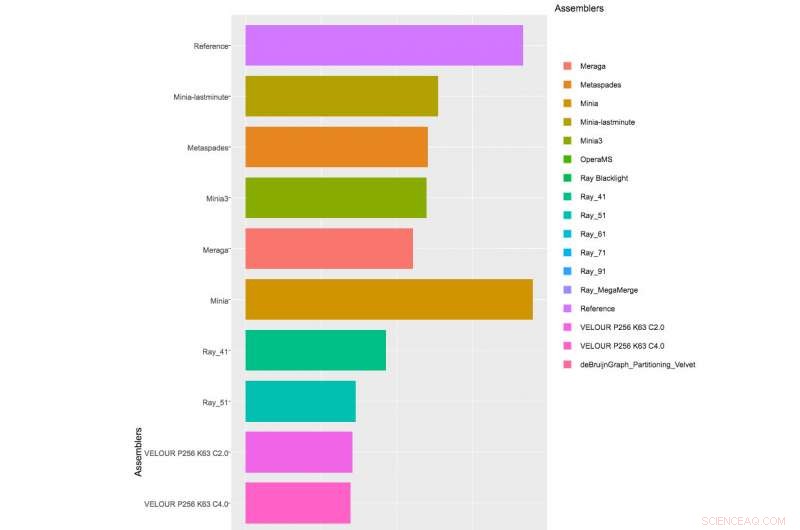
Tabel, der viser delvise resultater af samlere, der blev ansøgt til 1. CAMI Challenge, Datasæt 1. Klik her for at se hele tabellen.
De er overalt, men usynlig for det blotte øje. Mikrober er det usete, indflydelsesrige kræfter bag reguleringen af vigtige miljøprocesser såsom kulstofkredsløbet, dog forbliver de fleste af dem ukendte. I mere end et årti, US Department of Energy Joint Genome Institute (DOE JGI), en DOE Office of Science brugerfacilitet, har gjort det muligt for forskere at studere udyrkede mikrober, der ikke er i stand til at vokse i laboratoriet, ved hjælp af state-of-the-art tilgange såsom high-throughput genomisk sekventering af miljøsamfund ("metagenomics") og udvikling af beregningsværktøjer til at afdække og karakterisere mikrobielle samfund fra miljøet. At tackle samling af metagenomer til et sæt af overlappende DNA-segmenter, der tilsammen repræsenterer en konsensusregion af DNA eller contigs, derefter samle disse kontigs i genom bins, og endelig udføre taksonomisk profilering af genom bins, analytikere over hele verden har udviklet en række forskellige beregningsværktøjer, men indtil nu har der været mangel på konsensus om, hvordan man skal evaluere deres præstationer.
Udgivet 2. oktober, 2017 i Naturens metoder , et hold inklusive DOE JGI-forskere beskrev resultaterne af Critical Assessment of Metagenome Interpretation (CAMI) Challenge, den første nogensinde, samfundsorganiseret benchmarking vurdering af beregningsværktøjer til metagenomer. CAMI Challenge blev ledet af Alexander Sczyrba, leder af Computational Metagenomics-gruppen ved Bielefeld University og tidligere en DOE JGI postdoc-stipendiat, og Alice McHardy, leder af Computational Biology of Infection Research Lab ved Helmholtz Center for Infection Research.
"Det er meget svært for forskere at finde ud af, hvilket program de skal bruge til et bestemt datasæt og analyse baseret på resultaterne fra metodepapirer, " sagde McHardy. "Datasættene og evalueringsforanstaltningerne, der bruges i evalueringer, varierer meget. Et andet problem er, at udviklere normalt bruger meget tid på at benchmarke det nyeste, når de vurderer ydeevnen af ny software på den måde. CAMI ønsker at ændre disse ting og involverer samfundet i at definere standarder og bedste praksis for evaluering og anvende disse principper i benchmarking af udfordringer."
CAMI Challenge fandt sted over tre måneder i 2015. For at vurdere beregningsværktøjerne, arrangørerne udviklede 3 simulerede metagenomdatasæt ved hjælp af mere end 300 udkast til genomer af bakterielle og arkæale isolater sekventeret og samlet af DOE JGI, som var en del af Genomic Encyclopedia of Bacteria and Archaeal-projektet udgivet for nylig i Natur bioteknologi . Disse genomer blev delt med CAMI Challenges-konsortiet, før de blev frigivet til offentligheden for at lette objektiv benchmarking af forskellige værktøjer. Datasættene inkluderede også omkring det samme antal genomer fra Max Planck Institute i Köln, Tyskland, sammen med cirkulære elementer og vira. De simulerede datasæt var et enkelt prøvedatasæt på 15 milliarder baser (Gb), et 40 Gb datasæt med 40 genomer og 20 cirkulære elementer, og et 75 Gb tidsseriedatasæt bestående af fem prøver og inklusive hundredvis af genomer og cirkulære elementer.
"JGI har en stærk interesse i benchmarking af værktøjer og teknologier, der vil fremme analysen af metagenomer og forbedre kvaliteten af de data, vi leverer til brugerne. Efter at have offentliggjort den allerførste undersøgelse om brugen af simulerede datasæt til benchmarking af metagenomiske værktøjer fra JGI, det er fantastisk at se, hvordan denne metode er blevet udvidet gennem årene og nu gennem denne undersøgelse, udvikler sig til en model for standardiseret samfundsindsats på området, " sagde Nikos Kyrpides, DOE JGI Prokaryote Super Program leder.
"JGI er meget optaget af ikke kun benchmarking af laboratorieprotokoller, men også beregningsmæssige arbejdsgange, " tilføjede DOE JGI Microbial Genomics leder Tanja Woyke. "Dette gør vores deltagelse i kritiske samfundsindsatser såsom CAMI så vigtig."
Med mere end 40 hold tilmeldt udfordringen, og CAMI-arrangørerne modtog 215 bidrag fra 25 programmer rundt om i verden, selvom kun 17 teams var villige til at få deres softwareimplementeringer offentliggjort. CAMI-arrangørerne evaluerede beregningsværktøjer i 3 kategorier. Et halvt dusin assemblere og montagepipelines blev evalueret på samling af genomsekvenser genereret fra kortlæste sekventeringsteknologier. I binning-udfordringen, five genome binners and 4 taxonomic binners were evaluated on criteria including the tools' efficacy in recovering individual genomes. Endelig, 10 taxonomic profilers with various parameter settings were evaluated on how well they could predict the identities and relative abundances of the microbes and circular elements. The benchmarking results are available on https://data.cami-challenge.org/results.
The CAMI organizers are already planning future benchmarking challenges, for example to evaluate and aid method development for long read sequencing technologies. "CAMI is an ongoing initiative, " noted Sczyrba. "We are currently further automating the benchmarking and comparative result visualizations. And we invite everyone interested to join and work with CAMI on providing comprehensive performance overviews of the computational metagenomics toolkit, to inform developers about current challenges in computational metagenomics and applied scientists of the most suitable software for their research questions."
 Varme artikler
Varme artikler
-
 Los Angeles Zoos gamle indiske næsehorn aflivetDette udaterede billede leveret af Los Angeles Zoo viser Randa, et 48-årigt indisk næsehorn. Randa, som havde overlevet hudkræft, blev aflivet mandag, 6. nov., 2017, på grund af tegn på faldende helbr
Los Angeles Zoos gamle indiske næsehorn aflivetDette udaterede billede leveret af Los Angeles Zoo viser Randa, et 48-årigt indisk næsehorn. Randa, som havde overlevet hudkræft, blev aflivet mandag, 6. nov., 2017, på grund af tegn på faldende helbr -
 Illinois sportsfisk genopretning et resultat af 1972 Clean Water Act, videnskabsmænd rapportererForskere fra Illinois Natural History Survey har undersøgt fisk i Illinois-floden siden 1957. Her, holdet bruger elektricitet til at bedøve fiskene til fangst. Kredit:Aaron Yetter Populationer af
Illinois sportsfisk genopretning et resultat af 1972 Clean Water Act, videnskabsmænd rapportererForskere fra Illinois Natural History Survey har undersøgt fisk i Illinois-floden siden 1957. Her, holdet bruger elektricitet til at bedøve fiskene til fangst. Kredit:Aaron Yetter Populationer af -
 Nye bioressourcer til plantepeptidhormoner ved hjælp af genredigeringsteknologi1. Konstruktion af genmålrettingsvektorer 2. Plantetransformation 3. Første generations planteudvælgelse 4. Egenafbrydelseseffektivitetsvurdering 5. Begyndelse af genomredigeringshændelser 6. Udvælgel
Nye bioressourcer til plantepeptidhormoner ved hjælp af genredigeringsteknologi1. Konstruktion af genmålrettingsvektorer 2. Plantetransformation 3. Første generations planteudvælgelse 4. Egenafbrydelseseffektivitetsvurdering 5. Begyndelse af genomredigeringshændelser 6. Udvælgel -
 Tænk på din første hukommelse - hvorfor kan du ikke huske det endnu tidligere?BrainStuff:Hvorfor kan folk ikke huske at være født? HowStuffWorks Odds er, du husker mange forskellige ting. Din 18 års fødselsdag. Gå på gymnasiet den første dag. Sidste gang du spiste en burrito.
Tænk på din første hukommelse - hvorfor kan du ikke huske det endnu tidligere?BrainStuff:Hvorfor kan folk ikke huske at være født? HowStuffWorks Odds er, du husker mange forskellige ting. Din 18 års fødselsdag. Gå på gymnasiet den første dag. Sidste gang du spiste en burrito.
- Jorden sank to gange, oversvømmer den østlige Amazonas:Holdet finder hajtand i det nordvestlige Am…
- Superblocks -modellen kunne forhindre næsten 700 for tidlige dødsfald hvert år i Barcelona
- Indstilling af en TRAP for pandemi-fremkaldende vira
- Vandmasser i den løvfældende skov
- Fjernelse af menneskelig bias fra prædiktiv modellering
- 10 innovative arkitekter at se på


