AI-baseret screeningsmetode kunne øge hastigheden af opdagelse af nye lægemidler
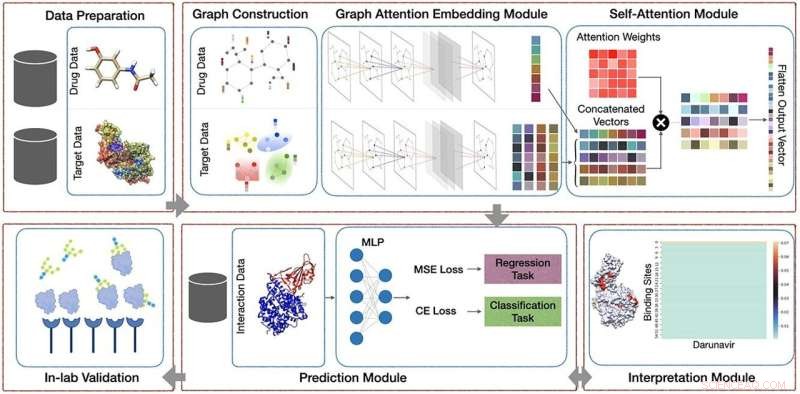
Vores foreslåede ramme omfatter fem hovedmoduler:(1) Forbehandlingsmodul, der består af at finde proteiners bindingssteder; (2) AttentionSiteDTI dybt læringsmodul, hvor vi konstruerer grafiske repræsentationer af liganders SMILE og proteiners bindingssteder, og vi skaber et grafkonvolutionelt neuralt netværk bevæbnet med en opmærksomhedspuljemekanisme til at udtrække indlæselige indlejringer fra grafer, såvel som en selv- opmærksomhedsmekanisme til at lære forholdet mellem ligander og proteiners bindingssteder; (3) Forudsigelsesmodul til at forudsige ukendt interaktion i et lægemiddel-mål-par, som kan adressere både klassificerings- og regressionsopgaver; (4) Tolkningsmodul for at give en dybere forståelse af, hvilke bindingssteder af et målprotein, der er mere sandsynligt at binde med en given ligand. (5) In-lab valideringer, hvor vi sammenligner vores beregningsmæssigt forudsagte resultater med eksperimentelt observerede (målte) lægemiddel-mål interaktioner i laboratoriet for at teste og validere det praktiske potentiale af vores foreslåede model. Kredit:Briefings in Bioinformatics (2022). DOI:10.1093/bib/bbac272
At udvikle livreddende medicin kan tage milliarder af dollars og årtier af tid, men University of Central Florida-forskere sigter mod at fremskynde denne proces med en ny kunstig intelligens-baseret lægemiddelscreeningsproces, de har udviklet.
Ved at bruge en metode, der modellerer lægemiddel- og målproteininteraktioner ved hjælp af naturlige sprogbehandlingsteknikker, opnåede forskerne op til 97% nøjagtighed i at identificere lovende lægemiddelkandidater. Resultaterne blev offentliggjort for nylig i tidsskriftet Briefings in Bioinformatics .
Teknikken repræsenterer lægemiddel-protein-interaktioner gennem ord for hvert proteinbindingssted og bruger dyb læring til at udtrække de funktioner, der styrer de komplekse interaktioner mellem de to.
"Når AI er blevet mere tilgængeligt, er dette blevet noget, som AI kan tackle," siger studiemedforfatter Ozlem Garibay, en assisterende professor i UCF's Institut for Industriel Teknik og Management Systemer. "Du kan prøve så mange variationer af proteiner og lægemiddelinteraktioner og finde ud af, hvilke der er mere tilbøjelige til at binde eller ej."
Den model, de har udviklet, kendt som AttentionSiteDTI, er den første, der kan fortolkes ved hjælp af sproget for proteinbindingssteder.
Arbejdet er vigtigt, fordi det vil hjælpe lægemiddeldesignere med at identificere kritiske proteinbindingssteder sammen med deres funktionelle egenskaber, hvilket er nøglen til at afgøre, om et lægemiddel vil være effektivt.
Forskerne opnåede denne præstation ved at udtænke en selvopmærksomhedsmekanisme, der får modellen til at lære, hvilke dele af proteinet, der interagerer med lægemiddelforbindelserne, samtidig med at den opnår avanceret forudsigelsesydelse.
Mekanismens selvopmærksomhedsevne virker ved selektivt at fokusere på de mest relevante dele af proteinet.
Forskerne validerede deres model ved hjælp af laboratorieeksperimenter, der målte bindingsinteraktioner mellem forbindelser og proteiner og sammenlignede derefter resultaterne med dem, som deres model forudsagde beregningsmæssigt. Da lægemidler til behandling af COVID stadig er af interesse, omfattede eksperimenterne også test og validering af lægemiddelforbindelser, der ville binde til et spidsprotein fra SARS-CoV2-virussen.
Garibay siger, at den høje overensstemmelse mellem laboratorieresultaterne og de beregningsmæssige forudsigelser illustrerer potentialet i AttentionSiteDTI til at forhåndsscreene potentielt effektive lægemiddelforbindelser og fremskynde udforskningen af nye lægemidler og genanvendelse af eksisterende lægemidler.
"Denne forskning med høj effekt var kun mulig på grund af tværfagligt samarbejde mellem materialeteknik og AI/ML og computerforskere for at adressere COVID-relateret opdagelse," siger Sudipta Seal, studiemedforfatter og formand for UCF's Institut for Materialevidenskab og Engineering.
Mehdi Yazdani-Jahromi, en ph.d.-studerende ved UCF's College of Engineering and Computer Science og undersøgelsens hovedforfatter, siger, at arbejdet introducerer en ny retning inden for præ-screening af lægemidler.
"Dette gør det muligt for forskere at bruge kunstig intelligens til at identificere lægemidler mere præcist for at reagere hurtigt på nye sygdomme," siger Yazdani-Jahromi. "Denne metode giver også forskerne mulighed for at identificere det bedste bindingssted for et viruss protein at fokusere på i lægemiddeldesign."
"Det næste trin i vores forskning vil være at designe nye lægemidler ved hjælp af AI's kraft," siger han. "Dette kan naturligvis være det næste skridt for at være forberedt på en pandemi." + Udforsk yderligere
Forskere identificerer nye lægemidler ved hjælp af fortolkelige forudsigelser om dyb læring
 Varme artikler
Varme artikler
-
 Hvorfor er solen så lys?Solen, en gennemsnitlig middelaldrende stjerne, giver jorden lys, varme og energi, selvom den er 150 millioner kilometer langt væk. Hvad der gør solen så lys er dens strømkilde: en proces kaldet nukle
Hvorfor er solen så lys?Solen, en gennemsnitlig middelaldrende stjerne, giver jorden lys, varme og energi, selvom den er 150 millioner kilometer langt væk. Hvad der gør solen så lys er dens strømkilde: en proces kaldet nukle -
 Bådtrafik truer Panamas Bocas Del Toro-delfiners overlevelseUbæredygtig turismepraksis truer delfinpopulationer i Panamas Bocas del Toro-provins. Kredit:Sean Mattson, STRI Flaskedelfiner i Panamas Bocas Del Toro-øhav bør udpeges som truede, siger forfatter
Bådtrafik truer Panamas Bocas Del Toro-delfiners overlevelseUbæredygtig turismepraksis truer delfinpopulationer i Panamas Bocas del Toro-provins. Kredit:Sean Mattson, STRI Flaskedelfiner i Panamas Bocas Del Toro-øhav bør udpeges som truede, siger forfatter -
 Hvordan en molekylær switch former planteorganvækstDAR2-spaltning af TMK1-EGFP flytter den fra plasmamembranen til kernen af Arabidopsis rodprotoplaster. (A –H ) Konfokale billeder af dar2-1 rodprotoplaster transformeret med TMK1-EGFP, histon HT2B-m
Hvordan en molekylær switch former planteorganvækstDAR2-spaltning af TMK1-EGFP flytter den fra plasmamembranen til kernen af Arabidopsis rodprotoplaster. (A –H ) Konfokale billeder af dar2-1 rodprotoplaster transformeret med TMK1-EGFP, histon HT2B-m -
 Bezoars magiske medicinBezoarsten findes i maven og tarmen hos dyr og mennesker. Stenen til højre er 45 mm i diameter. Den til venstre er fra en kamel. Wikimedia Commons/Wellcome -billeder Kan du huske, hvordan professor S
Bezoars magiske medicinBezoarsten findes i maven og tarmen hos dyr og mennesker. Stenen til højre er 45 mm i diameter. Den til venstre er fra en kamel. Wikimedia Commons/Wellcome -billeder Kan du huske, hvordan professor S
- Undersøgelse afslører robust ydeevne i ældet detonatorsprængstof
- Ny undersøgelse skitserer skridt videregående uddannelse bør tage for at forberede en ny kvante a…
- Optimering af lithium-svovl batterielektrolytter for lang levetid
- Grumsede søer overgår nu klare, blå søer i USA
- Simuleringer afslører, at klippefyldte superjorder med tynde atmosfærer ofte er beskyttet af en Ju…
- Hyldest af papir i naturlig størrelse hylder Suffragette-ikoner og fejrer 100 års kvinders stemmer…


