Navngivning af unavngivne bakteriearter i big data-alderen
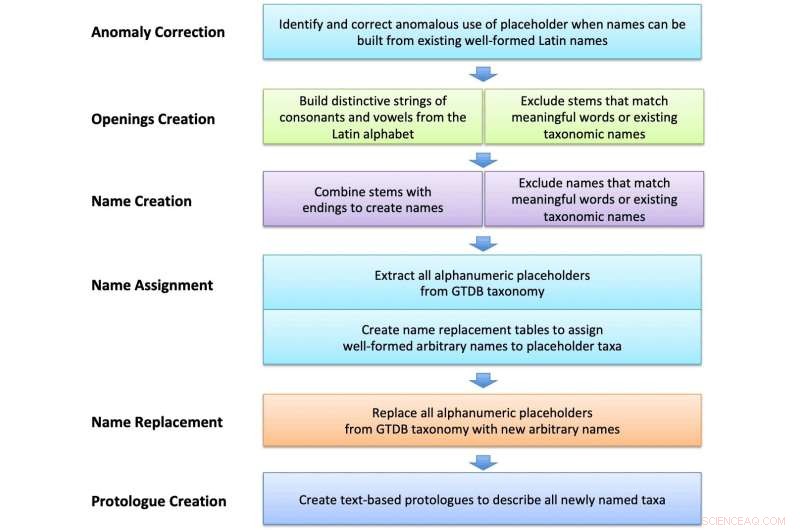
Skematisk arbejdsgang til oprettelse og tildeling af vilkårlige latinske navne. Strenge bygget af latinske bogstaver blev udvalgt og kurateret for at udelukke uønskede komponenter og meningsfulde ord. Disse blev kombineret med latinske suffikser eller ord for at skabe navne, som derefter blev brugt til at navngive pladsholdertaxa i GTDB-filer og skabe tekstbaserede protologer. Kredit:International Journal of Systematic and Evolutionary Microbiology (2022). DOI:10.1099/ijsem.0.005482
I en artikel for nylig offentliggjort i International Journal of Systematic and Evolutionary Microbiology , har forskere i Storbritannien og Østrig navngivet mere end 65.000 forskellige slags mikrober. Undersøgelsen, ledet af professor Mark Pallen ved Quadram Institute i Norwich, trækker på en lang tradition for at skabe velformede, men vilkårlige latinske navne for nye arter, men anvender denne tilgang i en skala uden fortilfælde i taksonomiens historie.
Prof. Pallen hævder, at mikrobiologi er et offer for sin egen succes, med titusindvis af nye arter opdaget i de senere år - men de fleste forbliver unavngivne. Tidligere har bakterier fået beskrivende navne eller er blevet opkaldt efter personer eller steder. Denne tilgang leverer i øjeblikket omkring tusind nye artsnavne om året. Men med et efterslæb på>50.000 velklassificerede, men unavngivne arter, med denne fremskridtshastighed, ville det tage mindst et halvt århundrede at navngive alle disse unavngivne bakterier - på hvilket tidspunkt vil videnskabsmænd stå over for problemet med at navngive de millioner flere arter opdaget i mellemtiden.
Pallen og kolleger har taget en effektiv, høj-gennemstrømnings-, big-data-tilgang, ved at bruge et computerprogram til at generere titusindvis af karakteristiske, men brugervenlige navne, der ikke ligner nogen eksisterende ord. For at overholde kravet om, at navne på bakterier skal være på latin, kombinerede holdet vilkårlige rækker af bogstaver fra det latinske alfabet med grammatisk velformede feminine suffikser. Resultatet er et sæt navne, der minder om latinens kendskab og gravitas, selvom de mangler nogen meningsfuld stamtavle. Eksempler omfatter Dupisella tifacia for en bakterie fra fåretarmen, Hopelia gocarosa for en bakterie, der lever i svensk grundvand eller Saxicetta apufaria fra en russisk saltsø.
Selvom dette kan virke radikalt, har dannelsen af navne på en vilkårlig måde en lang tradition, der strækker sig tilbage til den første kode for taksonomisk nomenklatur i 1869 og endda tilbage til taksonomiens fader, den svenske naturforsker Linnaeus. Det, der er anderledes her, er den forbløffende fornemmelse af skala, med et navnekatalog, der strækker sig over ti tusinde sider i, hvad der repræsenterer den største navngivning af arter nogensinde i en enkelt publikation.
Fordi de er blevet anvendt på arter opdaget ved DNA-sekventering, men endnu ikke dyrket i laboratoriet, forbliver de nye navne foreløbige temmelig permanente. Men da nomenklaturkoden insisterer på, at bakteriologer sigter efter stabilitet i navne, og ingen konkurrerende tilgange til navngivning i skala er på vej, forekommer det sandsynligt, at langt størstedelen af de nye navne vil blive brugt i år eller endda århundreder fremover.
Afslutningsvis siger Pallen "Jeg var medlem af den arbejdsgruppe, der gav os græske bogstaver for COVID-varianter, som hurtigt blev vedtaget af det videnskabelige samfund. Jeg håber, at de navne, der foreslås her, også hurtigt bliver vedtaget og brugt bredt. Dette er bare det første skridt. Tiden for mikrobiel opdagelse er langt fra forbi, men det vil være nemt at skabe fremtidige navne i massevis ved at bruge de principper, vi har etableret her." + Udforsk yderligere
Et automatiseret system til at generere en million nye navne til bakterier
 Varme artikler
Varme artikler
-
 Hvad forårsager Double Helix at vride i et DNA-billede?Forestil dig at du har to tynde tråde, hver ca. 3 1/4 fod lang, holdt sammen af uddrag af et vandafvisende materiale for at danne en tråd. Forestil dig nu at sætte den tråd i en vandfyldt beholder m
Hvad forårsager Double Helix at vride i et DNA-billede?Forestil dig at du har to tynde tråde, hver ca. 3 1/4 fod lang, holdt sammen af uddrag af et vandafvisende materiale for at danne en tråd. Forestil dig nu at sætte den tråd i en vandfyldt beholder m -
 Sådan fungerer virtuel medicinVirtuel medicin blev udviklet for at hjælpe de millioner af mennesker, der lider af fobier. Se mere moderne medicinbilleder. Foto med tilladelse til Virtually Better, Inc. Millioner af mennesker lide
Sådan fungerer virtuel medicinVirtuel medicin blev udviklet for at hjælpe de millioner af mennesker, der lider af fobier. Se mere moderne medicinbilleder. Foto med tilladelse til Virtually Better, Inc. Millioner af mennesker lide -
 Prag zoo føder sundhed for sjældne malaysiske tigerungerTo seks uger gamle malaysiske tigerunger blev født den 3. oktober, 2017 i zoologisk have i Prag Zoo i Prag fejrede tirsdag sundheden for to sjældne malaysiske tigerunger - en han og en hun - født
Prag zoo føder sundhed for sjældne malaysiske tigerungerTo seks uger gamle malaysiske tigerunger blev født den 3. oktober, 2017 i zoologisk have i Prag Zoo i Prag fejrede tirsdag sundheden for to sjældne malaysiske tigerunger - en han og en hun - født -
 Smart boksning:den enkle bevaringsstrategi, der redder truede RoseateternerLivet kan være hårdt for en Rockabill-vagt. Kredit:Brian Burke En simpel bevaringsstrategi, der er implementeret af naturbevarere og videnskabsmænd fra BirdWatch Ireland, Trinity College Dublin og
Smart boksning:den enkle bevaringsstrategi, der redder truede RoseateternerLivet kan være hårdt for en Rockabill-vagt. Kredit:Brian Burke En simpel bevaringsstrategi, der er implementeret af naturbevarere og videnskabsmænd fra BirdWatch Ireland, Trinity College Dublin og
- NASA ser hjertet på Tropical Cyclone Dineo på Valentinsdag
- Forskere bruger forstenede tænder til at afsløre kostændringer hos gamle planteædere og hominine…
- Modellering af uligevægtsforbindelser i nanoskala med steady-state densitet funktionel teori
- Den næstkoldeste afbildede exoplanet fundet til dato
- Røg ved brande kan reducere regndråber til meningsløs støvregn, undersøgelse siger. Her er hvor…
- Sådan beregnes Win-Loss-Tie Procentager


