Forskere afslører nyt system til navngivning af størstedelen af verdens mikroorganismer
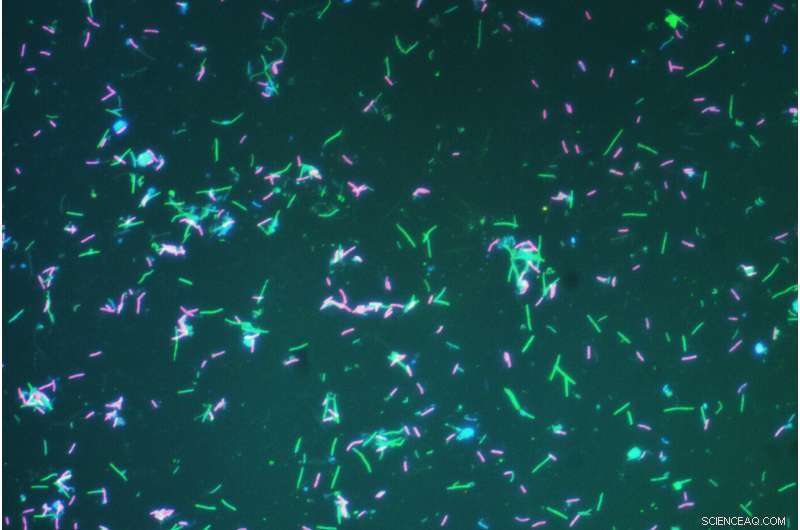
Fluorescerende-farvede bakterier (lyserøde) og archaea (grønne) fra næsten-kogende vand fra Great Boiling Spring i Gerlach, Nevada. Kredit:Jeremy Dodsworth
Hvad er i et navn? For mikroorganismer, tilsyneladende meget.
Prokaryoter er encellede mikroorganismer - bakterier er et eksempel - der er rigelige over hele verden. De findes i havene, i jordbunden, i ekstreme miljøer som varme kilder og endda ved siden af og inde i andre organismer, herunder mennesker.
Kort sagt, de er overalt, og videnskabsmænd verden over arbejder på både at kategorisere og kommunikere om dem. Men her er gnisten:De fleste har ikke et navn.
Mindre end 0,2% af kendte prokaryoter er blevet formelt navngivet, fordi de nuværende regler - beskrevet i den internationale kode for nomenklatur for prokaryoter (ICNP) - kræver, at nye arter dyrkes i et laboratorium og frit distribueres som rene og levedygtige kulturer i samlinger. I det væsentlige, for at nævne det, skal du have flere fysiske prøver for at bevise det.
I en artikel offentliggjort 19. september i tidsskriftet Nature Microbiology , præsenterer et team af videnskabsmænd et nyt system, SeqCode, og en tilsvarende registreringsportal, der kan hjælpe mikrobiologer effektivt at kategorisere og kommunikere om det enorme antal identificerede, men ukultiverede prokaryoter.
"Vores mål er at forene felt- og laboratorieundersøgelser i mikrobiologi og reagere på betydelige nylige fremskridt inden for miljøgenomik ved at give en vej til formelt at navngive størstedelen af identificerede, men unavngivne prokaryoter," sagde UNLV mikrobiolog Brian Hedlund, hovedforfatter på papiret og nøglen. samarbejdspartner om udviklingen af SeqCoden. "SeqCode bør tjene samfundet ved at fremme høje genomkvalitetsstandarder, god navngivningspraksis og en velordnet database."
Oprettelse af SeqCode
Næsten 850 videnskabsmænd, der repræsenterer flere discipliner fra mere end 40 lande, deltog i en række NSF-finansierede online workshops i 2021 for at udvikle den nye SeqCode, som bruger genomsekvensdata for både dyrkede og udyrkede prokaryoter som grundlag for at navngive dem.
Siden 2000'erne har videnskabsmænd, der studerer prokaryoter i miljøer over hele verden, brugt miljøgenomiske teknikker til at prøve og studere dem, og hundredtusindvis af genomsekvenser er tilgængelige i offentlige databaser. Samfundet, der deltog i workshopperne, som var organiseret af Hedlund og kollega Anna-Louise Reysenbach fra Portland State University, støttede overvældende udviklingen af et alternativ til ICNP, der ville acceptere DNA-sekvensdata og i sidste ende forbedre ressourcerne for forskere.
"Nøglestykkerne er på plads for en velordnet udvidelse af prokaryot systematik til hele livets prokaryote træ," sagde William B. Whitman, SeqCode-korresponderende forfatter og mikrobiolog ved University of Georgia. "Denne udvidelse vil tjene forskningen og det bredere samfund ved at give et fælles sprog for alle prokaryoter, der er systematisk organiseret og understøttet af datarige genomiske datasæt og tilhørende metadata."
For at kvalificere sig til inklusion i SeqCode skal genomer opfylde strenge videnskabelige standarder for at sikre kvalitet, stabilitet og åben datadeling. Og selvom den endnu ikke er universelt accepteret, er SeqCode grundlæggende på linje med etablerede internationale principper for navngivning af andre organismer, herunder planter og dyr.
"Enhver organisme med en genomsekvens af høj kvalitet - fra en ren kultur eller ej - kan navngives under SeqCode," sagde Hedlund. "Vi vil også automatisk acceptere alle navne dannet under ICNP. Jeg forventer med tiden, at SeqCode vil blive brugt meget hyppigere end ICNP."
Skaber klarhed blandt kaos
Et af de primære mål for det nye system, hævder forfattere, er at vende en tendens på området, hvor "uregulerede" navne bruges i litteraturen af nød. Dette kan føre til fejl, der øger sandsynligheden for efterfølgende omdøbning senere, hvilket gør det vanskeligt for forskere at gennemgå og sammenligne data og kommunikere effektivt. Omvendt hævder forfattere, at SeqCode "omfavner principperne om findebarhed, tilgængelighed, interoperabilitet og genbrugelighed."
Hedlund nævnte klamydia og beslægtede organismer som et eksempel. Da disse organismer ikke kan dyrkes, opbevares eller distribueres som rene kulturer, er de i øjeblikket ikke i stand til at blive officielt navngivet.
"Det kunne være ret forvirrende for klinikere ikke at have gyldige navne på nyopdagede klamydiaer," siger Hedlund. "Der er en risiko for, at disse navne bliver dårligt katalogiseret, hvilket kan kvæle sporing af sygdomsudbrud og kommunikation mellem forskere, læger og offentligheden."
Overvindelse af kontrovers
På trods af dets tilsigtede mål om at skabe klarhed og synergi med accepterede standarder for navngivning, er flytningen ikke uden kontroverser.
SeqCode følger et tidligere forsøg fra videnskabsmænd på at modificere ICNP for at tillade ukultiverede prokaryoter at blive navngivet baseret på at have en DNA-sekvens, der ville tjene som bevis (eller 'type') for organismen - i modsætning til ICNP-reglerne nu, som kræver en kultur i to permanente samlinger.
I 2020 offentliggjorde et hold ledet af Desert Research Institute-biologen Alison Murray et papir, også i Nature Microbiology , der var medforfatter eller godkendt af næsten 120 videnskabsmænd, der repræsenterer 22 lande, der opfordrer til handling på de foreslåede ændringer af ICNP for at acceptere DNA-sekvenser som typer eller for at gå en alternativ vej. Imidlertid blev de foreslåede ændringer afvist af Den Internationale Komité for Systematik af Prokaryoter, den gruppe, der er ansvarlig for at styre navngivningen af prokaryoter.
"Det er klart, at det globale samfund af videnskabsmænd er klar til en paradigmeændring i, hvordan vi navngiver prokaryoter - for at inkludere bredden af prokaryot liv," sagde Murray. "Moderne genomteknologier kan opløse genomer fra udyrkede organismer med den høje grad af præcision, der er nødvendig for at sikre integritet og give stabilitet til mikrobiologiområdet. Navngivning af disse taxa er måden at kommunikere deres eksistens, deres evolutionære historie og forudsige deres fysiologiske evner."
Tilbageslaget i 2020 førte til en fordobling af indsatsen blandt den voksende kadre af videnskabsmænd og i sidste ende den "alternative rute", som førte til dannelsen af SeqCode.
"Mange mennesker kom til bordet for at dele deres perspektiver, deres energi og deres evner til at få det til at ske," sagde Hedlund. "Reaktionen på vores workshops fra videnskabsmænd over hele verden var utrolig og hjalp med at validere, hvorfor tiden er inde til formelt at lave en ændring i, hvordan prokaryoter navngives."
Spændinger eksisterer stadig blandt nogle videnskabsmænd, som hævder, at man kan vide mindre om ukultiverede prokaryoter end dem, der kan dyrkes og manipuleres i et laboratorium som rene kulturer. Derudover kan nuancer i bearbejdning og fortolkning af DNA-sekvensdata potentielt føre til fejlagtige konklusioner, et punkt, som Hedlund hævder, også gælder for studier af rene kulturer.
Forfatterne siger, at dette nye system ikke er beregnet til at modvirke traditionel dyrkning af prokaryoter, men i stedet er designet af det videnskabelige samfund til at forbedre kommunikationen på tværs af de mikrobielle videnskaber.
"Vi betragter denne 'SeqCode v.1.0' som et nødvendigt første skridt mod et samlet system af nomenklatur til at kommunikere den fulde mangfoldighed af prokaryoter, og vi vil samarbejde med fællesskabet for at realisere denne vision," skriver forfatterne.
Artiklen, "SeqCode:a nomenclatural code for prokaryotesscribed from sequence data" blev offentliggjort 19. september i tidsskriftet Nature Microbiology . + Udforsk yderligere
Forskere foreslår nyt navngivningssystem for udyrkede bakterier og arkæer
 Varme artikler
Varme artikler
-
 Hvordan adskiller cytokinesis sig i planter og dyr?Cytokinesis er det sidste trin i cellereplikation efter mitose, duplikering af en celle, er afsluttet. Under denne proces halveres cytoplasmaet af den originale celle sig lige for de to resulterende c
Hvordan adskiller cytokinesis sig i planter og dyr?Cytokinesis er det sidste trin i cellereplikation efter mitose, duplikering af en celle, er afsluttet. Under denne proces halveres cytoplasmaet af den originale celle sig lige for de to resulterende c -
 PITT-vej:Forskere opdager, hvordan celler reparerer et langtidsfremmende genbrugssystemFluorescensmikroskopibilleder, der viser det endoplasmatiske retikulum-netværk (grønt), der vikler sig rundt om beskadigede lysosomer (rødt). Cellekernen er vist med blåt. Kredit:Jay Xiaojun Tan I
PITT-vej:Forskere opdager, hvordan celler reparerer et langtidsfremmende genbrugssystemFluorescensmikroskopibilleder, der viser det endoplasmatiske retikulum-netværk (grønt), der vikler sig rundt om beskadigede lysosomer (rødt). Cellekernen er vist med blåt. Kredit:Jay Xiaojun Tan I -
 AmbienSovepillen Ambien får nogle mennesker til at spise i søvne uden erindring. Se flere søvnbilleder. Bryce Lankard/Getty Images En given nat, folk kravler ud af sengen og går mod deres køkkener med usto
AmbienSovepillen Ambien får nogle mennesker til at spise i søvne uden erindring. Se flere søvnbilleder. Bryce Lankard/Getty Images En given nat, folk kravler ud af sengen og går mod deres køkkener med usto -
 Fra dråbe til opdagelseAxin- og APC-proteiner danner en dråbe omkring centrosomet, hvilket skaber et reaktionskammer fra Wnt-vejen. Kredit:Matt Perko Stamceller flyder over med potentiale. Deres evne til at blive andre c
Fra dråbe til opdagelseAxin- og APC-proteiner danner en dråbe omkring centrosomet, hvilket skaber et reaktionskammer fra Wnt-vejen. Kredit:Matt Perko Stamceller flyder over med potentiale. Deres evne til at blive andre c
- Virkningerne af torden og lynet på mennesker & natur
- Rød glød hjælper med at identificere nanopartikler til levering af RNA-terapier
- Illinois slagteri største kvælstofforurener af vandveje i USA, undersøgelse finder
- Medforbrydere er nøglen til kriminalitetsmønstre
- Hvad er den sjældneste metal?
- Hvid nanolyskilde til optisk nanobilleddannelse


