Gener organiserer rumligt for effektiv mRNA-splejsning, viser undersøgelse
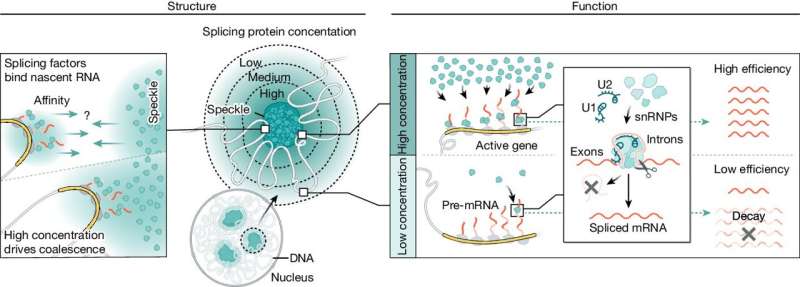
Kernen i hver af dine celler indeholder al den genetiske information (genomet), der er nødvendig for at opbygge enhver type celle og protein i hele din krop. Som et komplekst bibliotek i et lille rum, der er 50 gange mindre end bredden af et menneskehår, er gener organiseret i præcise områder i tre dimensioner.
Ud over genomet indeholder kernen strukturer kaldet kernelegemer, der indeholder høje koncentrationer af specifikke proteiner og nukleinsyrer. Nukleare organers rolle har imidlertid været et mysterium i næsten et århundrede. En ny undersøgelse fra laboratoriet af Caltechs Mitch Guttman, professor i biologi, viser, at disse nukleare kroppe kan fungere som miniaturefabrikker for at muliggøre effektiv produktion af mRNA.
mRNA-splejsning er en grundlæggende del af den proces, der opstår, når kodede DNA-instruktioner omdannes til et funktionelt protein. Efter at et gen er transskriberet fra DNA til RNA, skal uvedkommende sektioner (kaldet introner) skæres ud, en proces, der lettes ved at splejse enzymer.
I det nye studie undersøgte Caltech-forskerne en særlig type kernelegeme, kaldet kernespletter, som indeholder høje koncentrationer af splejsningsenzymer. Guttmans team opdagede, at genomet fysisk flytter sig selv, så stærkt transskriberede gener er tæt på pletter, hvilket muliggør mere effektiv splejsning.
Resultaterne har implikationer for både grundlæggende videnskabelig forståelse af forholdet mellem nuklear struktur og cellulær funktion og i sidste ende for fremtidig terapeutisk udvikling for sygdomme, hvor mRNA-splejsning er påvirket, herunder for neurodegenerative tilstande som ALS.
Papiret, der beskriver resultaterne, blev offentliggjort i tidsskriftet Nature den 8. maj. Den har titlen "Genomorganisation omkring nukleare pletter driver mRNA-splejsningseffektivitet."
I et årti har forskere i Guttman-laboratoriet studeret, hvordan kernen er rumligt organiseret - layoutet af biblioteket, med andre ord. De 3D-strukturer, som DNA er bestilt i, gør visse gener mere eller mindre tilgængelige for maskineriet, der omdanner DNA til mRNA, og det nye studie viser, at genomets fysiske struktur forbinder transkription med splejsningsprocessen.
En muskelcelle vil for eksempel flytte sit genom, så stærkt transskriberede gener for muskelaktivitet er i fysisk nærhed af nukleare pletter, hvor høje koncentrationer af splejsningsenzymer gør RNA-splejsning særlig effektiv. En neuronal celle vil på den anden side omorientere sit genom i rummet, så de gener, der er nødvendige for at producere celler, der er specialiserede til neurologisk funktion, er tættere på pletterne.
"Vi ved, at splejsning er afhængig af enzymer, og enhver mutation, der forstyrrer koncentrationen af enzymer, har dramatiske virkninger," siger Prashant Bhat, en postdoc-forsker i biologi og biologisk ingeniørvidenskab og undersøgelsens første forfatter.
"Tidligere havde vi en tendens til at tænke på splejsning som en konstant – du laver et præ-mRNA; du splejser det. Nu ved vi, at kernens fysiske organisering gør en stor forskel i effektiviteten af splejsning og i forlængelse heraf andre processer ."
Mange sygdomme, herunder neurodegenerative lidelser og kræft, har en fælles karakteristik:dysfunktionel RNA-splejsning. Selvom det ikke vides, om ukorrekt splejsning er en primær årsag til sygdom eller en effekt, er splejsningsprocessen i sidste ende et lovende mål for behandlinger.
For eksempel kan sygdomme, hvor et utilstrækkeligt antal proteiner fremstilles (kendt som haploinsufficiens-sygdomme) behandles ved at genplacere den normale kopi af genet for det protein nær nukleare pletter for at øge splejsningen og genoprette sunde proteinniveauer.
Flere oplysninger: Prashant Bhat et al., Genomorganisation omkring nukleare pletter driver mRNA-splejsningseffektivitet, Nature (2024). DOI:10.1038/s41586-024-07429-6
Journaloplysninger: Natur
Leveret af California Institute of Technology
 Varme artikler
Varme artikler
-
 Hvorfor stinker stinkplanten?Burde de mennesker ikke holde næsen? Denne sjældne blomstrende ligblomst er en af omkring et dusin eller har blomstret i USA i det sidste århundrede. Se flere ligblomstbilleder. David McNew/Newsmake
Hvorfor stinker stinkplanten?Burde de mennesker ikke holde næsen? Denne sjældne blomstrende ligblomst er en af omkring et dusin eller har blomstret i USA i det sidste århundrede. Se flere ligblomstbilleder. David McNew/Newsmake -
 Kan bedrifter producere maksimalt og stadig reducere drivhusgasemissionerne?Kredit:Unsplash/CC0 Public Domain Dekarbonisering af landbruget er afgørende for, at USA kan nå netto nul-emissioner i 2050. En ny datadrevet tilgang ser på praksis, der er god for jorden og rentab
Kan bedrifter producere maksimalt og stadig reducere drivhusgasemissionerne?Kredit:Unsplash/CC0 Public Domain Dekarbonisering af landbruget er afgørende for, at USA kan nå netto nul-emissioner i 2050. En ny datadrevet tilgang ser på praksis, der er god for jorden og rentab -
 Med ekstra sukker, blade bliver også fedeEt Brookhaven Lab forskerhold ledet af John Shanklin (stående, bag) undersøgte strategier til at øge olieproduktionen og akkumulering i planteblade. Zhiyang Zhai (siddende, holding plant) udførte de b
Med ekstra sukker, blade bliver også fedeEt Brookhaven Lab forskerhold ledet af John Shanklin (stående, bag) undersøgte strategier til at øge olieproduktionen og akkumulering i planteblade. Zhiyang Zhai (siddende, holding plant) udførte de b -
 Nabocelletyper påvirker enkeltcelle-genekspressionsvariabilitetKredit:Meletios Verras/Shutterstock Nogle gange ved du ikke, hvad du leder efter, før du finder det; og det gælder især, når det kommer til de enorme datasæt, der kan genereres ved hjælp af moderne
Nabocelletyper påvirker enkeltcelle-genekspressionsvariabilitetKredit:Meletios Verras/Shutterstock Nogle gange ved du ikke, hvad du leder efter, før du finder det; og det gælder især, når det kommer til de enorme datasæt, der kan genereres ved hjælp af moderne
- Vand er forfriskende i varmen, ikke? I dele af Florida i sidste uge, ikke så meget
- NZs fossiler viser, at flere arter levede i varmere farvande, men de nuværende opvarmningstendenser…
- Forskere opfinder en ny type mikroskop, der kan se gennem et intakt kranium
- FN advarer om stigende niveauer af giftig saltlage, efterhånden som afsaltningsanlæg opfylder voks…
- Asteroid orus okkultation observeret for første gang nogensinde af eVscope
- Huawei:Amerikanske kontroller har ingen indflydelse, taler med Google


