AlphaFold 3-opgradering muliggør forudsigelse af andre typer biomolekylære systemer
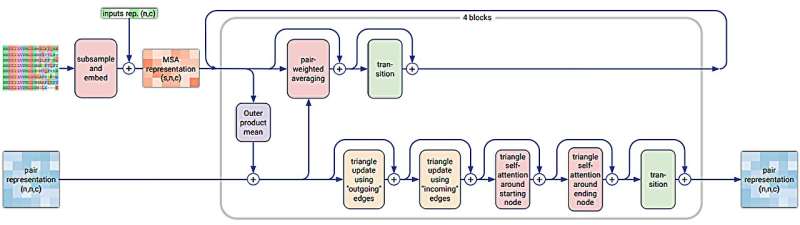
Et kombineret team af medicinske forskere og AI-systemspecialister fra Googles Deep Mind-projekt og Isomorphic Labs, begge i London, har foretaget, hvad gruppen beskriver som væsentlige forbedringer af AlphaFold 2, der gør det muligt for applikationen at forudsige strukturen af en lang række forskellige af biomolekylære systemer mere bredt og præcist. Den nye iteration kaldes AlphaFold 3.
I deres undersøgelse offentliggjort i tidsskriftet Nature , brugte gruppen diffusionsteknikker til at lave forbedringer af den underliggende arkitektoniske model af applikationen for at give mulighed for at lave mere generelle forudsigelser.
Den første version af det deep-learning-baserede AI-system AlphaFold blev udgivet for blot fire år siden og blev bebudet for sin evne til at lave præcise forudsigelser om strukturen af proteiner ved hjælp af sekvenser af aminosyrer. Det har også hjulpet forskere med bedre at forstå, hvordan proteiner virker. AlphaFold 2 byggede på sådanne egenskaber og udvidede de komplekser, der kunne forudsiges.
I denne nye iteration har forskerholdet givet applikationen evnen til at forudsige biomolekylære systemer ud over proteiner. Det kan forudsige ligander, for eksempel, eller RNA- eller DNA-strukturer. De bemærker, at det endda kan komme med forudsigelser om strukturen af ioner, nukleinsyrer, andre proteiner og interaktioner mellem antistoffer og antigener.
Disse evner, bemærker forskerne, gør det til et nyttigt værktøj til opdagelsen af nye lægemidler. Et lægemiddelopdagelsesfirma (og DeepMind-spinoff) bruger allerede det nye system til at gøre netop det.
Ud over at komme med forudsigelser om andre biomolekylære strukturer hævder forskerholdet, at AlphaFold 3 også er meget mere præcis end dets tidligere iterationer og dets konkurrenter. Men de anerkender også, at der er plads til at vokse:AlphaFold 3 har for eksempel en chiralitetsfejlrate på 4,4 %. Det hallucinerer også nogle gange, hvilket reducerer udseendet af bånd.
De bemærker, at arbejdet vil fortsætte med AlphaFold-systemet, da teamet søger at forbedre nøjagtigheden og tilføje flere typer systemer, som det kan anvendes til. De planlægger også at indføre en rangordningsstruktur for at hjælpe brugerne med at vurdere resultaterne fra systemet.
Flere oplysninger: Josh Abramson et al., Nøjagtig strukturforudsigelse af biomolekylære interaktioner med AlphaFold 3, Nature (2024). DOI:10.1038/s41586-024-07487-w
Journaloplysninger: Natur
© 2024 Science X Network
 Varme artikler
Varme artikler
-
 Ingen tryllestav påkrævet:Forskere foreslår måde at omdanne enhver celle til en hvilken som hels…Kredit:CC0 Public Domain I eventyr, alt det kræver at forvandle en frø til en prins, en tjener til en prinsesse eller en mus til en hest er bølgen af en tryllestav. Men i den virkelige verden,
Ingen tryllestav påkrævet:Forskere foreslår måde at omdanne enhver celle til en hvilken som hels…Kredit:CC0 Public Domain I eventyr, alt det kræver at forvandle en frø til en prins, en tjener til en prinsesse eller en mus til en hest er bølgen af en tryllestav. Men i den virkelige verden, -
 Dette træ er Europas ældste levende organisme - sådan,Slags, måske Adonis, en bosnisk fyr, er et af de ældste træer i Europa. Dr. Oliver Konter, Mainz Turister, der tager til Europa, gør det ofte til et punkt at besøge antikviteterne. Men hvem s
Dette træ er Europas ældste levende organisme - sådan,Slags, måske Adonis, en bosnisk fyr, er et af de ældste træer i Europa. Dr. Oliver Konter, Mainz Turister, der tager til Europa, gør det ofte til et punkt at besøge antikviteterne. Men hvem s -
 Chile kæmper for at redde en yndlingsmuslingEn fisker viser saltvandsmuslinger kendt som machas (Mesodesma Donacium) efter at have samlet dem fra kysten ved en strand i La Serena, omkring 400 km nord for Santiago, den 5. september, 2017. I augu
Chile kæmper for at redde en yndlingsmuslingEn fisker viser saltvandsmuslinger kendt som machas (Mesodesma Donacium) efter at have samlet dem fra kysten ved en strand i La Serena, omkring 400 km nord for Santiago, den 5. september, 2017. I augu -
 At redde padder fra en dødelig svamp betyder at handle uden at kende alle svareneEn boreagtig tudse venter på muligheder for at parre sig i nærheden af en bjergsø i Colorado. Kredit:Brittany Mosher, CC BY-ND Frøernes kald på varme nætter om foråret er en velkommen lyd, fortæ
At redde padder fra en dødelig svamp betyder at handle uden at kende alle svareneEn boreagtig tudse venter på muligheder for at parre sig i nærheden af en bjergsø i Colorado. Kredit:Brittany Mosher, CC BY-ND Frøernes kald på varme nætter om foråret er en velkommen lyd, fortæ
- Ny radiomærkningsmetode til personlig kræftbehandling
- Forviklingen af to kvantehukommelsessystemer 12,5 km fra hinanden
- Hvordan NASA sporede den mest intense solstorm i årtier
- Lufthansa reducerer flådestørrelsen, Luk Germanwings, når virus rammer
- Super-Earth Ross 508b skimmer den beboelige zone af rød dværg
- Pandemiens indvirkning på det brasilianske arbejdsmarked


