Nabocelletyper påvirker enkeltcelle-genekspressionsvariabilitet
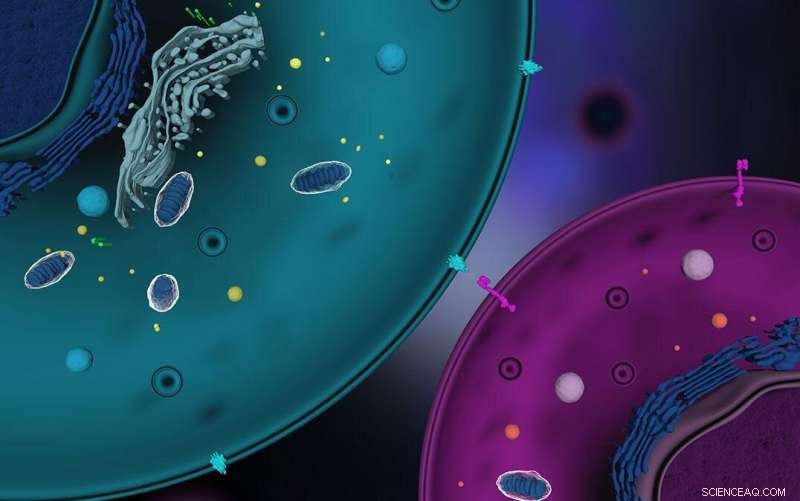
Kredit:Meletios Verras/Shutterstock
Nogle gange ved du ikke, hvad du leder efter, før du finder det; og det gælder især, når det kommer til de enorme datasæt, der kan genereres ved hjælp af moderne sekventeringsteknikker. Nu rapporterer forskere fra Japan om udviklingen af en statistisk ramme, der kan udføre upartisk udvinding af biologisk relevant celle-celle-kommunikation fra et hav af rumlige genekspressionsdata.
I en undersøgelse offentliggjort i september i Bioinformatics , har forskere fra University of Tsukuba afsløret, at en ny statistisk analysemetode nøjagtigt kan identificere celle-celle-kommunikation, der påvirker genekspression på enkeltcelleniveau.
Kommunikation mellem celler regulerer genekspression på måder, der er afgørende for normal funktion såvel som sygdomsudvikling. Mens enkeltcellet RNA-sekventering og rumligt opløst transkriptomik kan give en vis indsigt i denne kommunikation, har de nuværende metoder til at analysere disse typer data nogle vigtige begrænsninger.
"De fleste eksisterende statistiske analysemetoder tager ikke højde for den rumlige organisering af celler i et organ, der er sammensat af forskellige celletyper," siger lektor Haruka Ozaki, seniorforfatter af undersøgelsen. "Men placeringen af celler, antallet af celler og celletyperne i nærheden påvirker genekspression i naboceller."
For at fange denne kompleksitet skabte forskerne en statistisk ramme kaldet CCPLS (Cell-Cell Communications Analysis by Partial Least Square regression modeling), der analyserer rumlige genekspressionsdata ved enkeltcelle opløsning. Formålet med dette system var at identificere og kvantificere indflydelsen af nabocelletyper på celle-til-celle variabilitet i genekspression.
"Vi anvendte først CCPLS på et simuleret datasæt og fandt ud af, at det nøjagtigt estimerede virkningerne af flere nabocelletyper på genekspression," siger lektor Ozaki. "Derefter anvendte vi systemet på et datasæt fra den virkelige verden og viste, at astrocytter fremmer differentiering af oligodendrocytprecursorceller til oligodendrocytter, hvilket er i overensstemmelse med tidligere museeksperimenter."
Derefter blev CCPLS anvendt på et andet datasæt fra den virkelige verden indeholdende genekspressionsdata fra ni forskellige celletyper fundet i tyktarmen. Analysen viste, at epitelcelleudvikling af umodne B-celler sker gennem kommunikation med IgA B-celler, hvilket ikke tidligere er blevet rapporteret.
"Vores resultater viser, at CCPLS kan bruges til at udtrække biologisk relevant indsigt om celle-celle-kommunikation fra komplekse datasæt," siger professor Ozaki.
I betragtning af at CCPLS udkonkurrerede en eksisterende statistisk ramme til at identificere genekspressionsvariabilitet reguleret af celle-cellekommunikation, er det sandsynligt, at det vil være et yderst nyttigt værktøj til datasætanalyse i fremtiden. Det kan være særligt effektivt til at udforske lægemiddelmål og i tilfælde, hvor cellearrangementet forårsager ændringer i genekspression. + Udforsk yderligere
Matematiske metoder til analyse af enkeltcellede transkriptomiske data
 Varme artikler
Varme artikler
-
 Nyfundet protein kan forhindre virusinfektion og herpes-induceret kræftForskerne fandt et protein, der hjælper med at forhindre humant herpesvirus 8 i at replikere ved at detektere en specifik modifikation på viral messenger-RNA. Kredit:Brandon Tan Keck School of Med
Nyfundet protein kan forhindre virusinfektion og herpes-induceret kræftForskerne fandt et protein, der hjælper med at forhindre humant herpesvirus 8 i at replikere ved at detektere en specifik modifikation på viral messenger-RNA. Kredit:Brandon Tan Keck School of Med -
 Brandtilpassede insekter får mest muligt ud af ynglepladser steriliseret af skovbrandeNy USask-forskning viser, at disse ildelskende biller (Sericoda obsoleta, venstre) drager fordel af at lægge æg i et brændt miljø efter skovbranden (til højre, Fort à la Corne, brændt i 2020). Kredit:
Brandtilpassede insekter får mest muligt ud af ynglepladser steriliseret af skovbrandeNy USask-forskning viser, at disse ildelskende biller (Sericoda obsoleta, venstre) drager fordel af at lægge æg i et brændt miljø efter skovbranden (til højre, Fort à la Corne, brændt i 2020). Kredit: -
 Forskere sporer hajer, der plukker DNA-fragmenter fra havetKredit:University of Salford Havøkologer har vist, at spor af DNA i havet kan bruges til at overvåge hajbestande. Nuværende metoder til lokkemad, at kroge og filme hajer, rokker og andre store fi
Forskere sporer hajer, der plukker DNA-fragmenter fra havetKredit:University of Salford Havøkologer har vist, at spor af DNA i havet kan bruges til at overvåge hajbestande. Nuværende metoder til lokkemad, at kroge og filme hajer, rokker og andre store fi -
 Den rigtige måde at reparere DNA påSalk-forskere opdager, at CYREN-mikroprotein hjælper celler med at vælge den bedste vej til at reparere gener og undgå kræft. Til venstre:Kromosomer (røde) med telomerer (grønne), der er uforstyrrede,
Den rigtige måde at reparere DNA påSalk-forskere opdager, at CYREN-mikroprotein hjælper celler med at vælge den bedste vej til at reparere gener og undgå kræft. Til venstre:Kromosomer (røde) med telomerer (grønne), der er uforstyrrede,
- LCO og NASAs Kepler arbejder sammen for at bestemme oprindelsen af supernova
- Ny undersøgelse viser, at røde tidevand kan forudsiges
- Encellede arkitekter inspirerer til ny nanoteknologi
- Mars-støv som grundlag for liv? Intet problem for visse bakterier
- Forskerhold udvikler rekordlaser på chip
- Maskinlytning efter jordskælv:Algoritmer udvælger skjulte signaler, der kan øge geotermisk energi…


