Undersøgelse finder, at genomsløjfer ikke varer længe i celler:Teorier om, hvordan kontrolgenekspression muligvis skal revideres
Forskerne udviklede en beregningsmæssig pipeline, der integrerer og analyserer enkeltcelle-billeddannelse, mikroskopidata og andre genom-dækkende datasæt. De kaldte tilgangen COLA (CO-localization and Looping Analysis). COLA finder DNA-looping-hændelser i superopløsningsmikroskopibilleder og forbinder dem med aktivitetsudlæsninger målt på tværs af genomet, som RNA-polymerasebinding.
Ved hjælp af COLA undersøgte holdet looping-begivenheder ved fire genomiske loci, der er kendt for at interagere på lang afstand. Overraskende nok fandt de ud af, at individuelle sløjfer sjældent holder længere end et par minutter, og mange er meget dynamiske og gennemgår hurtig montering og adskillelse. Disse resultater udfordrer den fremherskende opfattelse, at genomiske kontakter afspejler stabile, vedvarende kromatinstrukturer, der opretholder transkriptionelle programmer. I stedet foreslår holdet en model, hvor loops er forbigående og kun dannes forbigående som svar på specifikke regulatoriske signaler.
"Sløjfer er overalt i cellerne, men de er meget mere forbigående, end nogen tidligere har forstået," sagde co-senior forfatter Gene Yeo, PhD, professor i cellulær og molekylær medicin og direktør for Center for RNA Biology ved UC San Diego School af medicin. "Vores resultater omstøder antagelsen om, at disse sløjfer er fastkablede funktioner, der er fastkablet og indebærer, at sløjfedannelse er dynamisk reguleret."
Hvordan celler læser genomet for at lave funktionelle produkter
Celler skal nøje regulere, hvordan de læser instruktioner fra deres genomer for at producere funktionelle produkter, såsom proteiner, der udfører forskellige cellulære funktioner. Den måde, celler pakker, arrangerer og folder deres genom i tre dimensioner, spiller en afgørende rolle i genregulering. Disse organisatoriske principper bestemmer, hvilke segmenter af DNA der tilgås og fortolkes af det cellulære maskineri, der er ansvarligt for at læse og transskribere gener.
Den mest fundamentale enhed for genomfoldning involverer looping, hvor fjerne områder af DNA fysisk kommer i kontakt med hinanden for at hjælpe med at orkestrere genekspression. Loops menes at samle proteiner, der driver genekspression, hvilket giver dem mulighed for at interagere og udføre deres funktioner effektivt.
COLA belyser den dynamiske karakter af DNA-løkker
COLA-tilgangen gør det muligt for forskere at visualisere loops og andre genomiske træk samtidigt, og fange rumlige og tidsmæssige forhold i hidtil uset detaljer. "Vi har nu en metode, der direkte kan korrelere ændringer i RNA-polymerasebelægning med specifikke løkkeændringer i enkelte celler," sagde medforfatter Michael Niculescu III, en kandidatstuderende i Yeo Lab.
Forskerne siger, at deres resultater har brede implikationer for forståelsen af genekspression og genomorganisation. For eksempel antyder de, at den dynamiske og fluktuerende karakter af loops kunne tillade celler at reagere hurtigt på miljøændringer eller udviklingsmæssige tegn. I cancer og neurodegenerative sygdomme kan denne fleksibilitet også give celler mulighed for at skifte celleidentitet og omprogrammere genekspressionsprogrammer.
"Der er meget, vi nu kan gøre med dette værktøj," sagde medforfatter Matthew Huynh, en postdoc-stipendiat i Yeo Lab. "Vi kan teste nye hypoteser og undersøge sygdomsrelaterede reguleringsændringer på måder, vi ikke kunne før."
Yderligere medforfattere
Yderligere medforfattere til denne undersøgelse er:Yuzuru Kido, UC San Diego; James McGinnis, UC San Diego; og Nicholas Ingolia, UC Berkeley.
Denne forskning blev delvist finansieret af National Institutes of Health (R35 GM143669, T32 GM007240), National Science Foundation (MCB-2110538), Simons Foundation (540333) og Ludwig Institute for Cancer Research.
Sidste artikelHvordan nogle væv kan 'ånde' uden ilt
Næste artikelNy forskning viser, hvordan amyloid beta kommer ind i hjerneceller
 Varme artikler
Varme artikler
-
 Hungrig bjørne -krise griber fat i den østlige russiske regionRussiske skovbrugsembedsmænd sagde, at 86 bjørne skulle skydes ihjel, fordi de var fjendtlige Et stort antal sultne, aggressive bjørne nærmer sig mennesker og har dræbt to mennesker i Ruslands fje
Hungrig bjørne -krise griber fat i den østlige russiske regionRussiske skovbrugsembedsmænd sagde, at 86 bjørne skulle skydes ihjel, fordi de var fjendtlige Et stort antal sultne, aggressive bjørne nærmer sig mennesker og har dræbt to mennesker i Ruslands fje -
 Tænk på din første hukommelse - hvorfor kan du ikke huske det endnu tidligere?BrainStuff:Hvorfor kan folk ikke huske at være født? HowStuffWorks Odds er, du husker mange forskellige ting. Din 18 års fødselsdag. Gå på gymnasiet den første dag. Sidste gang du spiste en burrito.
Tænk på din første hukommelse - hvorfor kan du ikke huske det endnu tidligere?BrainStuff:Hvorfor kan folk ikke huske at være født? HowStuffWorks Odds er, du husker mange forskellige ting. Din 18 års fødselsdag. Gå på gymnasiet den første dag. Sidste gang du spiste en burrito. -
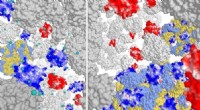 Hvordan ribosomer former proteometHøjre panel:interaktion af positivt ladede proteiner (mørkeblå) med ribosomkomplekset (lyseblå/gul). Negativt ladede proteiner interagerer ikke. Ved høj ionstyrke (venstre panel) interagerer de positi
Hvordan ribosomer former proteometHøjre panel:interaktion af positivt ladede proteiner (mørkeblå) med ribosomkomplekset (lyseblå/gul). Negativt ladede proteiner interagerer ikke. Ved høj ionstyrke (venstre panel) interagerer de positi -
 Kæledyr eller trusler? Guldfisk kan være skadelig for biodiversitetenKredit:Unsplash/CC0 Public Domain Invasive arter er en af de førende årsager til globalt tab af biodiversitet, og kæledyrshandlen er ansvarlig for en tredjedel af alle akvatiske invasive arter. K
Kæledyr eller trusler? Guldfisk kan være skadelig for biodiversitetenKredit:Unsplash/CC0 Public Domain Invasive arter er en af de førende årsager til globalt tab af biodiversitet, og kæledyrshandlen er ansvarlig for en tredjedel af alle akvatiske invasive arter. K
- 3D-materiale fundet til at nedbryde antidepressivt middel, der forurener vandområder verden over
- Astronauter fuldfører 2. rumvandring for at bytte stationsbatterier (opdatering)
- Beviser tyder på, at flere mega-tørke er på vej
- Tager gætværket ud af retsmedicinsk analyse af fingermærker
- Delhi ramt af sjælden sommerluftforureningsalarm
- Arktisk permafrosttø spiller en større rolle i klimaændringerne end tidligere anslået


