Hvordan ribosomer former proteomet
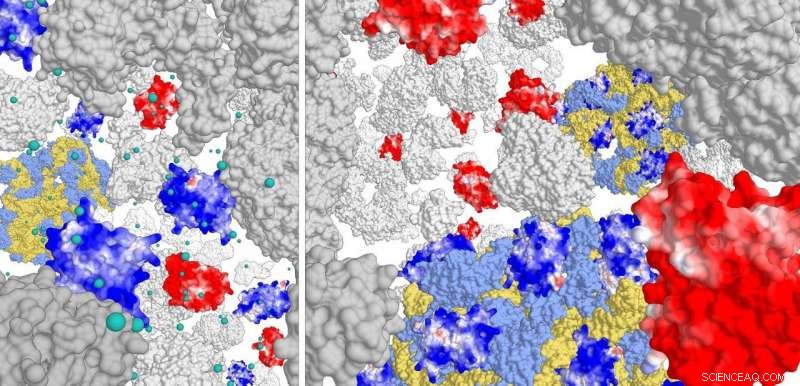
Højre panel:interaktion af positivt ladede proteiner (mørkeblå) med ribosomkomplekset (lyseblå/gul). Negativt ladede proteiner interagerer ikke. Ved høj ionstyrke (venstre panel) interagerer de positive proteiner næsten ikke med ribosomet. Kredit:Poolman lab, Universitetet i Groningen
Celler er fyldt med makromolekyler, som begrænser udbredelsen af proteiner, især i prokaryote celler uden aktiv transport i cytoplasmaet. Mens man undersøger forholdet mellem trængsel, ionstyrke og proteindiffusion, Biokemikere fra University of Groningen gjorde en fascinerende opdagelse:positivt ladede proteiner klæber til overfladen af ribosomkomplekser. Dette forklarer, hvorfor de fleste vandopløselige proteiner har en samlet negativ ladning. Opdagelsen vil snart blive vist i journalen eLife .
Bevægelseshastigheden af proteiner inde i celler er vigtig:Mange processer i biologiske celler afhænger af interaktioner mellem makromolekyler (proteiner og nukleinsyrer) og dermed af deres evne til at finde hinanden. "Men cellecytoplasmaet er et travlt sted, og dette vil påvirke protein- og RNA-diffusion", bemærkninger University of Groningen Professor i biokemi Bert Poolman.
Afgifter
Hans gruppe studerede virkningerne af crowding på diffusion, og fandt en sammenhæng mellem proteinstørrelse og diffusionshastighed. "Men for nogle proteiner fandt vi ikke denne sammenhæng, så vi satte os for at undersøge hvorfor.' Holdet brugte tre forskellige prokaryoter med stigende ionstyrke:den Gram-negative bakterie Escherichia coli, den gram-positive Lactococcus lactis og den ekstremofile Haloferax volcanii, som lever ved meget høje saltkoncentrationer.
Til denne undersøgelse, forskerne konstruerede forskellige varianter af Green Fluorescent Protein (GFP), med overfladeladninger fra -30 til +25. De undersøgte derefter bevægelsen af disse GFP-varianter i de tre celletyper. 'Vi så, at positivt ladede proteiner ville diffundere meget langsomt. De sad fast i cellen', forklarer Poolman. Yderligere analyse viste, at de positive proteiner ikke binder sig til DNA'et eller cellemembranen, men til ribosomkomplekset.
Interessant
En bioinformatisk analyse af mikroorganismers og eukaryote cellers proteomer viste, at i de fleste tilfælde er omkring 70 procent af proteinerne negativt ladede. 'Interessant nok, de resterende 30 procent er enten membranproteiner eller proteiner involveret i ribosomets eller mRNA's funktion eller foldning.'
Membranproteinerne er afskærmet af chaperoner under biogenese, så de ikke klæber til ribosomerne. Der er derfor ingen 'frie' cytoplasmatiske proteiner med en høj nok positiv ladning til at få dem til at sætte sig på ribosomer. Den negative ladning af ribosomkomplekset og den omgivende ionstyrke af cytoplasmaet ser ud til at have formet udviklingen af ladninger i det cellulære proteom.
Uventet
Den nye og uventede indsigt i, at proteinmobilitet er en funktion af proteinladning, kan forklare, hvorfor det er svært at udtrykke nogle proteiner i bakteriesystemer med lav ionstyrke. 'Vi observerede, at en højere ionstyrke reducerer klæbrigheden af positivt ladede proteiner. Det kunne være en værdifuld indsigt for konstruktionen af proteinekspressionsplatforme.'
En sidste observation i eLife papiret er, at genomerne fra flere endosymbionter viser en overflod af positivt ladede proteiner. 'Dette fund forbløffer os virkelig', indrømmer Poolman. 'Du ville forvente, at alle disse proteiner ville blive tiltrukket af endosymbionternes ribosomer. Indtil nu, vi har ingen forklaring på, hvordan disse organismer er i stand til at håndtere langsom diffusion og ribosomer, der bliver opslugt af positive proteiner.'
 Varme artikler
Varme artikler
-
 Sådan fungerer den biologiske antropologiFysisk udseende er en stor del af, hvordan vi laver et match i dating. Trods alt, er du ikke interesseret i din potentielle makkers øreflippelængde? Hvis du var en biologisk antropolog, det kan du bar
Sådan fungerer den biologiske antropologiFysisk udseende er en stor del af, hvordan vi laver et match i dating. Trods alt, er du ikke interesseret i din potentielle makkers øreflippelængde? Hvis du var en biologisk antropolog, det kan du bar -
 Hvorfor har de fleste mennesker 23 par kromosomer?Næsten hver menneskelig celle har 23 par kromosomer for i alt 46. Lawrence Lawry/Getty Images Du har måske hørt, at 137 er det magiske tal, men hvis du spørger en genetiker, de vil fortælle dig, at d
Hvorfor har de fleste mennesker 23 par kromosomer?Næsten hver menneskelig celle har 23 par kromosomer for i alt 46. Lawrence Lawry/Getty Images Du har måske hørt, at 137 er det magiske tal, men hvis du spørger en genetiker, de vil fortælle dig, at d -
 Tre grunde til, at celdeling er vigtigTidligt i biologiens historie mente forskere, at celler opstod spontant. Med udviklingen af celleteorien indså folk endelig, at kun celler kan få andre celler. Faktisk er to kategorier, der definere
Tre grunde til, at celdeling er vigtigTidligt i biologiens historie mente forskere, at celler opstod spontant. Med udviklingen af celleteorien indså folk endelig, at kun celler kan få andre celler. Faktisk er to kategorier, der definere -
 Forskere opdager ny kvægsygdom og forhindrer den i at sprede sigKredit:Københavns Universitet Efter genetiske undersøgelser af deforme kalve, er forskning udført på Københavns Universitet i stand til at afdække en hidtil ukendt sygdom fundet blandt Holstein-kv
Forskere opdager ny kvægsygdom og forhindrer den i at sprede sigKredit:Københavns Universitet Efter genetiske undersøgelser af deforme kalve, er forskning udført på Københavns Universitet i stand til at afdække en hidtil ukendt sygdom fundet blandt Holstein-kv
- Sådan finder du omvendt af en funktion
- Størrelse betyder noget, da nanokrystaller går gennem faser
- Forskere dokumenterer udbredte metanudsivninger ud for Oregons kyst
- Stamme nær Brasiliens største by kæmper for at holde fast
- Sådan beregner du et temperaturområde
- Ny indsigt i halvledere til spintroniske applikationer fra hård røntgenfotoemission


