Den skjulte orden i DNA-diffusion

En ny enkelt-molekyle sporingsmetode baseret på fluorescens molekylær billeddannelse afslørede ikke-tilfældig bevægelse af DNA-molekyler. Kredit:© KAUST Anastasia Khrenova
En anden tilgang til at analysere bevægelsen af diffuserende molekyler har hjulpet med at omstøde den langvarige antagelse om, at DNA-molekyler bevæger sig på en tilfældig måde. KAUST-forskere afslører for første gang, at DNA-molekyler bevæger sig ikke ved tilfældig Brownsk bevægelse, men ved en ikke-tilfældig gang relateret til polymerdynamik på en måde, der bevarer de overordnede Brownske egenskaber.
"Brownsk bevægelse er en proces, hvorved molekyler bevæger sig tilfældigt i en væske ved at kollidere med andre molekyler, " forklarede Dr. Maged Serag, en postdoc i Bioscience ved KAUST. "I levende celler, Brownsk bevægelse gør det muligt for molekyler at bevæge sig hurtigt og effektivt mellem celleorganeller og interagere med andre molekyler."
I mange årtier, forskere har brugt en relativt simpel test til at bestemme, om molekylær diffusion er Brownsk:når middel-kvadratforskydningen (MSD) af en population af molekyler stiger lineært over tid. I et ensartet medium som rent vand, dette betyder, at en dråbe saltvandsopløsning vil udvide sig med en hastighed, der får MSD til at stige lineært med tiden.
DNA er i overensstemmelse med denne makroskala diffusionsadfærd, og derfor er det blevet antaget, at dens bevægelse er Brownsk ligesom andre molekyler. Imidlertid, det er også kendt, at DNA, er et langt polymermolekyle, vrider sig spontant på grund af intramolekylære kræfter.
"DNA-molekylet kan ses som en semi-fleksibel kæde, " sagde Serag. "Hvis vi følger dens bevægelse på korte tidsskalaer og i et rum tæt på dens størrelse, vi ser ormelignende bevægelsesadfærd."
Serag og kollega lektor Satoshi Habuchi satte sig for at se, om denne vridningsbevægelse kunne påvirke spredningen af DNA.
"Dr. Serag kom med en unik idé til at beskrive bevægelsen af et molekyle baseret på sandsynligheden for at besætte gittersteder snarere end ved middel-kvadratforskydning, " sagde Habuchi. "MSD har været standardmetoden til at detektere afvigelser fra Brownsk bevægelse, men det afslører ikke nogen ikke-tilfældig bevægelse for DNA-molekyler. Ved at bruge denne probabilistiske tilgang i stedet, vi var i stand til at detektere og kvantificere skjult ikke-tilfældig bevægelse."
Ved at udvikle en ny teoretisk ramme, hvor bevægelse modelleres på en trinvis måde, der tager højde for molekylær bøjning, DNA-molekyler blev fundet at bevæge sig ikke-tilfældigt med varieret hastighed og molekylært 'spor' på en måde, der præcist bevarede den Brownske lineære MSD.
"Det vigtigste resultat af denne undersøgelse er, at vi har påvist, at en lineær MSD ikke altid indikerer underliggende Brownsk bevægelse, " forklarede Habuchi. "Med denne nye teoretiske ramme, vi kan detektere den ikke-tilfældige bevægelse af enkelte molekyler, som ikke kan fanges ved konventionel MSD-analyse."
 Varme artikler
Varme artikler
-
 Forskere opretter første sammenhængsforbindelsesforbindelseEt kunstnerindtryk af et kvante netværk baseret på kvælstof -ledige qubits i diamant. Kredit:Scixel/TU Delft Det er lykkedes forskere ved QuTech i Delft at generere kvanteindvikling mellem to kvan
Forskere opretter første sammenhængsforbindelsesforbindelseEt kunstnerindtryk af et kvante netværk baseret på kvælstof -ledige qubits i diamant. Kredit:Scixel/TU Delft Det er lykkedes forskere ved QuTech i Delft at generere kvanteindvikling mellem to kvan -
 Magnetisk effekt uden magnetMaterialet opfører sig, som om magnetiske monopoler var til stede. Kredit:Vienna University of Technology Elektrisk strøm afbøjes af et magnetfelt - i ledende materialer, dette fører til den såkal
Magnetisk effekt uden magnetMaterialet opfører sig, som om magnetiske monopoler var til stede. Kredit:Vienna University of Technology Elektrisk strøm afbøjes af et magnetfelt - i ledende materialer, dette fører til den såkal -
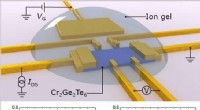 En elektrisk kontakt til magnetisme(Top) Skematisk felteffekttransistor baseret på ultratynd ferromagnetisk halvleder Cr2Ge2Te6. Materialet er dækket med en iongel for at forstærke felteffekten. (Bund) Magneto-modstand (MR) med stigend
En elektrisk kontakt til magnetisme(Top) Skematisk felteffekttransistor baseret på ultratynd ferromagnetisk halvleder Cr2Ge2Te6. Materialet er dækket med en iongel for at forstærke felteffekten. (Bund) Magneto-modstand (MR) med stigend -
 Ny optisk lagringsteknologi med ultrahøj tæthedKredit:Kazan Federal University Ifølge de nuværende skøn, snesevis af zettabytes information vil blive lagret elektronisk i 2020, som vil stole på fysiske principper, der letter brugen af enkelt
Ny optisk lagringsteknologi med ultrahøj tæthedKredit:Kazan Federal University Ifølge de nuværende skøn, snesevis af zettabytes information vil blive lagret elektronisk i 2020, som vil stole på fysiske principper, der letter brugen af enkelt
- Zink-luftbatterier giver strøm i fjerntliggende områder
- Forbedrer teknologien virkelig vores beslutningsevne?
- Californiens skovbrand eksploderer, brænder over 25 miles om dagen
- Hvordan frostvæskeproteiner får iskrystaller til at vokse
- Måder at øge kinetisk energi
- Sådan beregnes forbrændingsvarmen af paraffinvoks


