Team identificerer universalitet og specificitet i proteinbevægelser
Selvom proteiner har meget forskellige funktionsfunktioner, eller specialiteter, i levende celler, de deler de generelle karakteristika – den samme universalitet – i deres bevægelser, siger forskere fra University of Oregon.
Deres bevægelse er meget som bjergskred eller skovbrande, rapporterer UO-holdet i et papir offentliggjort i tidsskriftet Fysisk gennemgangsbreve .
Fundene, som var afhængig af teoretiske metoder udviklet i laboratoriet hos UO-kemikeren Marina Guenza, kan hjælpe med at finde ud af, hvor proteinbinding kan forekomme, og hvordan proteinbevægelse er involveret i processen.
"Dynamikken er ofte uudforsket i studiet af proteinbinding, men det kunne være en afgørende faktor for den måde, proteiner regulerer deres egen biologiske aktivitet på, " sagde Guenza. "Proteiner i biologiske systemer er begrænset til en maksimal størrelse og bevæger sig inden for en smal, temperaturafhængigt tidsvindue, hvori de er tilgængelige til at binde med andre molekyler, så det ser ud til, at alle proteiner burde have nogle universelle egenskaber, der styrer deres adfærd. Vi søgte at finde denne universelle adfærd ved at bruge vores teoretiske tilgang."
I det interval, hvor saltkoncentrationer og volumen også er vigtige, fandt forskerne, et kritisk hydrogenbindingsnetværk leverer tilfældige energiudsving, der gør eller bryder et proteins evne til at binde sig til et andet molekyle.
Energiudsving og proteinbevægelser blev undersøgt ved at bruge Langevin Equation for Protein Dynamics-metoden udviklet i Guenza-laboratoriet.
"Mange velkendte teoretiske metoder forsøger at studere proteindynamik, men de mangler vigtige aspekter af den involverede fysik, " sagde Mohammadhasan Dinpajooh, en postdoc-forsker. "Ved at inkorporere den væsentlige fysik, vi kan optrevle biologiske mekanismer, som stadig er uhåndgribelige i velavancerede røntgen- eller kernemagnetiske resonanseksperimenter."
Arbejdet var et resultat af teamwork startet af Jeremy Copperman, en tidligere fysikdoktorand i laboratoriet, og fortsat af Dinpajooh og Eric Beyerle, en tredjeårs kandidatstuderende.
"Vi udviklede en måde at præcist beskrive den specifikke funktionelle dynamik af et protein på protein-for-protein-basis. I processen, vi bemærkede en tendens – et skaleringsmønster – som ikke havde nogen grund til at være der, " sagde Copperman, nu postdoc-forsker ved University of Wisconsin-Milwaukee. "Vi simulerede proteinbevægelsen på supercomputere, brugte måneder på at skrive analysekoder og fandt mængder af dette simple skaleringsmønster."
Med den information, Guenza sagde, holdet var i stand til at kortlægge ligevægten af proteiner, der eksisterer i et stadigt svingende system, der bevæger sig tilfældigt og på samme måde til frontlinjen af en naturbrand.
Forskerne tog udgangspunkt i strukturel information baseret på nuklear magnetisk resonans eller røntgenteknikker, og simulerede 14 adfærdsdynamikker i 12 proteiner i tider fra 50 nanosekunder til 1,23 millisekunder. Kort sagt, forskerne foreslår, at proteiners bevægelse kan beskrives ved en simpel fluktuation, der udsættes for tilfældige energiske lyde, hvilket er som at klatre over en bjergkæde, der konstant skifter og omarrangerer sig tilfældigt.
Undersøgelsens tilgang til at forstå proteindynamik kan være nyttig for medicinalindustrien, sagde Guenza.
"Brancheforskere tester en masse organiske molekyler og kan se, at et molekyle stopper et proteins funktion, men de ved ikke hvordan det virker, " sagde hun. "Vores tilgang kunne give dem mulighed for at forstå disse mekanismer og i sidste ende at manipulere strukturen af deres lægemiddel, så det finder bindingsstedet og sikrer en bedre tilpasning på målstedet for proteinet."
 Varme artikler
Varme artikler
-
 Subkritisk eksperiment indfanger videnskabelige målinger for at fremme lagersikkerhedenKollaboratørklistermærker prikker overfladen af indespærringsfartøjet, der blev brugt i Lawrence Livermore National Laboratorys Ediza subkritiske eksperiment. Kredit:Lawrence Livermore National Labo
Subkritisk eksperiment indfanger videnskabelige målinger for at fremme lagersikkerhedenKollaboratørklistermærker prikker overfladen af indespærringsfartøjet, der blev brugt i Lawrence Livermore National Laboratorys Ediza subkritiske eksperiment. Kredit:Lawrence Livermore National Labo -
 Forskere afslører 3D-bevægelse af nematoden C. elegansJerzy Blawzdziewicz, professor, medformand og direktør for kandidatstudier på Institut for Maskinteknik, og Siva Vanapalli, en lektor og Bill Sanderson fakultet stipendiat i Department of Chemical Eng
Forskere afslører 3D-bevægelse af nematoden C. elegansJerzy Blawzdziewicz, professor, medformand og direktør for kandidatstudier på Institut for Maskinteknik, og Siva Vanapalli, en lektor og Bill Sanderson fakultet stipendiat i Department of Chemical Eng -
 Undersøgelse af et øjebliksbillede af eksploderende iltUnder eksplosionen af et iltmolekyle:røntgenlaseren XFEL slår elektroner ud af de to atomer i iltmolekylet og sætter det i gang. Under fragmenteringen, røntgenlaseren frigiver endnu en elektron fra
Undersøgelse af et øjebliksbillede af eksploderende iltUnder eksplosionen af et iltmolekyle:røntgenlaseren XFEL slår elektroner ud af de to atomer i iltmolekylet og sætter det i gang. Under fragmenteringen, røntgenlaseren frigiver endnu en elektron fra -
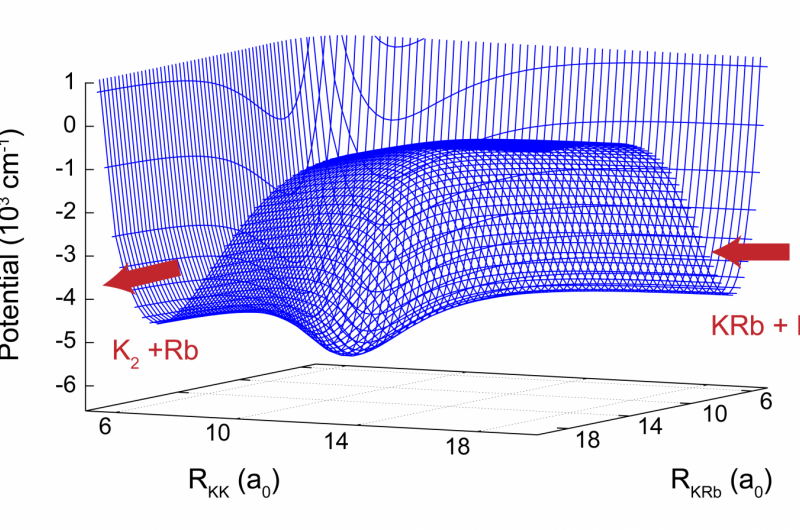 Simulering afslører universel signatur af kaos i ultrakølede reaktionerEt todimensionalt udsnit af den potentielle energioverflade til K + KRb-reaktionen. Reaktionen fortsætter fra højre til venstre. I mellemområdet er en dyb brønd tydeligt synlig, hvilket fører til kaot
Simulering afslører universel signatur af kaos i ultrakølede reaktionerEt todimensionalt udsnit af den potentielle energioverflade til K + KRb-reaktionen. Reaktionen fortsætter fra højre til venstre. I mellemområdet er en dyb brønd tydeligt synlig, hvilket fører til kaot
- Sådan beregnes Ampacity
- Hvad er zinklegering?
- Magnonic nanoantenner:optisk inspireret databehandling med spin-bølger et skridt nærmere
- Forskere opretter advarselssystem for giftige alger i søer
- Konkurrenter, der propper tv-streamingarena
- Vestantarktis iskollaps kan forhindres ved at sne havvand på den


