Fysik forklarer proteinets uforudsigelighed
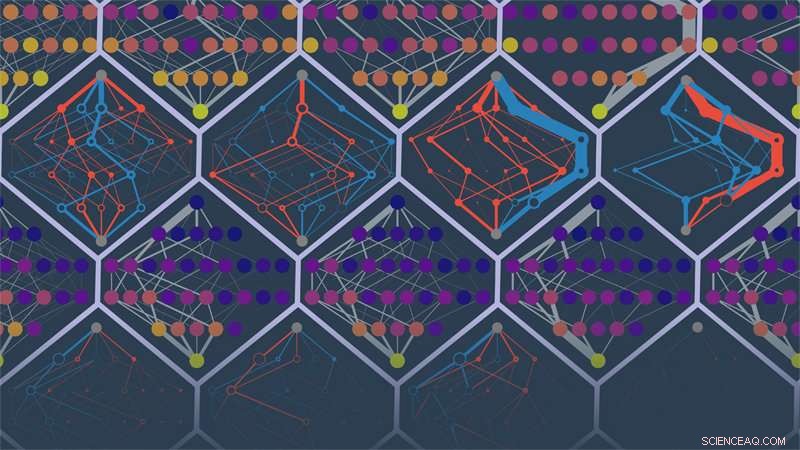
Forskere fra University of Oregon lab af Michael Harms forudsagde de evolutionære stier for forskellige simulerede proteiner. Punkterne på billedet er proteinsekvenser. Kanterne angiver sandsynligheder for baner. Farver angiver lav til høj kondition (lilla til gul) eller falsk utilgængelige eller forkert tilgængelige ruter (rød og blå). Kredit:Michael Harms og Zach Sailer
Forskere fra University of Oregon teoretiserede, at de kunne manipulere et protein en mutation ad gangen og forudsige dets udvikling. De søgte at bevise det. Og mislykkedes.
De tror, imidlertid, at de har fundet en grundlæggende sandhed, der ligger til grund for uforudsigeligheden i et biologisk system. Grundlæggende fysiske begrænsninger gør usikkerhed til normen, de rapporterede i et papir offentliggjort online 23. oktober i Proceedings of the National Academy of Sciences .
"Selvom vi fik et overraskende negativt resultat, vi var i stand til at sige hvorfor, " sagde Michael J. Harms, en professor i UO Institut for Kemi og Biokemi og videnskabsmand ved Institut for Molekylær Biologi. "Det er positivt. Vores enkle undersøgelse bekræfter, hvad mange mennesker i feltet har observeret gentagne gange - uforudsigelighed. Det ser ud til, at det er universelt."
Forskningen var en digital affære, udført med computersimuleringer designet af UO ph.d.-studerende Zachary R. Sailer. Han og Harms skabte et simpelt gitterprotein, ved at bruge en tilgang, der tidligere er skabt i Harms lab, med en tilfældig sekvens på 12 aminosyrer. De kørte derefter evolutionære simuleringer for at optimere stabiliteten, en fysisk egenskab ved proteinet.
Målet var at bruge virkningerne af alle 228 mutationer, der vides at være forbundet med startproteinet, til at forudsige disse simulerede baner:hvilken mutation ville forekomme, hvornår, over tid. Evnen til at projicere frem forsvandt hurtigt efter de første to mutationer. Efter det, de forventede baner kom på afveje midt i et stigende antal omdirigeringssandsynligheder.
"Kvaliteten af dine oplysninger falder faktisk over tid, " sagde Sailer. "Når mutationer akkumuleres, virkningerne af de mutationer, du målte, begynder at ændre sig, så du ikke kan forudsige, hvor du er på vej hen."
I deres papir, Sailer og Harms antyder, at fysik, især termodynamik, er på spil. Hver mutation ændrer proteinet i en lille, men ikke-lineær måde. Det betyder, at effekten af hver mutation afhænger af alle mutationer, der er sket før.
"Jeg tror, at det, vi viste, grundlæggende, er, at selvom du ved meget om et system, om et protein, du kan ikke forudsige, hvordan det udvikler sig på grund af systemets fysik, " sagde Harms. "Der er fysiske regler, der begrænser evolutionen og dens forudsigelighed."
Hvordan proteiner udvikler sig er et grundlæggende spørgsmål i evolutionær biologi, fra både et filosofisk perspektiv, at lære mere om maskineriet i biologiske systemer, og for spor, der kan føre til forbedrede eller bedre stoffer.
"Praktisk set, Harms sagde, "vores forskning kan hjælpe os med at lære, hvordan man forhindrer udviklingen af antibiotikaresistens i bakterier." Næsten alle bakteriebårne infektioner udvikler resistens over for antibiotika, skabe et førende folkesundhedsproblem rundt om i verden.
"I stedet for at studere virkningerne af alle individuelle mutationer, " Sailer tilføjede, "måske skulle vi studere tilfældige kombinationer af mange mutationer. En sådan tilgang kan måske hjælpe os med at forudsige udviklingen af resistens."
Arbejdet er i gang i Harms-laboratoriet for at teste for denne mulighed på rigtige proteiner. "Vi bygger beregningsværktøjer, der lader os analysere antibiotikaresistensdatasæt, og vi får hints om, at en kombinatorisk tilgang virker, " sagde Harms. "Det er mere kompliceret end at studere individuelle mutationer, men vores arbejde viser, at det er usandsynligt, at den individuelle tilgang virker."
Sidste artikelForskere udvikler databus til kvantecomputer
Næste artikelChipbaserede sensorer med utrolig følsomhed
 Varme artikler
Varme artikler
-
 Forøgende ladningstæthed bølgesvingninger ses i superledende kobberoxider for første gangDenne skitse viser, hvordan resonant uelastisk røntgenspredning (RIXS) hjælper forskere med at forstå den elektroniske adfærd af kobberoxidmaterialer. En røntgenfoton rettet mod prøven (blå pil) absor
Forøgende ladningstæthed bølgesvingninger ses i superledende kobberoxider for første gangDenne skitse viser, hvordan resonant uelastisk røntgenspredning (RIXS) hjælper forskere med at forstå den elektroniske adfærd af kobberoxidmaterialer. En røntgenfoton rettet mod prøven (blå pil) absor -
 Brud på Wiedemann-Franz-lovenKunstnerisk indtryk af de to temperatur-ubalancerede reservoirer af kolde atomer forbundet via en kvantepunktskontakt. Temperaturbalancen induceres af en laserstråle. Kredit:ETH Zurich/Esslinger -grup
Brud på Wiedemann-Franz-lovenKunstnerisk indtryk af de to temperatur-ubalancerede reservoirer af kolde atomer forbundet via en kvantepunktskontakt. Temperaturbalancen induceres af en laserstråle. Kredit:ETH Zurich/Esslinger -grup -
 Ultrakoldt atominterferometri i rummetAbsorptionsbillede af atomskyen ved en udgangsport på interferometeret. To stribeformede moduleringer er synlige, hvilket fører til et tjekket mønster i tæthedsfordelingen. Den førstnævnte struktur er
Ultrakoldt atominterferometri i rummetAbsorptionsbillede af atomskyen ved en udgangsport på interferometeret. To stribeformede moduleringer er synlige, hvilket fører til et tjekket mønster i tæthedsfordelingen. Den førstnævnte struktur er -
 Flip-flop qubits:Radikalt nyt kvanteberegningsdesign opfundetKunstners indtryk af flip-flop qubits, der udviser kvanteforvikling. Kredit:Tony Melov/UNSW Ingeniører ved Australiens University of New South Wales har opfundet en radikal ny arkitektur til kvant
Flip-flop qubits:Radikalt nyt kvanteberegningsdesign opfundetKunstners indtryk af flip-flop qubits, der udviser kvanteforvikling. Kredit:Tony Melov/UNSW Ingeniører ved Australiens University of New South Wales har opfundet en radikal ny arkitektur til kvant
- Estimater af emissioner fra naturgasdrevne anlæg er alt for lave, undersøgelse finder
- Tredimensionel selvsamling ved hjælp af dipolær interaktion
- Sådan konverteres tommer til Mm
- Nanokrystaller udsender lys ved effektivt at tunnelere elektroner
- Insekter, der lever underjordiske
- Fremskridt inden for førerløse fly kan styre fremtiden for droner, flyvningen


