Fysiker optimerer DNA-mikroskopiteknik for at forbedre billeddannelseshastigheden, tilføje farve
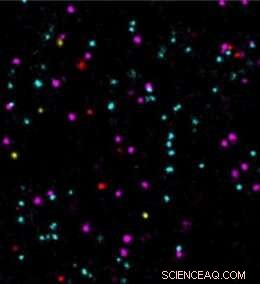
Kredit:S. Strauss/MPI of Biochemistry
Super-opløsning fluorescensmikroskopi kan bruges til at visualisere strukturer mindre end 200 nanometer, dvs. under lysets diffraktionsgrænse. En af mikroskopiteknikkerne, kaldet DNA-PAINT, blev udviklet af Ralf Jungmann, forskningsgruppeleder ved MPI for Biokemi og professor i eksperimentel fysik ved LMU, sammen med kollegaer. Teknikken bruger korte 'imagere', farvestof-mærkede DNA-strenge, der midlertidigt binder til deres målmolekyler på en komplementær måde for at frembringe det nødvendige "blink" til superopløsningsrekonstruktion af billederne.
"Vi har for nylig forbedret DNA-PAINTs traditionelt ret langsomme optagelseshastighed med en størrelsesorden ved at optimere DNA-sekvensdesign, " siger Jungmann. "Men dette kom på bekostning af at miste multipleksing, hvilket betyder, at flere strukturer i cellen ikke kan observeres samtidigt, " tilføjede Jungmann. Den samtidige observation af flere proteiner, imidlertid, er vigtig for en bedre forståelse af komplekse signalkaskader mellem tumorceller og normale celler.
Denne multipleksingsevne var ikke opnåelig i hastighedsoptimeret DNA-PAINT, da kun en enkelt optimeret sekvens med forbedrede hybridiseringskarakteristika var tilgængelig. "Vi spurgte os selv, hvordan vi tillader multiplekset billeddannelse og, på samme tid, skub billedoptagelseshastigheden endnu længere, " siger Sebastian Strauss, første forfatter til papiret og kollega i Jungmanns gruppe.
I den aktuelle undersøgelse, forskerne præsenterer et nyt koncept, der med succes har forbedret billeddannelseshastigheden. De udnyttede det faktum, at frekvensen af binding af billedkameraerne til deres målstrenge skaleres lineært med antallet af tilgængelige bindingssteder. "Jo flere bindingssteder der er, jo hurtigere forløber billedoptagelsen. Imidlertid, blot at sammenkæde bindingssteder ville føre til uønsket lange docking-sekvenser, potentielt reducere opnåelig billedopløsning og øge uspecifik binding, " siger Strauss. For at omgå disse problemer, forskerne designet gentagne sekvensmotiver, f.eks. (TCC)n, som kunne sammenkædes for at tilvejebringe overlappende bindingssteder, men dog kun lidt stigende strenglængde. "Vi designede seks individuelle, periodiske sekvensmotiver, som giver os mulighed for at introducere multipleksing til hastighedsoptimeret DNA-PAINT, " sagde Strauss. "I kombination med tidligere forbedringer, vi kan nu fremskynde DNA-PAINT med en faktor på 100, " tilføjer Jungmann.
For at optimere de nye sekvensmotiver og benchmarke deres forbedringer, gruppen brugte DNA origami strukturer, som er selvmonterede, nanometer-store DNA-objekter, der autonomt foldes til foruddefinerede former. Disse strukturer kan bruges til at arrangere DNA-PAINT-bindingssteder med præcis afstand ved f.eks. 5-nm afstande. Dette gjorde det muligt for forskerne at evaluere forbedringerne af DNA-PAINT under definerede forhold. "De nye optimerede DNA-sekvenser gjorde det muligt for os at løse seks forskellige DNA-origami-strukturer i stedet for kun én, på få minutter, " forklarer Strauss.
"Vi er begejstrede for at anvende den nu yderligere forbedrede billeddannelseshastighed i DNA-PAINT til at tackle biologiske spørgsmål. F.eks. tumormarkører kunne tidligere kun undersøges langsomt og ikke klart på et enkelt molekyle niveau. I vores undersøgelse, målingen af fire forskellige tumormarkører bekræfter en hurtig og nøjagtig analyse af deres molekylære positioner og interaktioner. Dette kunne give vigtig indsigt i lægemiddeludvikling og deres virkningsmekanismer, " slutter Jungmann.
 Varme artikler
Varme artikler
-
 Forskere satte et nyt spin på køling af elektroniske hotspotsLektor Junxia “Lucy” Shi og hendes team af forskere i Advanced Semiconductor Materials and Devices Laboratory ved UIC fokuserer på at bruge en ny metode til afkøling af hotspots i elektronik og fange
Forskere satte et nyt spin på køling af elektroniske hotspotsLektor Junxia “Lucy” Shi og hendes team af forskere i Advanced Semiconductor Materials and Devices Laboratory ved UIC fokuserer på at bruge en ny metode til afkøling af hotspots i elektronik og fange -
 Dråber udfører vovede bedrifter på geloverfladerMartin Coux, Piaget Scientific Award 2018. Kredit:DR Velkommen til den fantastiske verden af bløde underlag. Disse materialer er lavet af silikonegeler og har samme tekstur som panna cotta - men
Dråber udfører vovede bedrifter på geloverfladerMartin Coux, Piaget Scientific Award 2018. Kredit:DR Velkommen til den fantastiske verden af bløde underlag. Disse materialer er lavet af silikonegeler og har samme tekstur som panna cotta - men -
 Dynamisk optisk kohærens tomografi i fuld felt:3D-billeddannelse af nethindeorganoideren, Opsætning til at oprette FFOCT- og D-FFOCT-billeder, kombineret med fluorescens til validering. b, 3D-repræsentation af en del af en 28 dage gammel (D28) hiPSC-afledt retinal organoid fanget med D
Dynamisk optisk kohærens tomografi i fuld felt:3D-billeddannelse af nethindeorganoideren, Opsætning til at oprette FFOCT- og D-FFOCT-billeder, kombineret med fluorescens til validering. b, 3D-repræsentation af en del af en 28 dage gammel (D28) hiPSC-afledt retinal organoid fanget med D -
 Forskere gør spring i simulering af kvantepartiklerKredit:University of St Andrews Ny kvanteteorisk forskning, ledet af akademikere ved University of St Andrews School of Physics, kunne transformere den måde, forskere forudsiger, hvordan kvantepar
Forskere gør spring i simulering af kvantepartiklerKredit:University of St Andrews Ny kvanteteorisk forskning, ledet af akademikere ved University of St Andrews School of Physics, kunne transformere den måde, forskere forudsiger, hvordan kvantepar


