Komplekse mønstre:Byg en bro fra det store til det lille
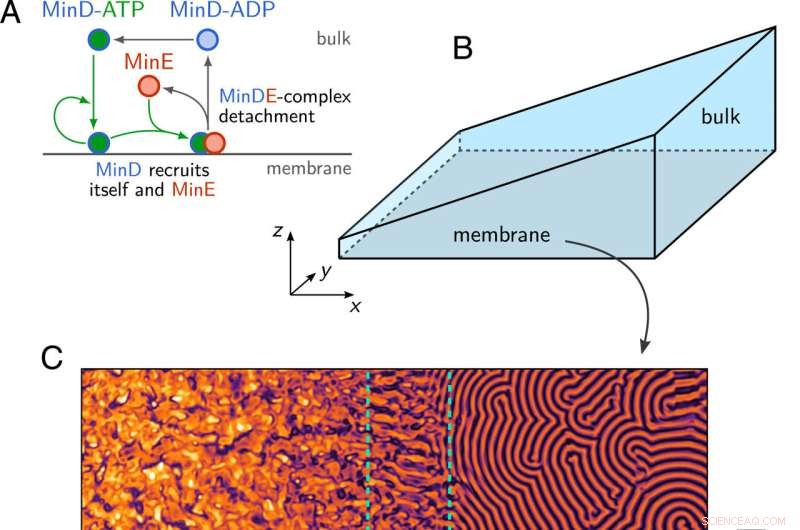
(A) Skematisk illustration af Min-proteinreaktionsnetværket. (B) Kilegeometri med en membranoverflade i bundplanet (z =0) og bulkhøjde H(x) stigende lineært langs x-retningen. (C) Øjebliksbillede af membrandensiteten af MinD, opnået ved numerisk simulering af Min-dynamikkens ligninger. 1–3 i geometrien vist i B. Man observerer områder med kaotiske mønstre, stående bølger (SW, stiplet grøn omrids) og vandrende bølger (TW) langs membranen og i forskellige bulkhøjder. Kredit:Proceedings of the National Academy of Sciences (2022). DOI:10.1073/pnas.2206888119
For mange processer, der er vigtige for livet, såsom celledeling, cellemigration og udvikling af organer, er den rumlige og tidsmæssige korrekte dannelse af biologiske mønstre afgørende. For at forstå disse processer består hovedopgaven ikke i at forklare, hvordan mønstre dannes ud af en homogen starttilstand, men i at forklare, hvordan simple mønstre ændres til stadig mere komplekse. At belyse mekanismerne i denne komplekse selvorganisering på forskellige rumlige og tidsmæssige skalaer er en nøgleudfordring for videnskaben.
Såkaldte "grovkornede" teknikker gør det muligt at forenkle sådanne flerskalasystemer, således at de kan beskrives med en reduceret model i store længde- og tidsskalaer. "Prisen, man betaler for grovkorning, er imidlertid, at vigtig information om mønstrene i små skalaer – som mønstertypen – går tabt. Men sagen er, at disse mønstre spiller en afgørende rolle i biologiske systemer. For at give et eksempel , de styrer vigtige cellulære processer," forklarer Laeschkir Würthner, medlem af teamet ledet af LMU-fysiker Prof. Erwin Frey og hovedforfatter af en ny undersøgelse offentliggjort i Proceedings of the National Academy of Sciences der overvinder dette problem.
Freys team har i samarbejde med forskergruppen af prof. Cees Dekker (TU Delft) udviklet en ny grovkornet tilgang til såkaldte massebevarende reaktionsdiffusionssystemer, hvor den storstilede analyse af de samlede tætheder af de involverede partikler muliggør forudsigelse af mønstre i små skalaer.
Forskerne illustrerede potentialet i deres tilgang med Min-proteinsystemet, en paradigmatisk model for biologisk mønsterdannelse. Bakterien E. coli bruger forskellige Min-proteiner, der cirkulerer i en celle til at bestemme, hvor celledeling finder sted. En afgørende faktor her er, at de involverede proteiner forekommer med forskellige frekvenser afhængigt af deres placering i cellen og kemiske tilstand - hvilket vil sige, at de har en række forskellige tætheder.
"Vi har nu formået at reducere kompleksiteten af dette system ved at udvikle en teori, der udelukkende er baseret på proteinernes samlede tætheder, således at vi fuldstændigt kan spejle dynamikken i mønsterdannelsen," siger Frey. "Dette er en enorm reduktion. De numeriske beregninger er nu udført på få minutter i stedet for måneder."
Forskerne var i stand til eksperimentelt at bekræfte teoretiske forudsigelser af modellen, ifølge hvilken fordelingen af proteinerne afhænger af miljøets geometri. De gjorde dette ved at rekonstruere Min-proteinsystemet i en in vitro-flowcelle, hvor resultaterne viste de samme proteinmønstre, som blev afsløret i simuleringen.
"En sådan rekonstruktion af information i en lille skala fra reduceret dynamik på makroskopisk niveau åbner nye veje for en bedre forståelse af komplekse multiskalasystemer, som forekommer i en bred vifte af fysiske systemer," siger Frey. + Udforsk yderligere
Biologiske mønsterdannende systemer karakteriseret bedre gennem geometri end simuleringer
 Varme artikler
Varme artikler
-
 Sætter et nyt spin på Majorana fermionerTeoretiske beregninger udført af (venstre til højre) Neil Robinson, Robert Konik, Alexei Tsvelik, og Andreas Weichselbaum fra Brookhaven Labs afdeling for kondenseret materie i fysik og materialer vid
Sætter et nyt spin på Majorana fermionerTeoretiske beregninger udført af (venstre til højre) Neil Robinson, Robert Konik, Alexei Tsvelik, og Andreas Weichselbaum fra Brookhaven Labs afdeling for kondenseret materie i fysik og materialer vid -
 Mikroskopi plus AI er lig med hurtig COVID-19-detektion:undersøgelseGabriel Popescu, en professor i elektro- og computerteknik ved University of Illinois Urbana-Champaign, var en del af et team, der kombinerede mærkefri mikroskopisk billeddannelse med kunstig intellig
Mikroskopi plus AI er lig med hurtig COVID-19-detektion:undersøgelseGabriel Popescu, en professor i elektro- og computerteknik ved University of Illinois Urbana-Champaign, var en del af et team, der kombinerede mærkefri mikroskopisk billeddannelse med kunstig intellig -
 Undersøgelse åbner en ny vej til at opnå usynlighed uden brug af metamaterialer(a) Lys med en bølgelængde på 700 nm, der bevæger sig fra bund til top, forvrænges, når cylinderens radius (i midten) er 175 nm. (b) Der er næsten ingen forvrængning, når cylinderen har en radius på 1
Undersøgelse åbner en ny vej til at opnå usynlighed uden brug af metamaterialer(a) Lys med en bølgelængde på 700 nm, der bevæger sig fra bund til top, forvrænges, når cylinderens radius (i midten) er 175 nm. (b) Der er næsten ingen forvrængning, når cylinderen har en radius på 1 -
 Sådan fungerer hologrammerHvis du river et hologram i halve, du kan stadig se hele billedet i hvert stykke. Det samme er tilfældet med mindre og mindre stykker. Hvis du vil se et hologram, du behøver ikke se meget længere end
Sådan fungerer hologrammerHvis du river et hologram i halve, du kan stadig se hele billedet i hvert stykke. Det samme er tilfældet med mindre og mindre stykker. Hvis du vil se et hologram, du behøver ikke se meget længere end
- Forskere udvikler ny røntgendetekteringsteknologi
- Big Brother ansigtsgenkendelse kræver etiske regler
- YouTube for at fremme autoritativt virusindhold på hjemmesiden
- Er folk raske nok til at gå på pension senere?
- I et land med gamle giganter, disse små mærkelige sæler kaldte engang Australiens hjemsted
- Jordens vejrtrækning:bjergerosion en manglende brik i klimapuslespillet


