Fornyet design kunne tage kraftige biologiske computere fra reagensglasset til cellen
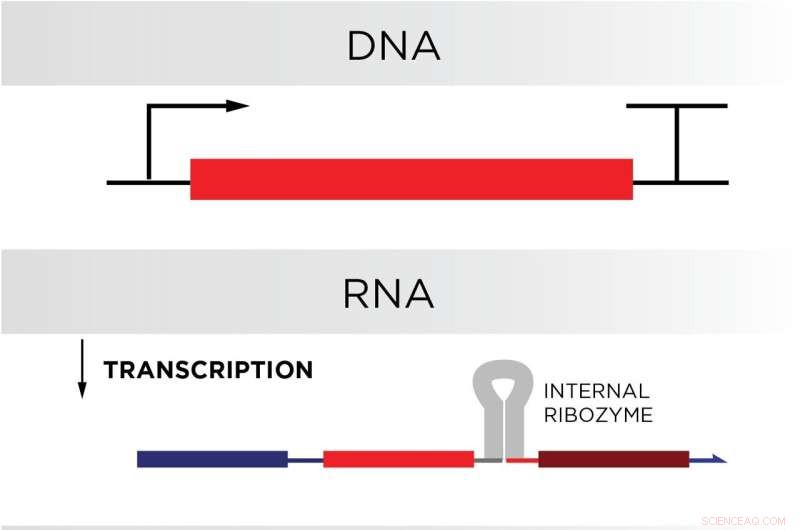
NIST-forskere sigter mod at forvandle cellen til en biologisk computerfabrik ved at designe og indsætte DNA i en celles genom. Celleproteiner ville producere RNA baseret på DNA'et gennem transkription. RNA-strengen ville derefter folde, binde sig til sig selv og dele sig i to takket være en speciel selvspalende RNA-sekvens kaldet et ribozym. Den resulterende struktur, en RNA-kredsløbsport, ville kun fortrydes og udløse yderligere kemiske reaktioner under visse forhold. Kredit:N. Hanacek/NIST
Små biologiske computere lavet af DNA kan revolutionere den måde, vi diagnosticerer og behandler en række sygdomme på, når først teknologien er fuldt udbygget. En stor anstødssten for disse DNA-baserede enheder, som kan fungere i både celler og flydende opløsninger, har dog været, hvor kortlivede de er. Kun én gang, og computerne er brugte.
Nu kan forskere ved National Institute of Standards and Technology (NIST) have udviklet langlivede biologiske computere, der potentielt kan fortsætte inde i celler. I et papir offentliggjort i tidsskriftet Science Advances , giver forfatterne afkald på den traditionelle DNA-baserede tilgang og vælger i stedet at bruge nukleinsyre-RNA'et til at bygge computere. Resultaterne viser, at RNA-kredsløbene er lige så pålidelige og alsidige som deres DNA-baserede modstykker. Hvad mere er, kan levende celler muligvis skabe disse RNA-kredsløb kontinuerligt, noget der ikke er let muligt med DNA-kredsløb, hvilket yderligere positionerer RNA som en lovende kandidat til kraftfulde, langtidsholdbare biologiske computere.
Ligesom den computer eller smartenhed, du sandsynligvis læser dette på, kan biologiske computere programmeres til at udføre forskellige slags opgaver.
"Forskellen er, at i stedet for at kode med enere og nuller, skriver du strenge af A, T, C og G, som er de fire kemiske baser, der udgør DNA," siger Samuel Schaffter, NIST postdoc-forsker og hovedforfatter af undersøgelsen .
Ved at samle en specifik sekvens af baser til en streng af nukleinsyre, kan forskere diktere, hvad den binder sig til. En streng kunne konstrueres til at binde sig til specifikke bits af DNA, RNA eller nogle proteiner forbundet med en sygdom og derefter udløse kemiske reaktioner med andre strenge i det samme kredsløb for at behandle kemisk information og til sidst producere en slags nyttigt output.
Det output kan være et detekterbart signal, der kan hjælpe medicinsk diagnostik, eller det kan være et terapeutisk lægemiddel til at behandle en sygdom.
DNA er dog ikke det mest robuste materiale og kan hurtigt gå fra hinanden under visse forhold. Celler kan være fjendtlige miljøer, da de ofte indeholder proteiner, der hugger nukleinsyrer op. Og selvom DNA-sekvenser hænger længe nok til at detektere deres mål, gør de kemiske bindinger, de danner dem, dem ubrugelige bagefter.
"De kan ikke gøre ting som kontinuerligt at overvåge mønstre i genekspression. De er én anvendelse, hvilket betyder, at de bare giver dig et øjebliksbillede," sagde Schaffter.
Da det også er en nukleinsyre, deler RNA mange af DNA's problemer, når det kommer til at være en biologisk computerbyggesten. Det er modtageligt for hurtig nedbrydning, og efter at en streng binder sig kemisk til et målmolekyle, er den færdig. Men i modsætning til DNA kunne RNA være en vedvarende ressource under de rette forhold. For at udnytte denne fordel skulle Schaffter og hans kolleger først vise, at RNA-kredsløb, som celler teoretisk ville være i stand til at producere, kunne fungere lige så godt som den DNA-baserede slags.
RNA's kant over DNA stammer fra en naturlig cellulær proces kaldet transkription, hvor proteiner producerer RNA på en kontinuerlig basis ved hjælp af en celles DNA som skabelon. Hvis DNA'et i en celles genom kodede for kredsløbskomponenterne i en biologisk computer, så ville cellen producere computerkomponenterne kontinuerligt.
I den biologiske beregningsproces kan enkeltstrenge af nukleinsyrer i et kredsløb nemt ende med at blive bundet til andre strenge i det samme kredsløb, en uønsket effekt, der forhindrer kredsløbskomponenter i at binde sig til deres tilsigtede mål. Designet af disse kredsløb betyder ofte, at forskellige komponenter vil passe naturligt til hinanden.
For at forhindre uønsket binding syntetiseres DNA-sekvenser, der er en del af computere kendt som strengforskydningskredsløb, sædvanligvis (i maskiner i stedet for celler) separat og i en dobbeltstrenget form. Med hver kemisk base på hver streng bundet til en base på den anden, fungerer denne dobbeltstreng som en låst gate, der kun ville låse op, hvis målsekvensen kom og overtog pladsen for en af strengene.
Schaffter og Elizabeth Strychalski, leder af NISTs Cellular Engineering Group og medforfatter af undersøgelsen, forsøgte at efterligne denne "låste port"-funktion i deres RNA-kredsløb, idet de huskede på, at celler i sidste ende selv skulle producere disse låste porte. For at sætte celler op til succes skrev forskerne sekvenserne, så den ene halvdel af strengene kunne binde sig til den anden halvdel. Ved at binde på denne måde ville RNA-sekvenser foldes på sig selv som en pølsebolle, hvilket sikrer, at de er i en låst tilstand.
Men for at fungere korrekt, skal portene være to kemisk bundne, men adskilte tråde, mere som en hamburgerbolle eller sandwich end en hotdogbolle. Holdet opnåede det dobbeltstrengede design i deres porte ved at kode i en strækning af RNA kaldet et ribozym nær foldningspunktet for portene. Dette særlige ribozym - taget fra genomet af en hepatitisvirus - ville skille sig af efter RNA-strengen, den var indlejret i foldet, hvilket ville skabe to separate strenge.
Forfatterne testede, om deres kredsløb kunne udføre grundlæggende logiske operationer, som kun at låse deres porte op under specifikke scenarier, såsom hvis en af to specifikke RNA-sekvenser var til stede, eller kun hvis begge var på samme tid. De byggede og undersøgte også kredsløb lavet af flere porte, der udførte forskellige logiske operationer i serie. Kun når disse kredsløb stødte på den rigtige kombination af sekvenser, ville deres porte låses op en efter en som dominobrikker.
Eksperimenterne involverede udsættelse af forskellige kredsløb for stykker af RNA - hvoraf nogle af kredsløbene var designet til at binde sig til - og måling af output fra kredsløbene. I dette tilfælde var outputtet i slutningen af hvert kredsløb et fluorescerende reportermolekyle, der ville lyse op, når den endelige gate blev låst op.
Forskerne sporede også den hastighed, hvormed portene blev låst op, mens kredsløbene behandlede input og sammenlignede deres målinger med forudsigelserne fra computermodeller.
"For mig skulle disse fungere i et reagensglas lige så prædiktivt som DNA-beregning. Det gode ved DNA-kredsløb er det meste af tiden, man kan bare skrive en sekvens ud på et stykke papir, og det vil fungere på den måde. du vil have," sagde Schaffter. "Nøglen her er, at vi fandt ud af, at RNA-kredsløbene var meget forudsigelige og programmerbare, meget mere end jeg troede, de ville være."
Lighederne i ydeevne mellem DNA- og RNA-kredsløb kunne tyde på, at det kan være fordelagtigt at skifte til sidstnævnte, da RNA kan transskriberes for at genopbygge et kredsløbs komponenter. Og mange eksisterende DNA-kredsløb, som forskere allerede har udviklet til at udføre forskellige opgaver, kunne teoretisk set byttes ud med RNA-versioner og opføre sig på samme måde. For at være sikker er forfatterne af undersøgelsen dog nødt til at skubbe teknologien videre.
I denne undersøgelse demonstrerede forfatterne, at transskriberbare kredsløb fungerer, men de har endnu ikke produceret dem ved hjælp af det rigtige cellulære maskineri af transskription. I stedet syntetiserede maskiner nukleinsyrerne gennem en proces svarende til den, der blev brugt til at producere DNA til forskning. At tage det næste skridt ville kræve indsættelse af DNA i genomet af en organisme, hvor det ville tjene som en plan for RNA-kredsløbskomponenter.
"Vi er interesserede i at sætte disse i bakterier næste gang. Vi vil gerne vide:Kan vi pakke kredsløbsdesign til genetisk materiale ved hjælp af vores strategi? Kan vi få den samme slags ydeevne og adfærd, når kredsløbene er inde i celler?" sagde Schaffter. "Det har vi potentialet til." + Udforsk yderligere
Forskere udvikler nye DNA-logiske kredsløb
 Varme artikler
Varme artikler
-
 Biolog bestrider konklusionerne af nyere artikler om biologisk magnetismeKredit:Caltech Caltech-biolog Markus Meister bestrider nyere forskning og hævder at have løst, hvad han beskriver som det sidste sande mysterium inden for sensorisk biologi - dyrenes evne til at d
Biolog bestrider konklusionerne af nyere artikler om biologisk magnetismeKredit:Caltech Caltech-biolog Markus Meister bestrider nyere forskning og hævder at have løst, hvad han beskriver som det sidste sande mysterium inden for sensorisk biologi - dyrenes evne til at d -
 Undersøgelse sætter nye begrænsninger for mørke fotoner ved hjælp af et nyt dielektrisk optisk …Det mørke fotons mørke stoffelt konverteres til fotoner i et lagdelt dielektrisk mål. Disse fotoner fokuseres af en linse på en lille SNSPD-detektor med lav støj. Strålen, der udsendes fra stakken, er
Undersøgelse sætter nye begrænsninger for mørke fotoner ved hjælp af et nyt dielektrisk optisk …Det mørke fotons mørke stoffelt konverteres til fotoner i et lagdelt dielektrisk mål. Disse fotoner fokuseres af en linse på en lille SNSPD-detektor med lav støj. Strålen, der udsendes fra stakken, er -
 Ny teknologi giver et glimt af solbrændstofproduktion i aktionHoldets elektrokemiske celle til observation af solbrændstofgenererende katalysatorer (gul enhed), opsat ved en røntgenstråle ved Stanford Synchrotron Radiation Lightsource. Kredit:Walter Drisdell/Ber
Ny teknologi giver et glimt af solbrændstofproduktion i aktionHoldets elektrokemiske celle til observation af solbrændstofgenererende katalysatorer (gul enhed), opsat ved en røntgenstråle ved Stanford Synchrotron Radiation Lightsource. Kredit:Walter Drisdell/Ber -
 Registrering af mellem-infrarødt lys, en foton ad gangenKredit:Sean Kelly/NIST I omkring 30 år, videnskabsmænd har brugt superledende materialer til at registrere de mindste lyspletter man kan forestille sig – individuelle fotoner, eller enkelte lyspar
Registrering af mellem-infrarødt lys, en foton ad gangenKredit:Sean Kelly/NIST I omkring 30 år, videnskabsmænd har brugt superledende materialer til at registrere de mindste lyspletter man kan forestille sig – individuelle fotoner, eller enkelte lyspar
- Skoler beder eleverne om at medbringe digitale enheder til klassen, men bliver de faktisk brugt?
- Yellowballs giver ny indsigt i stjernedannelse
- Interaktiv styring til at guide industrirobotter
- Kan granatplaneter være beboelige?
- Undersøgelse afslører en af universets hemmelige ingredienser for livet
- Hvad sker der, når mitose går forkert, og i hvilken fase vil det gå galt?


