Forstå arkitekturen for indre mitokondrielle membranproteiner
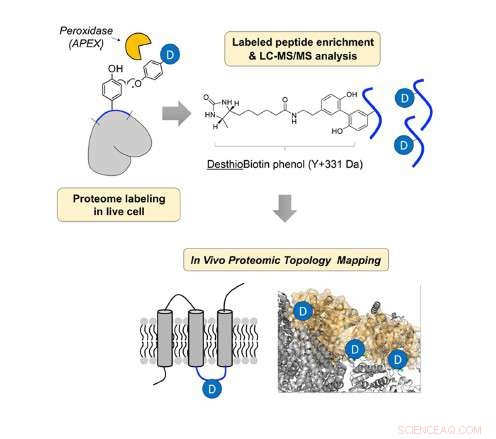
In vivo strukturel identifikation af mitokondrieprotein, ved hjælp af peroxidase og desthiobiotin-phenol. Kredit:UNIST
Membranproteiner udgør cirka en fjerdedel af alle genprodukter og er mål for over 50 procent af alle moderne farmaceutiske lægemidler. Den indre mitokondrielle membran (IMM) proteom spiller en central rolle i opretholdelsen af mitokondriell fysiologi og cellulær metabolisme. På trods af deres betydning, der har ikke været nogen metode til at afsløre topologien for mitokondrielle membranproteiner i levende celler, indtil nu.
En nylig undersøgelse, tilknyttet UNIST har udviklet en ny teknik til at forstå de korrekte arkitekturer af IMM -proteiner, ved hjælp af specielle kemiske værktøjer. Ved at løse den vanskeligste fase af udviklingen af nye lægemidler, deres arbejde vil hjælpe med at fremskynde udviklingen af nye terapier og kure.
Denne forskning er blevet ledet af teamet af professor Hyun-Woo Rhee of Chemistry på UNIST i samarbejde med professor Jong-Seo Kim ved Center fra Center for RNA Research, inden for Institute for Basic Science (IBS) ved Seoul National University og professor Jeong-Kon Seo fra UNIST Central Research Facilities (UCRF). Resultaterne af undersøgelsen er blevet vist i den 15. marts udgave af Journal of the American Chemical Society ( JACS ).
IMM er et af de mest aktive steder for cellulær metabolisme, og det er dybt beslægtet med forskellige menneskelige stofskiftesygdomme, herunder kræft og neurodegenerative sygdomme. Derfor, det er afgørende at forstå den korrekte arkitektur for IMM-proteomet i levende celler for en vellykket og effektiv udvikling af mitokondri-målrettede terapier.
I undersøgelsen, Professor Lee og hans forskerhold afslørede in vivo topologisk retning af 135 IMM -proteiner, ved hjælp af en in situ-genereret desthiobiotin-phenoxylradikalprobe med genetisk målrettet peroxidase (APEX).
"Bestemmelsen af membranproteinstruktur er en af de mest udfordrende opgaver inden for proteinstrukturanalyse, "siger professor Lee." Vores identifikation af strukturelle oplysninger om mitokondrielt indre membranproteom kan give værdifuld indsigt for arkitekturen og forbindelsen af IMM-proteomet i levende celler. "
Forskergruppen designede en ny kemisk sonde, desthiobiotin-phenol og påførte det på IMM-proteiner i levende celler. Derefter, de identificerede strukturen af membranproteiner via massespektrometri (MS).
Peroxidase kan reagere med hydrogenperoxid for at gøre phenoxylradikalet. Derefter, phenoxylradikalen kan reagere med tyrosinresten på det proksimale protein og danne en kovalent binding. I undersøgelsen, forskergruppen opnåede topologioplysningerne ved at analysere det mærkede tyrosinsted for membranproteinet.
Størstedelen af proteinsekvensanalysen anvender i dag massespektrometri (MS), som fordøjer proteinprøven til peptider under anvendelse af et passende enzym. Tidligere analyser, som brugte genetisk målrettet ascorbatperoxidase (APEX), kunne ikke løse strukturel identifikation, fordi disse analyser var baseret på umærket peptiddetektion. Imidlertid, kun det mærkede peptid kan levere nyttig strukturel information, ifølge forskerholdet.
I modsætning til biomolekyler, der er mærket med biotin-phenol, proteiner og andre mål, der er mærket med desthiobiotin-phenol, kan elueres uden hårde, denaturerende forhold. I øvrigt, efterhånden som antallet af tilgængelige membranproteinstrukturprøver opnået via MS stiger, effektiviteten af strukturel identifikation af membranproteiner øges også.
På grund af den korte levetid for phenoxylradikaler, der genereres in situ ved submitokondrielt målrettet APEX og IMM's uigennemtrængelighed for små molekyler, de opløsningsmiddel-eksponerede tyrosinrester af både matrix- og intermembranrums (IMS) sider af IMM-proteiner blev udelukkende mærket med den radikale probe i levende celler af Matrix-APEX og IMS-APEX, henholdsvis og identificeret ved massespektrometri.
Gennem denne analyse, forskergruppen bekræftede 58 IMM -proteintopologier og bestemte den topologiske retning for 77 IMM -proteiner, hvis topologi ved IMM ikke er blevet fuldt ud karakteriseret.
 Varme artikler
Varme artikler
-
 En ny gelelektroforese teknik til hurtig biomarkørdiagnose via massespektrometriOpløselige BAC-tværbundne geler muliggør hurtig og tabsfri genvinding af proteinbiomarkører adskilt af SDS-polyacrylamidgelelektroforese og letter analyse ved massespektrometri. Kredit: Journal of Pro
En ny gelelektroforese teknik til hurtig biomarkørdiagnose via massespektrometriOpløselige BAC-tværbundne geler muliggør hurtig og tabsfri genvinding af proteinbiomarkører adskilt af SDS-polyacrylamidgelelektroforese og letter analyse ved massespektrometri. Kredit: Journal of Pro -
 Forskere opdager nøglemekanismen bag dannelsen af edderkoppesilkeKredit:CC0 Public Domain En gruppe videnskabsmænd ledet af forskere fra RIKEN Center for Sustainable Resource Science (CSRS) har undersøgt den opløselige forløber for edderkoppesilke og fundet ud
Forskere opdager nøglemekanismen bag dannelsen af edderkoppesilkeKredit:CC0 Public Domain En gruppe videnskabsmænd ledet af forskere fra RIKEN Center for Sustainable Resource Science (CSRS) har undersøgt den opløselige forløber for edderkoppesilke og fundet ud -
 Stabil forsyning af elektrokrom metallo-supramolekylær polymerDæmpende EC-glas bestående af en metallo-supramolekylær polymer. Kredit:National Institute for Materials Science National Institute for Materials Science (NIMS) og Tokyo Chemical Industry Co., Ltd
Stabil forsyning af elektrokrom metallo-supramolekylær polymerDæmpende EC-glas bestående af en metallo-supramolekylær polymer. Kredit:National Institute for Materials Science National Institute for Materials Science (NIMS) og Tokyo Chemical Industry Co., Ltd -
 Ny teknik til at karakterisere kemisk sammensætning og struktur af prøverKredit:Forance, Shutterstock Raman -spektroskopi er en væsentlig teknik, der bruges til undersøgelse af materialer - herunder nanostrukturer - og biologiske systemer til at analysere deres sammens
Ny teknik til at karakterisere kemisk sammensætning og struktur af prøverKredit:Forance, Shutterstock Raman -spektroskopi er en væsentlig teknik, der bruges til undersøgelse af materialer - herunder nanostrukturer - og biologiske systemer til at analysere deres sammens
- Mønster af Mozambique-storme uden fortilfælde:FN
- Enkel, billig metode til at beskytte kulfiber
- Forskere bygger en partikelaccelerator, der passer på en chip
- Religiøs tro, der ikke er forbundet med intuition eller rationel tænkning:undersøgelse
- Levende sensor kan potentielt forhindre miljøkatastrofer fra brændstofspild
- NASA ser den tropiske storm Fernanda glide ind i det centrale Stillehav


