At finde ud af molekylernes 3D-form med et tryk på en knap
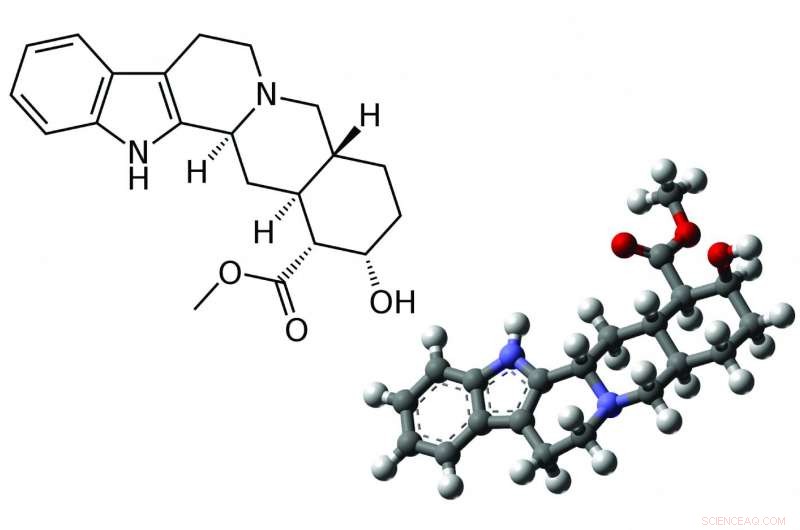
Et computerprogram kan finde ud af 3D-strukturen (til højre) af molekyler som Yohimbine med et tryk på en knap. Molekyler med den samme 2-D struktur (venstre) kan have forskellige 3-D strukturer. Kredit:2D-struktur:acdx (Wikipedia)3D-struktur:MindZiper (Wikipedia)
Et internationalt team af forskere ledet af Carnegie Mellon University kemiker Roberto R. Gil og Universidade Federal de Pernambuco kemiker Armando Navarro-Vázquez har udviklet et program, der automatiserer processen med at finde ud af et molekyles tredimensionelle struktur. Teknikken, beskrevet i et papir i Angewandte Chemie , komprimerer en proces, der normalt tager dage ind i minutter og kan forkorte pipelinen af lægemiddelopdagelse ved at reducere menneskelige fejl.
At finde ud af den kemiske struktur af et molekyle fra bunden er en væsentlig del af forskningen i kemikalier, der kommer fra naturen, eller "naturlige produkter". For stoffer med mulig farmaceutisk anvendelse, denne struktur kan afsløre, hvordan stoffet kan interagere med den menneskelige krop.
"Hvis molekylet skal være et lægemiddel, du skal kende formen på molekylet for at vide, hvordan det vil interagere med en receptor, " siger Gil, en professor i Institut for Kemi ved Carnegie Mellon's Mellon College of Science.
Det første trin i at bestemme et molekyles struktur er at bestemme dets atomare byggesten, efterfulgt af at finde ud af dens todimensionelle struktur, som viser, hvordan hvert atom er forbundet med hinanden. Mens nogle atombindinger er stive, andre kan rotere rundt om et led, gør det muligt for molekyler med de samme komponenter og todimensionelle (2D) strukturer at have forskellige tredimensionelle (3D) former.
Små forskelle i form kan udmønte sig i store ændringer i, hvordan stoffer virker i kroppen. For eksempel, rotation af én binding i det populære smertestillende middel ibuprofen gør det helt inaktivt. Tilsvarende stivelse og cellulose deler den samme 2D-struktur, men har forskellige 3D-former. Den forskel er årsagen til, at mennesker kan fordøje korn og ikke træ.
Gil og Navarro-Vázquez har arbejdet i otte år på at forenkle processen med at finde og sortere gennem de mulige 3D-former for enhver given 2D-struktur. Og med laboratorieværktøjerne til at indsamle data om et molekyles 3D-struktur bliver mere udbredte og tilgængelige, tiden var inde til at udvikle en metode til at automatisere og strømline processen.
Forskerne lavede et program, skrevet i programmeringssproget Python, der gør brug af Residual Dipolar Coupling (RDC) information, et mål for afstanden mellem atomer, der strækker sig fra roterende bindinger. Fodret med data om et givet molekyle fra RDC-eksperimenter, programmet genererer mulige måder, hvorpå molekylet kan eksistere i tre dimensioner, og vælger den mest sandsynlige mulighed.
Teknikken er mest effektiv til at tackle 3D-strukturen af organiske molekyler, der er små til mellemstore, og relativt stiv, med kulstofatomer pakket ind i ringe i stedet for bundet til lange, bøjede kæder. Holdet testede deres program på seks sådanne molekyler, inklusive naltrexon, en medicin, der bruges til at blokere virkningerne af opioider, og stryknin, et pesticid.
Først, de bestemte 2D-strukturen af hvert molekyle ved hjælp af et computerassisteret strukturopklaringsprogram (CASE), hjulpet af et samarbejde med Clemens Anklin, vicepræsident for NMR-applikationer og træning hos Bruker Corporation. De fodrede den information, sammen med RDC-data om molekylet, ind i deres nye program. I hvert tilfælde, deres program var i stand til at udvælge den korrekte 3D-struktur.
"Du trykker på en knap, og med lidt menneskelig eller ingen menneskelig indgriben, du går fra 2D til 3D struktur i ét skud, " siger Gil.
Men lige så vigtigt som programmets hastighed er dets grundighed.
"Mængden af naturlige produkter, der er fejlagtigt rapporteret, hvor den rapporterede struktur ikke svarer til den reelle struktur, er virkelig stor, " siger Navarro-Vázquez, en kemiprofessor ved Universidade Federal de Pernambuco i Brasilien. Dette program kunne være en værdifuld måde at tjekke for mulige strukturer, som forskere ellers kunne gå glip af, hjælpe dem med at undgå deres egne iboende skævheder.
Gil sporer sin vision om denne proces tilbage til sin tid som kandidatstuderende og en samtale med en mentor for 30 år siden om ideen om, at kemikere en dag kunne putte et stof ind i en maskine og se dets struktur med et tryk på en knap. Snart, han håber, programmer som hans og Navarro-Vázquez' skabelse vil integrere de tidligere trin i processen, bringer denne vision endnu tættere på virkeligheden.
"Vi er meget tætte, " siger Gil. "Dette er enhver kemikers drøm."
 Varme artikler
Varme artikler
-
 Ny metode til at kopiere barske forhold for materialerKredit:Chris Bolin, Wikimedia Commons Indeslutning af en plasmastråle kan være stress-inducerende, især på afskærmningsmaterialerne. Bemærk de begrænsninger, der ligger i de testmetoder, der i øje
Ny metode til at kopiere barske forhold for materialerKredit:Chris Bolin, Wikimedia Commons Indeslutning af en plasmastråle kan være stress-inducerende, især på afskærmningsmaterialerne. Bemærk de begrænsninger, der ligger i de testmetoder, der i øje -
 Nye forskningsresultater kan føre til sikrere og mere kraftfulde lithium-ion-batterierKredit:CC0 Public Domain Virginia Commonwealth University-forskere arbejder på at forbedre ledningsevne og sikkerhed i lithium-ion-batterier, som bruges til at drive mange elektroniske enheder run
Nye forskningsresultater kan føre til sikrere og mere kraftfulde lithium-ion-batterierKredit:CC0 Public Domain Virginia Commonwealth University-forskere arbejder på at forbedre ledningsevne og sikkerhed i lithium-ion-batterier, som bruges til at drive mange elektroniske enheder run -
 Bioinspireret syrekatalyseret C2-prenylering af indolderivaterBiomimetisk katalyse er et nyt begreb, der efterligner nøglefunktioner i enzymatisk proces. Prenylering er en allestedsnærværende proces, der findes i næsten alle levende organismer. Inspireret af den
Bioinspireret syrekatalyseret C2-prenylering af indolderivaterBiomimetisk katalyse er et nyt begreb, der efterligner nøglefunktioner i enzymatisk proces. Prenylering er en allestedsnærværende proces, der findes i næsten alle levende organismer. Inspireret af den -
 Konvertering af vedvarende råvarer til platin viser uventet adfærdAcetone (molekyle i midten) reagerer på propanol (venstre) eller propan (højre). De blå atomer i Pt(553) har et højt koordinationstal, de røde atomer i Pt(510) et lavt koordinationstal. Kredit:Leiden
Konvertering af vedvarende råvarer til platin viser uventet adfærdAcetone (molekyle i midten) reagerer på propanol (venstre) eller propan (højre). De blå atomer i Pt(553) har et højt koordinationstal, de røde atomer i Pt(510) et lavt koordinationstal. Kredit:Leiden


