Forbedret proteinanalyse kan hjælpe med tidlig sygdomsdetektion
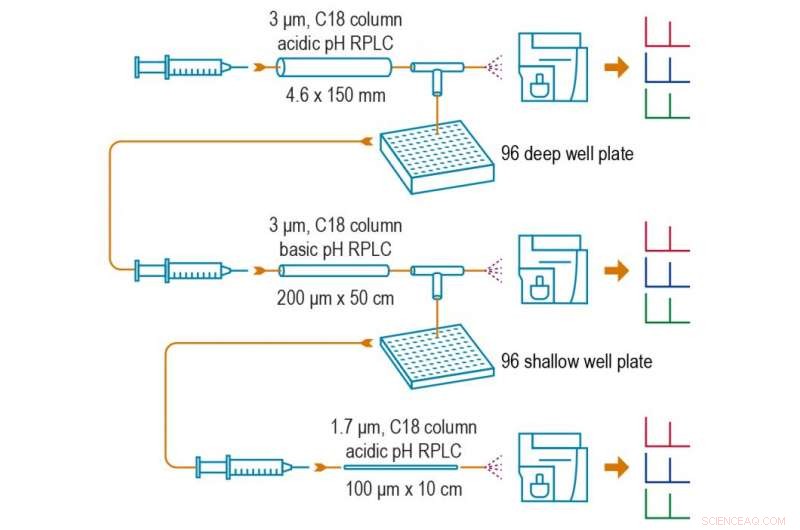
PNNL's dybdyk-SRM-metode tilbyder en ny måde at forberede og analysere blodprøver på, og gør det muligt at påvise proteiner, der findes i meget lave koncentrationer – så lidt som 10 pikogram pr. milliliter af en blodprøve. Metoden har en tre-trins proces:1) Skær den indledende prøve i 96 fraktioner og isoler fraktionen med proteinet af interesse, 2) Adskil netop denne fraktion i yderligere 96 fraktioner og isoler igen fraktionen, der indeholder proteinet af interesse, og 3) Injicer den resulterende berigede prøve i et massespektrometer for at analysere de tilstedeværende proteiner. Kredit:Pacific Northwest National Laboratory
Folk kunne begynde at modtage livreddende behandling for kræft og andre sygdomme meget tidligere ved hjælp af en ny metode til at analysere blodproteiner.
Et papir fra forskere ved Pacific Northwest National Laboratory beskriver en ny måde at forberede og analysere blodprøver på, som markant forbedrer forskernes evne til at påvise proteiner, der findes i meget lave koncentrationer - så lidt som 10 picogram pr. milliliter blod. Sådan lille, svære at opdage koncentrationer er almindelige med sygdomsindikerende proteiner, som også kaldes biomarkører.
Mange forskere identificerer proteiner ved at bruge antistoffer, Y-formede proteiner, der griber ind i proteiner, som de er specifikt designet til at målrette mod. Men mange proteiner har enten ikke matchende antistoffer, eller også er de antistoffer, de har, ikke gode nok til at finde proteiner, når få af dem er til stede.
Forskere har længe brugt antistoffer på denne måde, men massespektrometri giver et attraktivt alternativ, der er mere effektivt til at identificere proteiner. For at analysere et givet protein i blod eller andre biologiske prøver, forskere bruger en massespektrometrisk tilgang kaldet udvalgt reaktionsovervågning, også kendt som SRM. SRM leder specifikt efter unikke peptider, der indikerer tilstedeværelsen af et målrettet protein.
I 2012 PNNL-forskere forbedrede standard SRM, da de udviklede en ny metode kaldet PRISM-SRM, som adskiller en prøve i 96 fraktioner og bestemmer hvilken af disse fraktioner der indeholder målrettede proteiner. Dette gør det muligt for forskere at køre en beriget prøve med mere målrettede proteiner gennem massespektrometeret. Som resultat, den kan påvise proteiner, der findes i koncentrationer mellem 1 nanogram og 100 picogram per milliliter i blodet. 100 picogram-niveauet af følsomhed opstår, når almindelige proteiner, der ikke er relateret til sygdom, fjernes gennem en proces kaldet udtømning, mens følsomhed på 1 nanogram-niveau opstår, når almindelige proteiner ikke er opbrugt.
Men de ønskede stadig at opdage proteiner, der er til stede i endnu mindre mængder. For at køre en prøve med endnu flere af de ønskede proteiner gennem et massespektrometer, et team af PNNL-forskere ledet af Tao Liu skabte en ny proces, der starter med større prøvemængder på forhånd. Efter at have skåret den indledende prøve i 96 fraktioner, forskerne isolerede fraktionen med det målrettede protein svarende til, som de havde gjort med PRISM. Næste, de adskilte netop den fraktion i en anden 96 - og isolerede igen fraktionen, der indeholdt proteinet af interesse.
Kaldet deep-dive SRM, denne nye tilgang gør det muligt at målrette proteiner, der er til stede i den oprindelige blodprøve i koncentrationer så små som 10 pikogram pr. milliliter - og uden behov for antistoffer eller forudgående udtømning. Det øger denne metodes følsomhed med omkring 100, 000 gange sammenlignet med konventionel SRM-analyse og omkring 100 gange sammenlignet med PRISM-SRM.
Deep-dive SRM kunne bruges af forskere til at identificere sygdomsindikerende proteiner i enhver biologisk eller klinisk prøve, før de bliver mere rigelige - herunder potentielt diagnosticering af kræft, før en tumor kan påvises på anden måde. Sådan tidlig opdagelse kan øge chancerne for at redde en patients liv.
 Varme artikler
Varme artikler
-
 Deuteration af molekyler ved hjælp af enzymerKredit:SINE2020 Typiske ruter til fremstilling af per-deutererede molekyler til neutronspredning bruger kemiske metoder, der er uegnede til molekyler med følsomme funktionelle grupper, for eksempe
Deuteration af molekyler ved hjælp af enzymerKredit:SINE2020 Typiske ruter til fremstilling af per-deutererede molekyler til neutronspredning bruger kemiske metoder, der er uegnede til molekyler med følsomme funktionelle grupper, for eksempe -
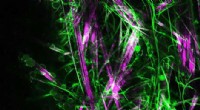 Et nyt geleringsmolekyle til voksende neuroner i 3DCeller indlejret i fibrene i N-heptyl-galactonamid-molekylgelen. Cellerne er runde og grønne. Lige, stive fibre er lyserøde. Buet, fleksible fibre er grønne. Kredit:Anaïs Chalard (IMRCP)-Laurence Vays
Et nyt geleringsmolekyle til voksende neuroner i 3DCeller indlejret i fibrene i N-heptyl-galactonamid-molekylgelen. Cellerne er runde og grønne. Lige, stive fibre er lyserøde. Buet, fleksible fibre er grønne. Kredit:Anaïs Chalard (IMRCP)-Laurence Vays -
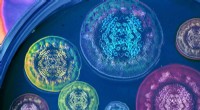 Forskere løser, hvordan svampe producerer forbindelser med potentielle farmaceutiske anvendelserRepræsentation af theof Diels-Alderase kompleks struktur med premalbrancheamid, et af de naturlige produkter, der produceres af de viste svampe. Kredit:Life Sciences Institute multimediedesigner Rajan
Forskere løser, hvordan svampe producerer forbindelser med potentielle farmaceutiske anvendelserRepræsentation af theof Diels-Alderase kompleks struktur med premalbrancheamid, et af de naturlige produkter, der produceres af de viste svampe. Kredit:Life Sciences Institute multimediedesigner Rajan -
 Sådan bukker du fladt glas perfekt rundt om hjørnerEt dobbeltglas hjørneelement produceret med den nye glasbukkeproces. Kredit:Fraunhofer IWM Forskere fra Fraunhofer Institute for Mechanics of Materials IWM har udviklet en ny proces, der kan bøje
Sådan bukker du fladt glas perfekt rundt om hjørnerEt dobbeltglas hjørneelement produceret med den nye glasbukkeproces. Kredit:Fraunhofer IWM Forskere fra Fraunhofer Institute for Mechanics of Materials IWM har udviklet en ny proces, der kan bøje


