Færdiggørelse af lægemiddeldesignstikksaven

Kredit:University of East Anglia
En kraftfuld ny måde at analysere, hvordan lægemidler interagerer med molekyler i kroppen, kan hjælpe med at designe bedre behandlinger med færre bivirkninger.
De fleste lægemidler virker ved at binde sig til et lille sted på de store proteiner, de målretter mod, får proteinet til at ændre form og dermed også dets aktivitet.
For at finde lægemidler, der virker specifikt mod et protein uden også at binde sig til andre, der ligner – og dermed forårsager bivirkninger – er det vigtigt at forstå dette bindingssted i detaljer. Mange nuværende teknikker kan kun give delvise oplysninger, give detaljer om, hvilke dele af selve stoffet der er vigtige og, i nogle tilfælde, proteinets overordnede struktur.
Forskere ved University of East Anglia har nu udviklet en ny tilgang, der kan afsløre den anden side af puslespillet - hvilke dele af proteinet, der interagerer med stoffet. Den tilpasser en teknik kendt som ligand-baseret kernemagnetisk resonans (NMR) spektroskopi for at afsløre, hvilke aminosyrer i proteinet der er involveret i bindingen til lægemidlet.
De var i stand til at gøre dette ved at undersøge stoffet og uden at skulle mærke proteinet, som det kræves i nogle andre metoder.
"At designe nye stoffer er lidt som at finde den rigtige brik, der passer ind i et puslespil, "sagde dr. Jesus Angulo, en lektor ved UEA's School of Pharmacy, der ledede forskningen. "Det er ikke kun formen, men også det grafiske indhold på værket, der skal matche det omgivende billede.
"Vores nye tilgang tillader os nu at finde det nøjagtige stykke, der matcher den komplementære form og det grafiske indhold på et proteinbindingssted."
Den nye NMR-teknik, som kaldes DEEP-STD NMR, er beskrevet i journalen Angewandte Chemie .
Den er baseret på en eksisterende NMR-teknik, der bruges til at studere lægemiddel-protein-interaktioner kaldet STD-NMR. Dette virker ved at spændende alle aminosyrerne i et protein ved at bestråle dem.
Det er derefter muligt at se efter, hvor denne exciterede tilstand overføres til kemiske steder på lægemidlet, når det binder sig til det. Denne fremgangsmåde svarer til at dække proteinet med maling og derefter trykke lægemidlet mod det for at se, hvilke dele der bliver plettet.
Men Dr. Angulo og hans kolleger, hvis arbejde blev finansieret af BBSRC, fandt det muligt at bestråle proteinet med forskellige frekvenser for at ophidse forskellige aminosyrer.
Dette gjorde det muligt for dem at udvælge, hvilke aminosyrer på proteinets bindingssted, der er direkte i kontakt med lægemidlet, fra de "farvemærker", de efterlader.
Det betyder, at de kun skal se på stoffet for at finde ud af de vigtige dele af proteinet, der er målrettet mod. De var i stand til at få yderligere information om de involverede aminosyrer ved at bruge en kombination af deuteriumoxid, eller tungt vand, og normalt vand som opløsningsmiddel.
Holdet, som omfattede forskere ved Quadram Institute i Norwich, demonstreret deres metode på to velstuderede proteiner-et enzym kaldet intramolekylær transsialidase, som produceres af en bakterie, der findes i den menneskelige tarm, og en underenhed af kolera-toksinet.
Dr. Angulo sagde:"Vores nye metode giver forskere et kraftfuldt værktøj til indirekte at forstå arkitekturen af den proteinbindende lomme.
"Dette vil give dem mulighed for at bestemme, hvad der er de bedste kemiske krav til et lægemiddel til at interagere specifikt med en given proteinreceptor. Dette kan føre til stærkere og mere selektive lægemiddelkandidater, mens lavere mængder ville være nødvendige for at udløse den ønskede effekt."
 Varme artikler
Varme artikler
-
 Polymerer springer gennem bøjler på vej til bæredygtige materialerKemisk og biomolekylær ingeniørprofessor Charles Schroeder, venstre, og kandidatstuderende Yuecheng (Peter) Zhou studerer flowdynamikken i ring- og lineære polymerløsninger for at aflæse spor om, hvor
Polymerer springer gennem bøjler på vej til bæredygtige materialerKemisk og biomolekylær ingeniørprofessor Charles Schroeder, venstre, og kandidatstuderende Yuecheng (Peter) Zhou studerer flowdynamikken i ring- og lineære polymerløsninger for at aflæse spor om, hvor -
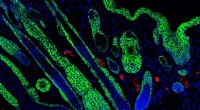 Sårhelende biomaterialer aktiverer immunsystemet for stærkere hudRegenererede hårsække i midten af et sår. Hårsækkene fremstår som dråbestrukturer, og de har Keratin 5 positive tips, som vises med grønt. Kredit:Tatiana Segura Lab, Duke University Forskere ved
Sårhelende biomaterialer aktiverer immunsystemet for stærkere hudRegenererede hårsække i midten af et sår. Hårsækkene fremstår som dråbestrukturer, og de har Keratin 5 positive tips, som vises med grønt. Kredit:Tatiana Segura Lab, Duke University Forskere ved -
 Opfindelsen af gelé kan helbrede sig selv som menneskelig hudDr. Zhen Jiang og lektor Luke Connal. Kredit:ANU Forskere fra The Australian National University (ANU) har opfundet et nyt gelémateriale, der efterligner biologisk materiale såsom hud, ledbånd og
Opfindelsen af gelé kan helbrede sig selv som menneskelig hudDr. Zhen Jiang og lektor Luke Connal. Kredit:ANU Forskere fra The Australian National University (ANU) har opfundet et nyt gelémateriale, der efterligner biologisk materiale såsom hud, ledbånd og -
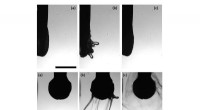 Polymerer forhindrer potentielt farlig tåge under tandlægebesøgBrug af polyakrylsyreopløsning i vand som vandingsvæske i tandplejen reducerer risikoen for aerosolformede patogener. Her, polymeren er vist og danner slangelignende tråde nær spidsen af en vibreren
Polymerer forhindrer potentielt farlig tåge under tandlægebesøgBrug af polyakrylsyreopløsning i vand som vandingsvæske i tandplejen reducerer risikoen for aerosolformede patogener. Her, polymeren er vist og danner slangelignende tråde nær spidsen af en vibreren
- UH-professor tager næste skridt med grafenforskning
- Ny enhed kaster lys over skønheden ved videnskab
- Grækenland beder EU om luftstøtte til at bekæmpe brande
- Sandheden om bioplast
- To neuropeptider i zebrafisk giver spor til de komplekse neurale mekanismer, der ligger til grund fo…
- Første teoretiske bevis:Måling af et enkelt nuklear spin i biologiske prøver


