Måling af molekylære interaktioner
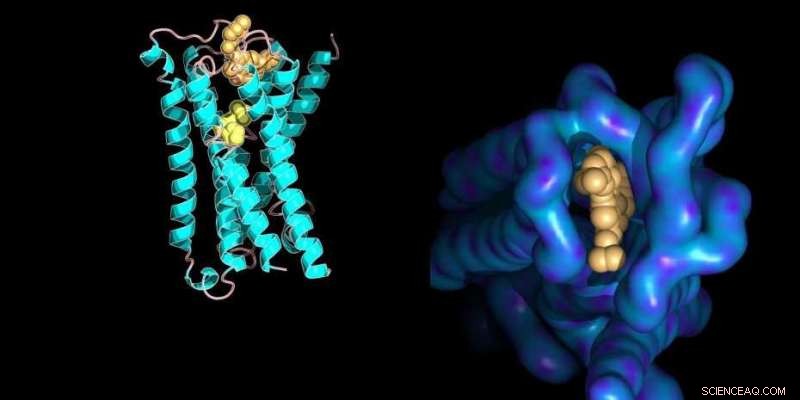
Molekylær model af en proteinmembranreceptor bundet til dens små molekyle ligander med strukturelle detaljer af det allosteriske bindingssted (til højre). Kredit:Ilaria Piazza / ETH Zürich / PDB databaseindgang 4MQT
ETH Zürich-forskere har brugt en ny tilgang til at opdage hidtil ukendte interaktioner mellem proteiner og små metaboliske molekyler i bakterieceller. Teknikken kan også bruges til at teste effekten af medicin.
Videnskaben har nu en ny "omics" - en interaktomik, som omhandler samspillet mellem proteiner og små molekyler. Har tidligere fokuseret på genomik eller proteomik, systembiologer kan nu tilføje protein-metabolit interactomics til blandingen. Genomics beskæftiger sig med den systematiske analyse af alle en organismes gener, mens proteomik beskæftiger sig med hele sættet af proteiner inden for en biologisk enhed.
Med protein-metabolit interaktomik, gruppen ledet af Paola Picotto, professor i Molekylær Systembiologi på ETH Zürich, har nu tilføjet endnu et niveau til den eksisterende "omics". Forskerne har for nylig offentliggjort en undersøgelse i tidsskriftet Cell, hvor de systematisk analyserede og kvantificerede interaktionerne mellem alle proteiner og metabolitter (små metaboliske molekyler) på niveauet af hele proteomet for første gang, etablere relationer mellem dem.
Forskellige skæresteder
Forskerne har vist, hvor mange af de proteiner og enzymer, der findes i en E. coli-bakteriecelle, der interagerer med metabolitter. For at gøre det, de brugte en tilgang kendt som begrænset proteolyse (LiP), kombineret med massespektrometrisk analyse.
Forskerne udtog cellulær væske, som indeholder proteiner, fra bakteriecellerne. De tilføjede derefter en metabolit til hver prøve og tillod den at interagere med proteinerne. Endelig, de skærer proteinerne i mindre stykker (peptider) ved hjælp af "molekylær saks". I alt, forskerne testede 20 forskellige metabolitter og deres interaktioner med proteiner på denne måde.
Når et protein interagerer med en metabolit, om det sætter sig på proteinets aktive sted eller binder sig til et andet sted, proteinstrukturen ændres. Den "molekylære saks" klippede den derefter på forskellige steder fra den oprindelige struktur, resulterer i et andet sæt peptider.
Ved hjælp af massespektrometeret, forskerne målte alle de stykker, der var til stede i prøven, og førte dataene ind i en computer for at rekonstruere de strukturelle forskelle og ændringer, og hvor i proteinet disse er lokaliseret.
Hundredvis af nye interaktioner opdaget
Viden om protein-metabolit-interaktomet (interaktionerne mellem proteiner og metabolitter og de dertil hørende molekylære (signal)netværk), var tidligere meget begrænset sammenlignet med, hvad man vidste om interaktioner mellem forskellige proteiner eller mellem proteiner og DNA eller RNA. Undersøgelsen har nu kraftigt øget denne viden.
Ved at bruge denne tilgang med E. coli, Picotto og hendes team opdagede omkring 1, 650 forskellige protein-metabolit-interaktioner, heraf over 1, 400 var tidligere ukendte. Tusindvis af bindingssteder på proteiner, som metabolitter kan binde sig til, kom også frem. "Selvom metabolismen af E. coli og associerede molekyler allerede er meget velkendt, Det lykkedes os at opdage mange nye interaktioner og de tilsvarende bindingssteder, " siger Picotti. Dette beviser metodens enorme potentiale:"De data, vi producerer med denne teknik, vil hjælpe med at identificere nye reguleringsmekanismer, ukendte enzymer og nye metaboliske reaktioner i cellen."
Strukturændringer regulerer aktivitet
I deres undersøgelse, forskerne viser også, at små stofskiftemolekyler foretrækker at binde sig til (og dermed regulere) proteiner, hvis koncentration er mere eller mindre konstant over tid. Dette tyder på, at bindingen af metabolitter til proteiner og ændringer i proteinkoncentrationer er to komplementære veje, hvormed celler regulerer proteinaktivitet.
Proteiner kan aktiveres eller inaktiveres relativt hurtigt ved en strukturel ændring medieret af metabolitter. "Denne form for strukturændring kan vendes hurtigere, " forklarer Picotto. Fra cellens perspektiv, det giver ofte mening, fordi brug af den vej, der involverer en ændring i koncentrationen, betyder, at cellen skal demontere eller genopbygge proteiner, hvilket koster mere tid, energi og ressourcer.
Picotti og hendes kolleger har også været i stand til at vise, at mange enzymer er mindre kræsne end hidtil antaget:De kan tilsyneladende binde sig til og kemisk ændre flere forskellige metabolitter. Indtil nu, det blev antaget, at enzymer for det meste var specifikke for nogle få meget lignende molekyler.
Medicinalindustrien er meget interesseret i den nye tilgang, da det kan bruges til at teste lægemidlers interaktion med cellulære proteiner og identificere et lægemiddels mål. Forskere kunne undersøge hvilke proteiner og hvilke steder dette lægemiddel binder sig til, hvordan det ændrer deres struktur og dermed påvirker deres aktivitet. Dette ville lette og fremskynde test og udvikling af nye lægemidler.
 Varme artikler
Varme artikler
-
 Cysteinsyntese var et nøgletrin i livets oprindelse:undersøgelseKredit:Unsplash/CC0 Public Domain I et vigtigt skridt under den tidlige udvikling af livet på Jorden, dannelsen af aminosyren cystein leverede vitale katalysatorer, som gjorde det muligt for de
Cysteinsyntese var et nøgletrin i livets oprindelse:undersøgelseKredit:Unsplash/CC0 Public Domain I et vigtigt skridt under den tidlige udvikling af livet på Jorden, dannelsen af aminosyren cystein leverede vitale katalysatorer, som gjorde det muligt for de -
 Team udvikler robust molekylær propel til ensrettede rotationerEt kig på hvordan propellen er sammensat og de ensrettede rotationer. Kredit:Saw-Wai Hla Et team af forskere fra Ohio University, Argonne National Laboratory, Universitié de Toulouse i Frankrig og
Team udvikler robust molekylær propel til ensrettede rotationerEt kig på hvordan propellen er sammensat og de ensrettede rotationer. Kredit:Saw-Wai Hla Et team af forskere fra Ohio University, Argonne National Laboratory, Universitié de Toulouse i Frankrig og -
 Tricker bakterier til hydroxylerende benzenFig.1 Phenolproduktion ved hjælp af bakterier. Kredit:Osami Shoji og Masayuki Karasawa Forskere ved Nagoya University bruger E.coli til at omdanne benzen til phenol, forenkling af en kemisk reakti
Tricker bakterier til hydroxylerende benzenFig.1 Phenolproduktion ved hjælp af bakterier. Kredit:Osami Shoji og Masayuki Karasawa Forskere ved Nagoya University bruger E.coli til at omdanne benzen til phenol, forenkling af en kemisk reakti -
 Lær af fotosyntese:Syntetiske kredsløb kan høste lysenergiSet i gråt, DNA DX-flisen danner et stillads, der muliggør en præcis placering af farvestofmolekyle kromoforer, som samler sig selv på stilladset i karakteristiske J-konfigurationer, ses i grønt. Blå
Lær af fotosyntese:Syntetiske kredsløb kan høste lysenergiSet i gråt, DNA DX-flisen danner et stillads, der muliggør en præcis placering af farvestofmolekyle kromoforer, som samler sig selv på stilladset i karakteristiske J-konfigurationer, ses i grønt. Blå
- Lermineraler kalder skudene med kulstof
- Sådan finder du afstanden mellem to punkter på en cirkel
- Deep-UV-probing-metoden detekterer elektronoverførsel i solcelleanlæg
- Efterforskerne mener, at anti-stall-system aktiveret i Etiopien-nedbrud:WSJ
- Flere børn til højtuddannede:Bredbåndsinternet skaber en digital fertilitetskløft
- Sådan gør du det selv Micro Flying Robot


