Kemikere udvikler motion capture-lignende teknologi til sporing af proteinform
Kredit:University of Pennsylvania
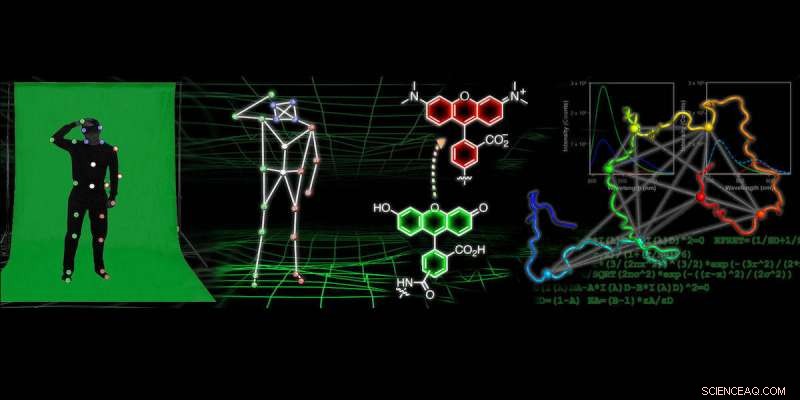
I mange moderne animationsfilm, tricket til at opnå realistiske bevægelser for individuelle karakterer og objekter ligger i motion-capture teknologi. Denne proces involverer ofte, at nogen bærer en træningsdragt dækket af små, farvede kugler, mens et kamera fanger positionen af de farvede kugler, som så bruges til at repræsentere, hvordan personen bevæger sig.
Forskere ved University of Pennsylvania udvikler en lignende teknologi til at opnå "film" med atomopløsning, der sporer, hvordan proteiner folder og ændrer form. For at generere disse film, forskerne mærker proteinet med prober i mange positioner og observerer bevægelsen af disse mærker. Fluorescensdataene om de relative positioner af proberne kan derefter bruges til at konstruere beregningsmodeller af proteinstrukturen i atomare detaljer. Denne forskning kan føre til forbedringer i lægemidler, der bruges til at behandle neurodegenerative sygdomme, samt nye billeddannelsesmetoder, der kan føre til tidligere påvisning.
Forskningen var en tværfaglig indsats ledet af E. James Petersson. Avisen blev udgivet i Biofysisk tidsskrift .
"Et af de store grundlæggende spørgsmål i biokemi er, hvordan proteiner foldes til en bestemt form, sagde Petersson, "og dette er dikteret af sekvensen af aminosyrer i proteinet. Informationen i alle interaktionerne af aminosyresidekæderne fører på en eller anden måde til, at den foldes til en ordentlig form."
I sunde scenarier, Petersson sagde, at korrekt form tillader proteinet at have forskellige funktioner, som at transportere ilt i blodet eller blive "molekylære maskiner", der i sidste ende fører til muskelbevægelser, som dem, der kræves til at gå og løbe.
Men i visse sygdomstilstande, især ved neurodegenerative sygdomme som Alzheimers og Parkinsons, proteinerne folder forkert til en usund form, hvilket kan få flere kopier af proteinerne til at aggregere til "spaghetti-lignende sammenfiltringer eller lange fibriller." Disse fibriller, Petersson sagde, er giftige for neuroner, som ligger til grund for Alzheimers og Parkinsons sygdom.
"Da alle disse involverer proteinfoldning til en bestemt form, så det, vi gerne vil gøre, er at spore ændringerne i form af proteiner, " sagde han. "Der er en række forskellige teknikker, der kan bruges til at gøre dette, men vi kan godt lide fluorescens, fordi du kan opnå fluorescensdata hurtigt nok til, at du faktisk kan se proteiner folde sig i realtid. I sidste ende vil vi gerne prøve at se proteiner folde sig i celler."
For at få information om proteinform ved hjælp af fluorescerende prober, forskere bruger en teknik kaldet fluorescensresonans energioverførsel, hvilket kræver, at de måler mange afstande mellem forskellige punkter på proteinet og derefter bruger denne information til at forstå dets form, svarende til motion-capture-teknologi.
I denne avis, forskerne lavede omkring 30 målinger af forskellige afstande inden for proteinet alfa-synuclein under forskellige tilstande, hvor det ændrer form. De brugte derefter denne samling af afstandsmålinger i kombination med kompleks beregningsmodellering for at få atomare opløsningsstrukturer af proteinets form.
Ferrie modtog et stipendium fra Parkinsons Disease Foundation for at tilbringe sommeren i David Bakers laboratorium på University of Washington, hvor Rosetta, et af de mest brugte programmer til modellering af proteiner, blev udviklet.
"Rosetta er designet til at modellere stabile velfoldede proteiner, " sagde Petersson, "ikke forstyrrede proteiner, der kan ændre form, så Jack måtte selv lave en masse omskrivning af koden for at kunne modellere disse uregerlige proteiner."
Ferrie og Yoon lavede en række eksperimentelle målinger, der kunne bruges til at styre proteinfoldningen, så den stemmer overens med de eksperimentelle målinger, hvilket gjorde det muligt for ham at modellere proteinform.
Da det var en ny tilgang, forskerne ønskede at demonstrere, at de strukturer, der kommer ud af beregningsmodellerne, var i overensstemmelse med virkeligheden. For at gøre dette, de udførte tre typer eksperimenter for at matche virkelige data med de modeller, der kom ud af disse fluorescenseksperimenter.
De samarbejdede med Rhoades' gruppe for at validere de modellerede strukturer ved hjælp af enkeltmolekyle fluorescensmålinger foretaget af Pan. Arbejder i Fakhraiis gruppe, Lin, brugt en anden type teknik kaldet atomkraftmikroskopi til at afbilde proteinet og validere modellerne. Chris Dobson, professor i kemi ved Cambridge University, delte kernemagnetiske resonansdata om alfa-synuclein, der gav yderligere bekræftelse af computermodellernes nøjagtighed.
Artiklen repræsenterer et af de største biblioteker af proteiner mærket med syntetiske fluoroforer, der endnu er rapporteret. Ifølge Petersson, forskerne skulle lave en masse afstandsmålinger over forskellige regioner af proteinet for at have nok data til at generere beregningsmodeller. Ferrie og Haney var nødt til at finde på en strømlinet tilgang til at fastgøre forskellige sæt af sonder, der ville fungere over forskellige afstandsområder.
Forskerne arbejder nu på at anvende denne teknik til at modellere proteinstruktur i de aggregerede former, der er toksiske for neuroner, og til at modellere dens reaktion på lægemidler, der ville få det til at ændre form, forhindre denne sammenlægning.
"Evnen til at se et protein, når det ændrer form, sagde Petersson, "og faktisk at få strukturer ud af det er et virkelig vigtigt grundlæggende videnskabeligt mål, som vi har arbejdet hen imod i 10 år. Der har været nogle meget imponerende gennembrud i at få strukturer af proteiner i neurodegenerative sygdomme, men fluorescensteknikken har potentialet til at gøre det i levende celler, som ingen anden teknik har evnen til at gøre."
Ifølge Petersson, proteiner, der antager flere former, stable forskellige kopier oven på hinanden og aggregere, som de gør ved neurodegenerative sygdomme, er et vigtigt grundlæggende biokemiproblem, som andre strukturbiologiske teknikker ikke rigtig kan tackle. En bedre forståelse af, hvordan det virker, og hvad disse former er, har potentialet til at påvirke sygdomme som Parkinsons og give forskere mulighed for at finde ud af, hvordan lægemiddel- eller diagnostiske modeller interagerer med proteinet.
"We're working on being able to generate model structures that actually show what is the effect of these drugs, " Petersson said. "We take the protein with the fluorescent labels, add the drug, allow the protein to change shape, make fluorescence measurements and then take those back to the computational modeling so we can actually see the structural effect of these drugs. Hopefully this will lead to more of a rational understanding so that better second and third generation drugs can be made."
The researchers are also collaborating with Robert Mach, the Britton Chance Professor of Radiology in Penn's Perelman School of Medicine. Mach's group is interested in developing positron emission tomography imaging probes that can be used to bind to the aggregated forms of proteins and image them in patients.
"There are some promising drugs for treating neurodegenerative diseases such as Alzheimer's and Parkinson's, that could block this formation of aggregates, " Petersson said, "but the problem is that, by the time people show cognitive or motor-tremor symptoms, it's too late to use these drugs because there's already too much neurodegeneration. If you're getting aggregates in your brain, even if you're not showing any behavioral changes or learning deficits, these probes could noninvasively image the aggregates. By achieving a rational understanding of what the protein structure is, we hope we can help with that work moving forward."
 Varme artikler
Varme artikler
-
 Dominerende svamp kunne være løsningen til at producere flere biobrændstoffer og kemikalierEt eksempel på en mulig ligninstruktur. Kredit:GFDL ver. 1.2 eller CC-by-sa ver. 2,5, 2.0, og 1.0 lub Public Domain (PD) Opdagelsen af et nyt enzym, der frigiver et værdifuldt kemikalie fra land
Dominerende svamp kunne være løsningen til at producere flere biobrændstoffer og kemikalierEt eksempel på en mulig ligninstruktur. Kredit:GFDL ver. 1.2 eller CC-by-sa ver. 2,5, 2.0, og 1.0 lub Public Domain (PD) Opdagelsen af et nyt enzym, der frigiver et værdifuldt kemikalie fra land -
 Fremtid for bærbar elektronik:Ny organisk halvleder med spændende egenskaberKredit:CC0 Public Domain Halvledere er stoffer, der har en ledningsevne mellem ledernes og isolatorernes. På grund af deres unikke egenskaber ved at lede strøm kun under særlige forhold, de kan ko
Fremtid for bærbar elektronik:Ny organisk halvleder med spændende egenskaberKredit:CC0 Public Domain Halvledere er stoffer, der har en ledningsevne mellem ledernes og isolatorernes. På grund af deres unikke egenskaber ved at lede strøm kun under særlige forhold, de kan ko -
 Nye sensorer kan muliggøre en mere overkommelig påvisning af forurening og sygdommeKredit:American Chemical Society Når det kommer til at teste for kræft, miljøforurening og fødevareforurenende stoffer, traditionelle sensorer kan hjælpe. Udfordringerne er, at de ofte er omfangsr
Nye sensorer kan muliggøre en mere overkommelig påvisning af forurening og sygdommeKredit:American Chemical Society Når det kommer til at teste for kræft, miljøforurening og fødevareforurenende stoffer, traditionelle sensorer kan hjælpe. Udfordringerne er, at de ofte er omfangsr -
 Et konkret skridt mod netto-nul kulstofemissioner i cementproduktionKredit:ElRoi, Shutterstock Som det mest udbredte byggemateriale, beton er uundværligt i udviklingen af infrastruktur, industri og boliger. Det er en blanding af tilslag (sand, grus, knust sten),
Et konkret skridt mod netto-nul kulstofemissioner i cementproduktionKredit:ElRoi, Shutterstock Som det mest udbredte byggemateriale, beton er uundværligt i udviklingen af infrastruktur, industri og boliger. Det er en blanding af tilslag (sand, grus, knust sten),
- Solaktivitet rekonstrueret i løbet af et årtusinde
- Forskel mellem transkription og DNA-replikation
- Hvorfor skysåning i stigende grad er attraktiv for det tørstige Vesten
- GPM satellit finder nedbør skubbet væk fra Tropical Cyclone Cebiles center
- Ligesom Star Trek:stemmeshopping set som ny grænse
- Instrumenter bruges til at studere stjerner


