Sådan kodes en funktionel molekylær maskine
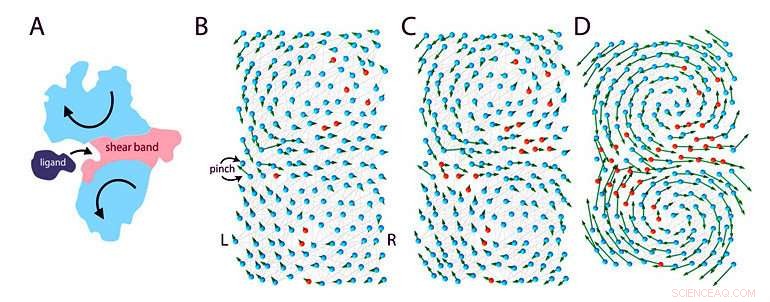
Figur 1:Elastisk model af et protein, der binder til en ligand. (A) Når et protein binder til en ligand, det gennemgår store bevægelser (pile), som er signaturerne for bøjede funktionelle proteiner. Dette er kun muligt takket være tilstedeværelsen af visse "floppy" regioner (pink "shear band") på tværs af proteinet, der adskiller de stive (blå) regioner af proteinet i to domæner. (B)-(D) Holdet modellerede et protein på 200 aminosyrer under forskellige udviklingsstadier:overgang fra en ikke-funktionel (B) til en funktionel (D) tilstand. Proteinet er modelleret som et elastisk fjedernetværk med to slags aminosyrer, modelleret som perler:lyserøde aminosyrer er fleksible og blå aminosyrer er stive. Forskerne efterligner evolutionen ved at ændre én tilfældig aminosyre ad gangen (mutation) fra pink til blå. I første omgang, proteinet er for det meste stift og ikke-funktionelt. Under evolutionen, fleksible aminosyrer tilsættes, nogle nyttige, nogle ikke. Over tid, en "floppy" region dannes i midten af molekylet, hvilket gør proteinet mere fleksibelt til at bøje og binde til liganden. Modellen vurderede, at en effektiv løsning opnås efter tusinde mutationer. Kredit:Institut for Grundvidenskab
Et internationalt team har udviklet en model, der simulerer proteinudvikling. Starter fra stiv, ufunktionelle proteiner, computermodellen viser, hvordan udviklende proteinkomponenter kan arbejde sammen for at skabe dynamiske og effektive molekylære maskiner. Fleksibilitet gør det muligt for proteiner at ændre deres 3D-konformation for at binde andre molekyler:denne egenskab er afgørende for deres funktion. Prof. Tsvi Tlusty og Dr. Sandipan Dutta ved Center for Soft and Living Matter, inden for Institut for Grundvidenskab (IBS, Sydkorea), i samarbejde med prof. Albert Libchaber fra Rockefeller University og prof. Jean-Pierre Eckmann fra University of Geneva har efterlignet genudvikling for at opnå proteiner, der kan bøje og binde sig til andre molekyler. Forståelse af dette forhold er et af de mest efterspurgte aspekter af proteinbiologi; det kunne hjælpe med at forklare den farmaceutiske virkning af lægemidler, der binder til deres mål.
Evolutionen har formet den levende verden, vi ser omkring os, i milliarder af år. Millioner af proteiner arbejder harmonisk for at holde disse livsprocesser i gang. De er ansvarlige for den gnidningsløse funktion af enhver organisme:de genkender andre molekyler (ligander), binde sig til dem og omvende dem. Andre har transportfunktion, give struktur, og støtte til cellerne. Gener gemmer informationen om produktion og design af disse molekylære maskiner. Imidlertid, trods årtiers forskning, det er ikke trivielt at tegne "kortet", der tegner vejen fra gener til proteinfunktion.
Ifølge en nylig hypotese, proteinfunktion er afhængig af "fleksible led." Dette studie, udgivet i Proceedings of the National Academy of Sciences ( PNAS ), undersøger sammenhængen mellem funktion og fleksibilitet ved at modellere proteiner som elastiske netværk. I denne model, proteiner er lavet af fleksible (polære) og stive (hydrofobe) aminosyrer forbundet med molekylære "fjedre". Hvis nogle områder af proteinet er fleksible nok, de danner en "floppy" kanal, og hele den molekylære maskine kan bøjes som et hængsel. Denne bevægelse giver dem mulighed for at binde sig effektivt til andre molekyler. Bindingen mellem en ligand og et stift eller fleksibelt protein kan opfattes som en bold, der lander på en sten eller en blød pude. Bolden hopper sandsynligvis væk efter at have ramt klippen, men puden er mere tilbøjelig til at acceptere det. Derfor, det fleksible protein er et bedre bindemiddel.
I denne model, gener lagrer detaljerne i proteindesignet på en binær måde:fleksible aminosyrer lagres som nuller og stive aminosyrer som enere. Som resultat, hele proteinstrukturen kan forenkles som en kode, gerne 11110001...111, ligner den digitale hukommelse på en computer. Imidlertid, ikke alle koder giver anledning til funktionelle proteiner, for eksempel en kode med kun én:111111…1111, ville give anledning til et helt stift protein, ude af stand til at bevæge sig, og ikke-funktionelle. Blandt alle mulige koder, kun nogle producerer et funktionelt protein med en "floppy" region i midten, der kan byde liganden velkommen.
Modellen efterligner evolution ved at ændre en tilfældig aminosyre ad gangen. Under evolutionen, nuller og etaller i genet bladres tilfældigt gennem en proces kaldet mutation. De fleste mutationer giver ingen forskel, eller føre til ikke-funktionelle proteiner, men nogle sjældne mutationer kan give anledning til et mere effektivt protein. I det væsentlige, både funktionelle og ikke-funktionelle proteiner produceres under evolution, men ifølge Darwins teori om "survival of the fittest", kun de funktionelle proteiner bevares, og de ikke-funktionelle proteiner dør til sidst ud.
Hvordan ser en "funktionel" kode ud? Svaret er ikke ligetil. Faktisk, antallet af koder for et funktionelt protein, selv et simpelt protein, er enorm, større end universets størrelse. Imidlertid, ved hjælp af teknikker til dataanalyse, det er muligt at søge efter skjulte mønstre i alle funktionelle koder for at lede efter nogle samlende egenskaber. For eksempel, den "floppy" kanal i proteinet har interessante og ejendommelige træk, og en mutation i den ene ende af kanalen har langtrækkende virkninger, som kraftigt kan påvirke opretholdelsen af mutationer af andre fjerne aminosyrer.
"I fremtiden, vi planlægger at undersøge, hvordan man anvender denne undersøgelse på rigtige proteiner, som kinaser, " sagde gruppeleder Tsvi Tlusty, en korrespondent i undersøgelsen. "I øvrigt, undersøgelsen åbner muligheder for at undersøge udviklingen af andre proteinfunktioner, som molekylær genkendelse. Ved hjælp af store databaser, som er udviklet gennem mange års forskning, kan sandsynligvis afdække nogle underliggende fænomener om udviklingen af proteiner."
 Varme artikler
Varme artikler
-
 Bærbar elektronik til kontinuerlig hjerte, respirationsovervågningEn lille og billig sensor, annonceret i Applied Physics Letters og baseret på et elektrokemisk system, kan potentielt bæres uafbrudt af hjertepatienter eller andre, der kræver konstant overvågning. Hø
Bærbar elektronik til kontinuerlig hjerte, respirationsovervågningEn lille og billig sensor, annonceret i Applied Physics Letters og baseret på et elektrokemisk system, kan potentielt bæres uafbrudt af hjertepatienter eller andre, der kræver konstant overvågning. Hø -
 Maling med halvlederePerovskite portræt af Marie Skłodowska-Curie. Kredit:Lukas Helmbrecht AMOLF-forskerne Lukas Helmbrecht og Wim Noorduin har udviklet en reaktiv blæk, der kan males på et lige så reaktivt lærred. Bl
Maling med halvlederePerovskite portræt af Marie Skłodowska-Curie. Kredit:Lukas Helmbrecht AMOLF-forskerne Lukas Helmbrecht og Wim Noorduin har udviklet en reaktiv blæk, der kan males på et lige så reaktivt lærred. Bl -
 Direkte konvertering af rustent rustfrit stålnet til stabilt, billige elektroder til kalium-ion-bat…Kinesiske videnskabsmænd har gjort god brug af affald, mens de har fundet en innovativ løsning på et teknisk problem ved at omdanne rustent rustfrit stålnet til elektroder med fremragende elektrokemis
Direkte konvertering af rustent rustfrit stålnet til stabilt, billige elektroder til kalium-ion-bat…Kinesiske videnskabsmænd har gjort god brug af affald, mens de har fundet en innovativ løsning på et teknisk problem ved at omdanne rustent rustfrit stålnet til elektroder med fremragende elektrokemis -
 Sølv forbedrer effektiviteten af solceller i monokornlagNæste generations letvægts, fleksible monograin-lagsolcelle udviklet af TalTech-forskere. Kredit:Professor Jüri Krustok Som et resultat af deres toårige fælles projekt, materialeforskerne fra Tall
Sølv forbedrer effektiviteten af solceller i monokornlagNæste generations letvægts, fleksible monograin-lagsolcelle udviklet af TalTech-forskere. Kredit:Professor Jüri Krustok Som et resultat af deres toårige fælles projekt, materialeforskerne fra Tall
- Ikke-magnetisk skalbelægning af magnetiske nanopartikler som nøglefaktor for cytotoksicitet
- En nanostruktur, der stimulerer vækst af stamceller til behandling af Parkinsons sygdom
- Flere Basra-vandkriser, medmindre den irakiske regering løser fejl:HRW
- EU går fremad med hensyn til retfærdig overgang, men risikerer at snuble på fossile brændstoffer
- Fornyet model for besætningsplanlægning reducerer flyselskabets forsinkelser med så meget som 30 …
- Hvorfor er vand vigtigt for levende organismer?


