Fluorescensaktiverende beta-tøndeprotein lavet fra bunden for første gang
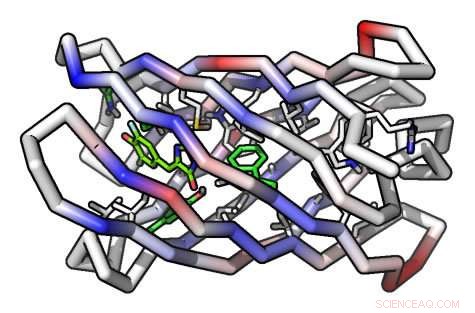
En computermodel af et beta-tønde-proteinmolekyle Kredit:Institute for Protein Design/UW Medicine
For første gang, videnskabsmænd har skabt, helt fra bunden, et protein, der er i stand til at binde til et lille målmolekyle. Forskere fra University of Washington School of Medicine rapporterer fremskridt i tidsskriftet 12. september i tidsskriftet Natur .
Tidligere har sådanne små molekyle bindende proteiner er blevet lavet ved at ændre proteiner, der allerede eksisterer i naturen. Den tilgang begrænsede mulighederne markant. Evnen til at lave sådanne proteiner fra bunden, eller "de novo, " åbner vejen for videnskabsmænd til at skabe proteiner, der ikke ligner nogen, der findes i naturen. Disse proteiner kan specialdesignes med høj præcision og affinitet til at binde til og virke på specifikke små molekyle-mål.
Papirets hovedforfattere er Jiayi Dou og Anastassia A. Vorobieva, begge seniorstipendiater i seniorforfatteren David Bakers laboratorium, professor i biokemi ved UW School of Medicine og direktør for Institute of Protein Design på UW Medicine. Baker er også efterforsker ved Howard Hughes Medical Institute.
Teknikken bør have bred anvendelse inden for forskning, medicin og industri, ifølge Baker.
"Det vellykkede de novo-design af specialbyggede proteiner med småmolekylebindingsaktivitet sætter scenen for skabelsen af stadigt mere sofistikerede bindingsproteiner, som ikke vil have de begrænsninger, der ses med proteiner, der er designet ved at ændre eksisterende proteinstrukturer, " forklarede han.
For at lave proteinet, forskerne skulle opnå en anden første:At skabe fra bunden af et cylinderformet protein kaldet en beta-tønde. Betatønderstrukturen var ideel, fordi den ene ende af cylinderen kunne designes til at stabilisere proteinet, mens den anden ende kunne bruges til at skabe et hulrum, der kan tjene som bindingssted for målmolekylet.
Proteiner er lavet af lange kæder af aminosyrer. Når først det er syntetiseret, disse kæder foldes til præcise former, der gør det muligt for proteinerne at udføre deres funktioner. Formerne, som disse kæder antager, er typisk utroligt krøllede, men to regelmæssige træk forekommer ofte:alfa-helixer, som dannes, når sektionernes kæde snor sig om en central akse, og arklignende strukturer, kaldet beta-ark.
Beta-sheets dannes, når to eller flere sektioner fra forskellige dele af aminosyrekæden, på grund af foldning, køre side om side i 3D-rum. Disse sektioner er "syet sammen" af hydrogenbindinger, skabe en arklignende struktur. Disse beta-ark, på tur, kan samles til tøndelignende strukturer, kaldet beta-tønder. I naturen, beta-tønder proteiner binder en lang række små molekyler.
For at designe det nye protein, Dou og Vorobieva brugte en softwareplatform, udviklet i Baker lab, kaldet Rosetta. Det kan forudsige, hvilken form en bestemt kæde af aminosyrer vil antage efter syntese og kan fortælle, hvordan ændring af individuelle aminosyrer langs kæden kan ændre denne form. Denne forudsigelsesevne gør det muligt at teste forskellige kombinationer af aminosyrer for at designe et protein med den ønskede form og funktion.
For at skabe hulrummet, forskerne brugte en kraftfuld ny docking-algoritme, kaldet "Rotamer Interaction Field" (RIF), udviklet af William Sheffler, en seniorforsker i Baker-laboratoriet. RIF identificerer hurtigt alle potentielle strukturer af hulrum, der opfylder forudsætningerne for at binde specifikke molekyler.
Udstyret med de nye RIF-dockingmetoder, Gør du, Vorobieva og Sheffler designede beta-tønderne til at binde en forbindelse kaldet DFHBI, en komponent, der ligner det, der er indeholdt i grønt fluorescerende protein, som fluorescerer, når de udsættes for visse lysfrekvenser. Grønt fluorescerende protein bruges rutinemæssigt i biologisk forskning til at lokalisere molekyler og strukturer i levende organismer og spore deres bevægelse.

Anastassia A. Vorobieva. holder sin nye søn, med sin forskerkollega Jiayi Dou. De to videnskabsmænd ledede designet og testningen af et beta-tøndeprotein, der aktiverer fluorescens. Det nye protein, bygget fra bunden, er et fremskridt inden for specialdesignede proteiner til præcist at målrette mod små molekyler. Kredit:Institute for Protein Design/UW Medicine
I deres papir, forskeren demonstrerede, at deres specialdesignede protein ivrig bandt og aktiverede DFHBI-forbindelsen.
"Det virkede i bakterielle, gær- og pattedyrceller, "sagde Dou, "og at være halvt så stor som grønt fluorescerende protein burde være meget nyttigt for forskere."
Baker sagde, at tilgangen vil give forskere mulighed for at udforske et effektivt ubegrænset sæt af rygradsstrukturer med former, der er tilpasset til at binde molekylet af interesse.
"Lige vigtigt, " han tilføjede, "det fremmer i høj grad vores forståelse af determinanterne for proteinfoldning og -binding ud over, hvad vi har lært af at beskrive eksisterende proteinstrukturer."
 Varme artikler
Varme artikler
-
 Forskere identificerer skadelige bakterier baseret på dets DNA til en meget lav prisMinION-enhed (højre) sekventering af bakterielle genomiske fragmenter efter ON-rep-seq-metoden. Her er enheden koblet til en MinIT til dataindsamling (til venstre) og styres af en smart telefon. Kredi
Forskere identificerer skadelige bakterier baseret på dets DNA til en meget lav prisMinION-enhed (højre) sekventering af bakterielle genomiske fragmenter efter ON-rep-seq-metoden. Her er enheden koblet til en MinIT til dataindsamling (til venstre) og styres af en smart telefon. Kredi -
 Fremskridt inden for cellulosebaseret fødevareemballage går videre til testfasen af industriel p…Kredit:Pixabay/CC0 Public Domain VTT tester Thermocell -plastfilm fremstillet af cellulose og fedtsyrer i produktionen af mademballage i samarbejde med Arla Foods, Paulig, og Wipak. Termocelle p
Fremskridt inden for cellulosebaseret fødevareemballage går videre til testfasen af industriel p…Kredit:Pixabay/CC0 Public Domain VTT tester Thermocell -plastfilm fremstillet af cellulose og fedtsyrer i produktionen af mademballage i samarbejde med Arla Foods, Paulig, og Wipak. Termocelle p -
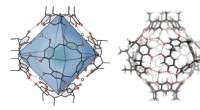 Ny tilgang til terpensynteserTil venstre, det omkring 1,4 kubik nanometer store hulrum i den molekylære kapsel er fremhævet med blåt. Til højre, samhørigheden af kapslen via hydrogenbindinger (grønne stiplede linjer) er synlig.
Ny tilgang til terpensynteserTil venstre, det omkring 1,4 kubik nanometer store hulrum i den molekylære kapsel er fremhævet med blåt. Til højre, samhørigheden af kapslen via hydrogenbindinger (grønne stiplede linjer) er synlig. -
 Udfordringer i udviklingen af elektrokatalysatorerKredit:Pixabay/CC0 Public Domain Regenerativ energihøst genererer ofte mere elektricitet, end der er behov for direkte. Elektrokemiske processer kunne bruges til at lagre den overskydende energi e
Udfordringer i udviklingen af elektrokatalysatorerKredit:Pixabay/CC0 Public Domain Regenerativ energihøst genererer ofte mere elektricitet, end der er behov for direkte. Elektrokemiske processer kunne bruges til at lagre den overskydende energi e
- Nysen og hoste fungerer som mini atombomber og overstiger regelmæssigt to meter
- Ny magnet uden manglerne ved konventionelle samarium- og neodymmagneter
- Amerikanske mål giver afkald på at lade Californien styre emissionsgrænser
- Lektioner fra Aliso Canyon lækagen
- NASA skovstrukturmission frigiver første data
- Plasmamembran: Definition, struktur og funktion (med diagram)


