Supercomputere hjælper med at samle proteiner

Ved hjælp af supercomputere, forskere er lige begyndt at designe proteiner, der samler sig for at kombinere og ligne livgivende molekyler som hæmoglobin. Kredit:Taylor et al.
Røde blodlegemer er fantastiske. De optager ilt fra vores lunger og bærer det over hele vores krop for at holde os i live. Hæmoglobinmolekylet i røde blodlegemer transporterer ilt ved at ændre sin form på en alt-eller-intet måde. Fire kopier af det samme protein i hæmoglobin åbner og lukker som blomsterblade, strukturelt koblet til at reagere på hinanden. Ved hjælp af supercomputere, forskere er lige begyndt at designe proteiner, der samler sig for at kombinere og ligne livgivende molekyler som hæmoglobin. Forskerne siger, at deres metoder kan anvendes på nyttige teknologier såsom farmaceutisk målretning, høst af kunstig energi, 'smarte' sanse- og byggematerialer, og mere.
Et videnskabsteam udførte dette arbejde ved at overlade proteiner, hvilket betyder, at de ændrede underenhederne af proteiner, aminosyrerne, at give proteinerne en kunstigt høj positiv eller negativ ladning. Ved hjælp af proteiner fra vandmænd, forskerne var i stand til at samle en kompleks seksten proteinstruktur bestående af to stablede oktamerer ved superladning alene, resultater, der blev rapporteret i januar 2019 i journalen Naturkemi .
Holdet brugte derefter supercomputersimuleringer til at validere og informere disse eksperimentelle resultater. Supercomputertildelinger på Stampede2 ved Texas Advanced Computing Center (TACC) og Comet i San Diego Supercomputer Center (SDSC) blev tildelt forskerne gennem XSEDE, Extreme Science and Engineering Discovery Environment finansieret af National Science Foundation (NSF).
"Vi fandt ud af, at ved at tage proteiner, der normalt ikke interagerer med hinanden, vi kan lave kopier, der enten er meget positivt eller meget negativt ladede, "sagde studieforfatter Anna Simon, en postdoktor i Ellington Lab i UT Austin. "Ved at kombinere de meget positivt og negativt ladede kopier, vi kan få proteinerne til at samles til meget specifikke strukturerede samlinger, "Sagde Simon. Forskerne kalder deres strategi for 'supercharged proteinsamling, 'hvor de driver definerede proteininteraktioner ved at kombinere konstruerede superladede varianter.
"Vi udnyttede et meget velkendt og grundlæggende princip fra naturen, at modsatte ladninger tiltrækker, "tilføjede studieforfatter Jens Glaser. Glaser er assisterende forsker i Glotzer-gruppen, Institut for Kemiteknik ved University of Michigan. "Anna Simons gruppe fandt ud af, at når de blander disse ladede varianter af grønt fluorescerende protein, de får stærkt ordnede strukturer. Det var en sand overraskelse, "Sagde Glaser.
Den stablede oktamerstruktur ligner en flettet ring. Den består af 16 proteiner - to sammenflettede ringe på otte, der interagerer i meget specifikke, diskrete patches. "Grunden til, at det er så svært at konstruere proteiner, der interagerer syntetisk, er, at det er at lave disse interaktive patches og få dem alle til at ligge lige op, så de tillader proteiner at samle sig til større, regelmæssige strukturer er virkelig hårde, "forklarede Simon. De kom uden om problemet ved at tilføje mange positive og negative ladninger til at konstruere varianter af grønt fluorescerende protein (GFP), et velstuderet 'lab mus' protein afledt af Aequorea victoria vandmænd.
Det positivt ladede protein, som de kaldte cerulean fluorescerende protein (Ceru) +32, havde yderligere muligheder for at interagere med det negativt ladede protein GFP -17. "Ved at give disse proteiner alle disse muligheder, disse forskellige steder, hvor de potentielt kan interagere, de var i stand til at vælge de rigtige, "Sagde Simon." Der var visse mønstre og interaktioner, der var der, ledig, og energisk begunstiget, at vi ikke nødvendigvis på forhånd forudsagde, at de ville kunne samles i disse specifikke former. "
For at få de konstruerede ladede fluorescerende proteiner, Simon og medforfattere Arti Pothukuchy, Jimmy Gollihar, og Barrett Morrow kodede deres gener, herunder et kemisk mærke, der bruges til oprensning på bærbare stykker DNA kaldet plasmider i E. coli, høstede derefter det mærkede protein, som E. coli dyrkede. Forskerne blandede proteiner sammen. De troede oprindeligt, at proteinerne bare kunne interagere for at danne store, uregelmæssigt strukturerede klumper. "Men derefter, hvad vi blev ved med at se var dette underlige, sjov top omkring 12 nanometer, det var meget mindre end en stor klump protein, men betydeligt større end det enkelte protein, "Sagde Simon.
De målte størrelsen på partiklerne, der dannedes ved hjælp af et Zetasizer -instrument ved Texas Materials Institute i UT Austin, og verificerede, at partiklerne indeholdt både cerulean- og GFP -proteiner Förster Resonance Energy Transfer (FRET), som måler energioverførslen mellem forskellige farvede fluorescerende proteiner producerer fluorescens som reaktion på forskellige lysenergier for at se om de er tæt sammen. Negativ pletelektronmikroskopi identificerede partiklernes specifikke struktur, ledet af gruppen af David Taylor, adjunkt i molekylær biovidenskab ved UT Austin. Det viste, at 12 nm -partiklen bestod af en stablet oktamer sammensat af seksten proteiner. "Vi fandt ud af, at det var disse smukt formede blomsterlignende strukturer, "Sagde Simon. Medforfatter Yi Zhou fra Taylors gruppe i UT Austin øgede opløsningen endnu mere ved hjælp af kryo-elektronmikroskopi til at afsløre detaljer på atomniveau i den stablede oktamer.

XSEDE gav forskere adgang til Comet -supercomputeren i San Diego Supercomputer Center (til venstre) og Stampede2 -supercomputeren på Texas Advanced Computing Center (til højre). Kredit:SDSC, TACC
Computational modellering raffinerede målingerne af, hvordan proteinerne var arrangeret, til et klart billede af den smukke, blomstlignende struktur, ifølge Jens Glaser. "Vi måtte finde på en model, der var kompleks nok til at beskrive fysikken i de ladede grønne fluorescerende proteiner og præsentere alle de relevante atomistiske detaljer, alligevel var den effektiv nok til at tillade os at simulere dette på en realistisk tidsplan. Med en fuldstændig atomistisk model, det ville have taget os over et år at få en enkelt simulering ud af computeren, hvor hurtig computeren end var, "Sagde Glaser.
De forenklede modellen ved at reducere opløsningen uden at ofre vigtige detaljer om interaktionerne mellem proteiner. "Derfor brugte vi en model, hvor proteinets form nøjagtigt repræsenteres af en molekylær overflade, ligesom den, der måles ud fra proteinets krystallografiske struktur, "Tilføjede Glaser.
"Det, der virkelig hjalp os med at vende dette og forbedre, hvad vi kunne få ud af vores simuleringer, var cryo-EM-dataene, "sagde Vyas Ramasubramani, en kandidatstuderende i kemiteknik ved University of Michigan. "Det var det, der virkelig hjalp os med at finde den optimale konfiguration til at sætte i disse simuleringer, som derefter hjalp os med at validere de stabilitetsargumenter, vi fremførte, og forhåbentlig fremover forudsige måder, hvorpå vi kan destabilisere eller ændre denne struktur, "Sagde Ramasubramani.
Forskerne krævede masser af computerkraft til at udføre beregningerne på den skala, de ønskede.
"Vi brugte XSEDE til stort set at tage disse enorme systemer, hvor du har mange forskellige stykker, der interagerer med hinanden, og beregne alt dette på én gang, så når du begynder at flytte dit system fremad gennem et stykke tid, du kunne få en idé om, hvordan det ville udvikle sig på noget reelle tidsskalaer, "Sagde Ramasubramani." Hvis du forsøgte at lave den samme slags simulering, som vi gjorde på en bærbar computer, det ville have taget måneder, hvis ikke år, virkelig at nærme sig forståelsen af, om en slags struktur ville være stabil eller ej. For os, ikke i stand til at bruge XSEDE, hvor du hovedsageligt kunne bruge 48 kerner, 48 beregner enheder på én gang for at gøre disse beregninger meget parallelle, vi ville have gjort det meget langsommere. "
Stampede2 -supercomputeren på TACC indeholder 4, 200 Intel Knights Landing og 1, 736 Intel Skylake X -beregningsknudepunkter. Hver Skylake -knude har 48 kerner, grundenheden i en computerprocessor. "Skylake-noder på Stampede2-supercomputeren var med til at opnå den ydelse, der var nødvendig for at beregne disse elektrostatiske interaktioner, der virker mellem de modsat ladede proteiner på en effektiv måde, " Glaser said. "The availability of the Stampede2 supercomputer was at just the right point in time for us to perform these simulations."
I første omgang, the science team tested their simulations on the Comet system at the SDSC. "When we were first figuring out what kind of model to use and whether this simplified model would give us reasonable results, Comet was a great place to try these simulations, " Ramasubramani said. "Comet was a great testbed for what we were doing."
Looking at the bigger scientific picture, the scientists hope that this work advances understanding of why so many proteins in nature will oligomerize, or join together to form more complex and interesting structures.
"We showed that there doesn't need to be a very specific, pre-distinguished set of plans and interactions for these structures to form, " Simon said. "This is important because it means that maybe, and quite likely we can take other sets of molecules that we want to make oligomerize and generate both positively charged and negatively charged variants, combine them, and have specifically ordered structures for them."
Natural biomaterials like bone, feathers, and shells can be tough yet lightweight. "We think supercharged protein assembly is an easier way to develop the kind of materials that have exciting synthetic properties without having to spend so much time or having to know exactly how they're going to come together beforehand, " Simon said. "We think that will accelerate the ability to engineer synthetic materials and for discovery and exploration of these nanostructured protein materials."
Studiet, "Supercharging Enables Organized Assembly of Synthetic Biomolecules, "blev offentliggjort i tidsskriftet Naturkemi in January of 2019.
 Varme artikler
Varme artikler
-
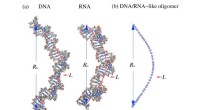 Ny teori om polymerlængde giver forbedrede estimater af DNA- og RNA -størrelseIllustrationer af dobbeltstrenget DNA, RNA og en ormelignende perlekædemodel.Den radiale fordeling af ende-til-ende-afstanden, Ree, og konturlængde, L, vises. Kredit:Xi Zhang/Bao/Wu/Zhu/Tan I mods
Ny teori om polymerlængde giver forbedrede estimater af DNA- og RNA -størrelseIllustrationer af dobbeltstrenget DNA, RNA og en ormelignende perlekædemodel.Den radiale fordeling af ende-til-ende-afstanden, Ree, og konturlængde, L, vises. Kredit:Xi Zhang/Bao/Wu/Zhu/Tan I mods -
 Nyt energiomdannelseslag til biosolcellerEn bioelektrode med proteinkomplekset Fotosystem I under bestråling med rødt lys til måling af fotostrømmens respons. Kredit: Felipe Conzuelo Et forskerhold fra Ruhr-Universität Bochum (RUB), samm
Nyt energiomdannelseslag til biosolcellerEn bioelektrode med proteinkomplekset Fotosystem I under bestråling med rødt lys til måling af fotostrømmens respons. Kredit: Felipe Conzuelo Et forskerhold fra Ruhr-Universität Bochum (RUB), samm -
 Varme elektroner sender kuldioxid tilbage til fremtidenKatalysatornanopartiklerne udviklet af KAUST-forskere bruger lysenergi til at omdanne kuldioxid og brint til metan. Kredit:KAUST; Anastasia Serin Atmosfærisk kuldioxid (CO 2 ) er en væsentlig dr
Varme elektroner sender kuldioxid tilbage til fremtidenKatalysatornanopartiklerne udviklet af KAUST-forskere bruger lysenergi til at omdanne kuldioxid og brint til metan. Kredit:KAUST; Anastasia Serin Atmosfærisk kuldioxid (CO 2 ) er en væsentlig dr -
 Ny indsigt i lithium-ion batteri fejlmekanismeForskere har identificeret en potentiel ny nedbrydningsmekanisme for batterier til elektriske køretøjer - et nøgletrin til at designe effektive metoder til at forbedre batteriets levetid. Forskerne,
Ny indsigt i lithium-ion batteri fejlmekanismeForskere har identificeret en potentiel ny nedbrydningsmekanisme for batterier til elektriske køretøjer - et nøgletrin til at designe effektive metoder til at forbedre batteriets levetid. Forskerne,
- Børn kan have brug for mere hjælp til at finde svar på deres spørgsmål i informationsalderen
- Og så var der lys:på udkig efter de første stjerner i universet
- Modificeret RNA skaber stabile terapeutiske nanopartikler
- Hvilke frø vil germinere hurtigst til et videnskabsmæssigt projekt?
- Stor test på vej for små satellitter, der følger Mars-landeren
- Sådan lyser du skærmen på et Texas Instruments TI-85 Calculator


