Brug af Raman mikrospektroskopi til hurtigt at opdage sygdomsfremkaldende bakterier
Kredit:Srividya Kumar
Forskere ved Indian Institute of Science (IISc) har udviklet en metode til hurtigt at identificere og kontrollere, om en sygdomsfremkaldende bakterie er levende eller død.
Den bruger Raman-spektroskopi, en teknik, der normalt bruges til at identificere kemiske bindinger i materialer, at genkende bakterier og kontrollere deres levedygtighed, eller deres tilstand af at være i live.
"Det unikke ved denne undersøgelse er hurtigheden og følsomheden af metoden, og dets potentiale for at blive ændret til en seng, bordplade enhed til diagnose, " siger Srividya Kumar, en tidligere ph.d. studerende ved Institut for Uorganisk og Fysisk Kemi, og førsteforfatter af undersøgelsen offentliggjort i tidsskriftet Analytisk og bioanalytisk kemi .
Hurtig påvisning af det sygdomsfremkaldende middel eller patogen, samt kontrol af, om det er i live eller ej i patientprøver, er nøglen til behandling af infektionssygdomme. At identificere patogenets levedygtighed hjælper også læger med at beslutte, hvilken dosis antibiotika der skal ordineres, og mindsker muligheden for overrecept, der kan føre til antibiotikaresistens.
Infektiøse bakterier identificeres normalt ved teknikker som dyrkning - dyrkning af dem på et næringsmedium i en petriskål. Det kan tage omkring to til tre dage at spore deres vækst og bekræfte, om de er døde eller levende. Ud over, bakterier, der er svære at dyrke i laboratoriekulturer, bliver ofte uopdaget med konventionelle metoder. Mere sofistikerede metoder såsom Polymerase Chain Reaction (PCR) skaber en genetisk profil af mikroben, men kan ikke sige, om den er levende eller død.
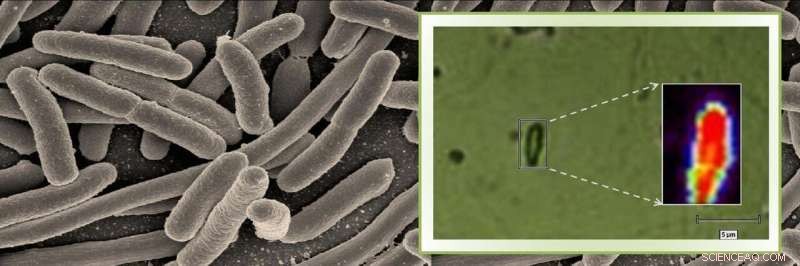
Raman-spektroskopi bruges i vid udstrækning inden for kemi til at undersøge strukturen af molekyler. I dette studie, imidlertid, Kumar og hendes kolleger har kombineret denne tilgang med avanceret mikroskopi for at kontrollere tilstedeværelsen af bakterier. De brugte denne teknik til at opdage, om tuberkulosebakterien (Mycobacterium tuberculosis) var til stede i et prøvemedium, der ligner sputum. De testede også prøver indeholdende fem andre mikrober.
Ved hjælp af mikrospektroskopet, forskerne kastede en laserstråle fra prøven og fangede lyset spredt af prøven i form af et spektrum, som varierer med den biokemiske sammensætning af bakterien. Hver bakterieart genererer et unikt spektrum med specifikke værdier for intensitet og bølgelængdeposition af det spredte lys, afhængig af typen af kemiske bindinger, der findes inde i bakteriecellen. Teknikken var følsom nok til at generere et spektrum selv for en enkelt bakteriecelle.
Materialet, der bruges til at montere bakterierne i denne teknik, kendt som substratet, er også afgørende, fordi nogle substrater kan tilføje støj til den biokemiske profil. Forskerne udviklede en neutral, aluminiumbaseret tyndfilmssubstrat, som ikke genererer baggrundssignaler, der kan interferere med spektrene.
Ved at bruge denne metode, forskerne var ikke kun i stand til at identificere bakterien, men var også i stand til at skelne mellem levende og døde bakterieceller - da den kemiske sammensætning af de to er forskellig - inden for to til tre timer efter prøvetagning.
Da hver bakterieart giver anledning til et unikt spektrum, det kan være muligt på sigt at opbygge en database over patogener, der kan bruges i klinisk diagnose.
"Selvom vi har valideret metoden til tuberkulose, metoden kan udvides til enhver type bakteriel infektion, " siger Deepak Saini, lektor ved Institut for Molekylær Reproduktion, Udvikling og genetik, IISc, og en af de ledende forfattere af undersøgelsen.
 Varme artikler
Varme artikler
-
 Blåt pigment fra manipulerede svampe kan hjælpe med at gøre tekstilindustrien grønLederforsker Aindrila Mukhopadhyay har et hætteglas med rensede indigoidine krystaller. Kredit:Marilyn Chung/Berkeley Lab Tit, resultaterne af grundlæggende videnskabelig forskning er mange skridt
Blåt pigment fra manipulerede svampe kan hjælpe med at gøre tekstilindustrien grønLederforsker Aindrila Mukhopadhyay har et hætteglas med rensede indigoidine krystaller. Kredit:Marilyn Chung/Berkeley Lab Tit, resultaterne af grundlæggende videnskabelig forskning er mange skridt -
 Ny immunterapiteknik kan specifikt målrette tumorceller, undersøgelsesrapporterDenne teknologi er særligt spændende, fordi den afmonterer store udfordringer inden for kræftbehandlinger, ” siger Weian Zhao. Kredit:Steve Zylius / UCI En ny immunterapi screening prototype udvik
Ny immunterapiteknik kan specifikt målrette tumorceller, undersøgelsesrapporterDenne teknologi er særligt spændende, fordi den afmonterer store udfordringer inden for kræftbehandlinger, ” siger Weian Zhao. Kredit:Steve Zylius / UCI En ny immunterapi screening prototype udvik -
 Aluminosilikatglas kan tjene som synlige luminescensmaterialerKarakteriseringer af Pr 3+ -doterede aluminiumsilikatglas. Kredit:SIOM Bredbåndsemissioner i det synlige spektralområde kan opnås ved at pumpe Pr 3+ dopet i krystal, keramik og glas. Fluorglas
Aluminosilikatglas kan tjene som synlige luminescensmaterialerKarakteriseringer af Pr 3+ -doterede aluminiumsilikatglas. Kredit:SIOM Bredbåndsemissioner i det synlige spektralområde kan opnås ved at pumpe Pr 3+ dopet i krystal, keramik og glas. Fluorglas -
 Forskere opdager, hvordan et protein reducerer den negative virkning af vandtab i cellerKredit:CC0 Public Domain En forsker fra University of Houston College of Medicine har fundet ud af, hvordan et protein inde i kroppen reducerer de negative virkninger af hypertonicitet, en ubalanc
Forskere opdager, hvordan et protein reducerer den negative virkning af vandtab i cellerKredit:CC0 Public Domain En forsker fra University of Houston College of Medicine har fundet ud af, hvordan et protein inde i kroppen reducerer de negative virkninger af hypertonicitet, en ubalanc
- Det magnetiske felt i den galaktiske udstrømning af M82
- At finde en ny måde at bekæmpe sepsis i sen fase ved at øge cellernes antibakterielle egenskaber
- Pludselige klimaændringer drev den tidlige sydamerikanske befolkningsnedgang
- Vrikkende atomer skifter krystallers elektriske polarisering
- Forbedring af naturværktøjer til fordøjelse af plast
- Hvordan investeringer i kunstvanding betaler sig for Etiopiens økonomi


