Trækker stikket på coronavirus kopimaskine
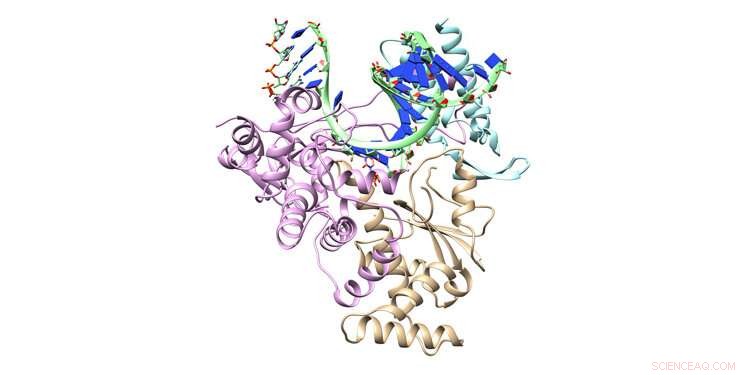
Antiviralt lægemiddel remdesivir udgør hovedlinjen i FDA-godkendt terapeutisk forsvar mod COVID-19-virussen. Forskere ved University of North Texas bruger Frontera-supercomputeren til at modellere, hvordan remdesivir blokerer coronavirus-reproduktion, i håb om at udvikle forbedringer af lægemidlet. Vist her er krystalstrukturerne af den RNA-afhængige RNA-polymerase-ternære kompleksmodel med dobbeltstrenget RNA og indkommende remdesivirtrifosfat. Kredit:Cisneros Research Group, UNT
Nøgleproteiner, der bruges af coronavirus til dets reproduktion, er modelleret på NSF-finansieret Frontera supercomputer af Andres Cisneros forskningsgruppe fra University of North Texas. Forskningsmål omfatter at finde måder at forbedre COVID-19-terapeutiske remdesivir på. NSF-finansieret Frontera-tildeling tildelt Cisneros gennem COVID-19 High Performance Computing Consortium.
I maj 2020, U.S. Food and Drug Administration godkendte det antivirale lægemiddel remdesivir til akut behandling af COVID-19, en af kun fire lægemidler i øjeblikket med denne status. Remdesivir stopper det kemiske maskineri, som coronavirus bruger til at kopiere sig selv, binding til et enzym, der udfører samlingen. Mens remdesivir har vist lovende at hjælpe patienter med at komme sig fra COVID-19, videnskabsmænd undersøger måder at forbedre dets effektivitet på.
Et team af videnskabsmænd ledet af G. Andres Cisneros fra University of North Texas modellerer de vigtigste dele af coronavirus, som det bruger til at kopiere sig selv. Simuleringerne udføres på supercomputerne Stampede2 og Frontera ved Texas Advanced Computing Center (TACC).
"Vi var meget heldige at få tildelt en tildeling på Frontera for at kunne arbejde på at undersøge mekanismen for lægemidler, der er målrettet mod to specifikke proteiner i COVID-19, " sagde Cisneros. Hans arbejde undersøger, hvordan remdesivir og andre tilgængelige lægemidler hæmmer proteinerne NSP-12 og hovedproteasen, begge enzymer, som coronavirus har brug for til replikation. "Ved at se på, hvordan disse stoffer gør deres arbejde, måske kan denne information bruges til at forbedre dem."
NSP-12-proteinet sammensætter de nukleotider, der udgør viralt RNA, forkortet til A, U, G, og C, bygge komplette sæt genetisk materiale til nye coronavirus-kopier. NSP-12 er faktisk en del af en større struktur kaldet den RNA-afhængige RNA-polymerase (RDRP), der kopierer det komplette RNA. Remdesivir binder til RDRP, tilslutning af maskineriet.
"Vi undersøger, hvordan denne proces sker, " sagde Cisneros. "Ved at gøre dette, måske er der en måde for os og andre forskere at komme med ideer til, om og hvordan remdesivir kan forbedres."
Det andet protein Cisneros studerer kaldes hovedproteasen. Det adskiller et polyprotein produceret af SARS-CoV-2 oversat fra viralt RNA til funktionelle proteiner, der sætter "kød" på dets virale knogler. Stop proteasen, og du stopper virussen i at dannes. Dette gør det til et fantastisk lægemiddelmål.
Cisneros forklarede, at han bruger den grundlæggende matematik og fysik i Newtons ligninger og kvantemekanik til at beregne proteinernes egenskaber, inklusive alt, der er relevant for dets funktion, såsom RNA og vand. En tilgang kaldet klassisk molekylær dynamik bruger Newtons ligninger til at simulere, hvordan proteinerne bevæger sig og interagerer dynamisk i tiden. "Vi taler om systemer, som vi simulerer, der er i hundredtusindvis af atomer, " sagde Cisneros.
Han simulerer også de kemiske reaktioner inde i proteinerne for at undersøge, hvordan stofferne stopper RDRP eller proteasen. En hybridmetode kaldet QM/MM (kvantemekanik/molekylær mekanik) sparer beregningstid og penge ved at fokusere mere intenst på interaktioner på det aktive sted, bruger den mere omtrentlige lige molekylære dynamik til alt andet.
Cisneros-gruppen udviklede og vedligeholder et program kaldet LICHEM, som lader dem bruge QM/MM-tilgangen. "Et af kendetegnene ved LICHEM er, at det giver os mulighed for at bruge tilgange til den klassiske mekanik del, der inkluderer en bedre beskrivelse af den fysik, der sker mellem molekylerne i det klassiske miljø, specifikt, AMOEBA-potentialet" sagde Cisneros. AMOEBA er udviklet af Pengyu Ren fra UT Austin; Jay Ponder fra University of Washington; og Jean-Philip Piquemal ved Sorbonne University i Paris med bidrag fra Cisneros-gruppen for ioniske væsker.
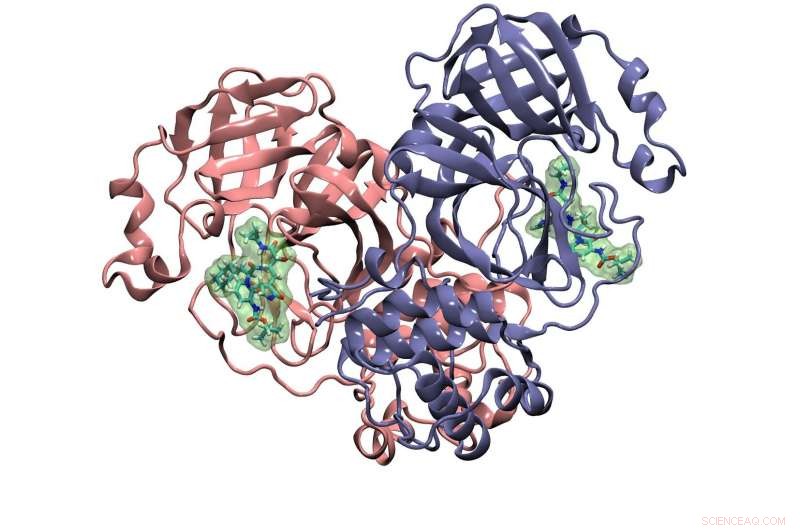
Et andet forskningsmål for Cisneros-gruppen, der modelleres på Frontera, er et protein kaldet hovedproteasen. Det spalter et polyprotein produceret af virussen, der opbygger de funktionelle proteiner af de kopier af sig selv, det genererer. Krystalstruktur af coronavirus-hovedproteasen med bundet inhibitor vist her. Kredit:Cisneros Research Group, UNT
"Frontera, med ikke kun regnekraft, men også interkommunikationen mellem knudepunkterne, giver os mulighed for at køre disse QM/MM-beregninger med meget højere, ikke kun hastighed, men også gennemløb, " sagde Cisneros. Frontera frigjorde dem til at køre flere systemer ad gangen. "I min gruppe, Jeg har fem forskellige videnskabsmænd, kandidatstuderende og post docs, der arbejder på begge disse systemer, men i forskellige brikker af puslespillet. Alle er i stand til at få adgang til disse ressourcer. Det er bestemt meget nyttigt, og vi sætter stor pris på tildelingen."
Det, der fik Cisneros i gang, var nyheder i april 2020 om krystalstrukturen af SARS-CoV-2 RDRP, der blev rapporteret. "Jeg kontaktede min gruppe og fortalte dem, at med disse oplysninger, der er noget vi kan gøre for at hjælpe med pandemien, " han sagde.
Inden for to dage efter nyheden, Cisneros foreslog med succes sin forskning om lægemiddelmål for coronavirus til COVID-19 High Performance Computing Consortium. Dusinvis af nationale og internationale supercomputing faciliteter, industri, og organisationer, herunder TACC, har givet deres ressourcer til konsortiet til støtte for videnskabsmænds indsats for at bekæmpe coronavirus.
Tildelingen blev oprindeligt tildelt netop på TACC's Stampede2, supercomputing-flagskibet for National Science Foundation (NSF), der er rangeret som den 21. hurtigste i verden og #2 for akademiske systemer ifølge Top500. "Så blev vi kontaktet af TACC og taknemmelige for, at vi fik adgang til Frontera. Nu har vi adgang til begge systemer, hvilket er rigtig godt, " sagde Cisneros.
Frontera supercomputeren er den #1 hurtigste akademiske supercomputer og #8 hurtigste på verdensplan. Både Frontera og Stampede2 er finansieret af NSF.
"Vi er meget glade for dette system. Vi var i stand til at overføre noget af den viden, vi havde fra Stampede2 til Frontera, " sagde Cisneros. En af hans nyligt dimitterede studerende, Erik Vazquez Montelongo, opsætte alle beregningerne for LICHEM på Frontera baseret på, hvad han lærte på Stampede2. "Det har virkelig været en velsignelse. Frontera for vores beregninger har kørt rigtig godt. Vi er rigtig glade for det."
En af postdoc'erne i The Cisneros Group, Sehr Nazeem-Kahn, genererede modellen for RDRP, remdesivir og andre lægemiddelkandidater, alt på det aktive websted. Med det i hånden, de begyndte at køre simuleringer.
"Vi var meget glade for at se, at hendes model faktisk var meget tæt på den eksperimentelle struktur. Det er virkelig nyttigt for os, fordi det validerer den model, som gruppen har bygget og viser, at vi er på rette vej, " han tilføjede.
I øjeblikket, Dr. Naseem-Khan kører molekylær dynamik simuleringer af denne model med remdesivir på Frontera. "Vi starter også med vores QM/MM-beregninger for RDRP. I tilfældet med hovedproteasen, der var strukturer, som også skulle modelleres og efterfølgende blev bekræftet. Det var også meget tilfredsstillende, " sagde Cisneros.
Med den strukturdata, de kigger på seks forskellige inhibitormolekyler. "En af de der, vi starter allerede QM/MM-beregninger på Frontera, og endnu en på Stampede2, " sagde Cisneros. Hvis alt går vel, han håber at få resultater inden for de næste fem til seks måneder. "Det er meget dyre beregninger, " tilføjede han. "Også, at køre analysen tager tid. Hvis vi kun skulle bruge ressourcerne derhjemme, det ville tage flere år."
 Varme artikler
Varme artikler
-
 Mod sikrere bortskaffelse af printkortKredit:American Chemical Society Printplader er vitale komponenter i moderne elektronik. Imidlertid, når de har tjent deres formål, de bliver ofte brændt eller begravet på lossepladser, forurener
Mod sikrere bortskaffelse af printkortKredit:American Chemical Society Printplader er vitale komponenter i moderne elektronik. Imidlertid, når de har tjent deres formål, de bliver ofte brændt eller begravet på lossepladser, forurener -
 Registrering af spormængder af flere klasser af antibiotika i fødevarerKredit:American Chemical Society Udbredt brug af antibiotika i menneskers sundhedspleje og husdyrhold har ført til, at spormængder af stofferne ender i fødevarer. Langtidsforbrug kan forårsage sun
Registrering af spormængder af flere klasser af antibiotika i fødevarerKredit:American Chemical Society Udbredt brug af antibiotika i menneskers sundhedspleje og husdyrhold har ført til, at spormængder af stofferne ender i fødevarer. Langtidsforbrug kan forårsage sun -
 Manipulerende ligander:En ny dimension til fremme af elektrokatalyse ydeevne af ædelmetal aerogelerI lyset af renligheden af laserproducerede metal nanopartikelforstadier, overflade-ren ædelmetal aerogeler er skabt. NMAernes iboende elektrokatalytiske egenskaber belyses, og ligandmodulerede elekt
Manipulerende ligander:En ny dimension til fremme af elektrokatalyse ydeevne af ædelmetal aerogelerI lyset af renligheden af laserproducerede metal nanopartikelforstadier, overflade-ren ædelmetal aerogeler er skabt. NMAernes iboende elektrokatalytiske egenskaber belyses, og ligandmodulerede elekt -
 Forskere forudser nye superhårde materialerKredit:Pixabay/CC0 Public Domain En gruppe Skoltech -forskere brugte machine learning (ML) metoder til at forudsige superhårde materialer baseret på deres krystalstruktur. Forskningen blev offent
Forskere forudser nye superhårde materialerKredit:Pixabay/CC0 Public Domain En gruppe Skoltech -forskere brugte machine learning (ML) metoder til at forudsige superhårde materialer baseret på deres krystalstruktur. Forskningen blev offent
- Sådan beregnes området med skraverede regioner
- Forskere deltager i månedslange eksperiment for at studere indendørs luftforurening
- Søgning efter menneskelige rester:Undersøgelse foreslår metode til at forbedre resultater
- Fejl betyder ekstra dag for amerikanerne til at indgive skat
- Nyt estimat af muonernes magnetiske feltstyrke stemmer overens med standardmodel for partikelfysik
- Snefnug er ikke så unikt, som du tror


