Simulering af 3D-AFM-billeder for systemer, der ikke er i ligevægt
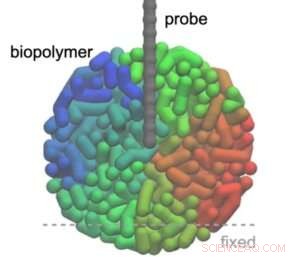
Kredit:Kanazawa University
Forskere ved Kanazawa University rapporterer i Journal of Physical Chemistry Letters hvordan man simulerer 3D atomic force mikroskopi billeder af ude af ligevægt systemer, der involverer biomolekyler. Fremgangsmåden gør brug af en berømt ligning fra termodynamik, der kan anvendes til ikke-ligevægtssituationer.
Tredimensionel atomkraftmikroskopi (3D-AFM) er en teknik, der bruges til at undersøge fordelingen af opløsningsmiddelmolekyler ved faststof-væske-grænseflader. Oprindeligt anvendt til at studere situationer, hvor opløsningsmidlet er vand, bliver metoden nu også brugt til andre molekyler. En nylig udvikling er at bruge 3D-AFM til at løse organiseringen af biopolymerer såsom kromosomer eller proteiner i celler. På grund af sådanne systemers kompleksitet er der imidlertid behov for simuleringer af 3D-AFM billeddannelsesprocessen for at hjælpe med fortolkningen. Simuleringsmetoder udviklet hidtil har antaget, at det sonderede system er i ligevægt under AFM-scanningscyklussen. Dette begrænser deres gyldighed til situationer, hvor opløsningsmiddelmolekylerne bevæger sig meget hurtigere end scanningssonden. Takeshi Fukuma fra Kanazawa University og kolleger har nu udviklet en 3D-AFM-simuleringstilgang, der fungerer for systemer, der ikke er i ligevægt, og som kan anvendes i situationer, hvor molekylær bevægelse sker på tidsskalaer, der er sammenlignelige med eller større end AFM-sonderingscyklussen.
Det grundlæggende princip for AFM er at lave en meget lille spids, fastgjort til en cantilever, for at scanne en prøves overflade. Spidsens reaktion på højdeforskelle i den scannede overflade giver strukturel information om prøven. I 3D-AFM er spidsen lavet til at penetrere prøven, og den kraft, spidsen oplever, er resultatet af interaktioner med nærliggende (dele af) molekyler. For en given vandret (xy) position af spidsen er afhængigheden af kraften F af spidsens lodrette (z) position, når den trænger ind i prøven, fanget i en kraft-afstand (F versus z) kurve. Kombination af alle kraft-afstandskurver opnået under xy-scanningen giver 3D-AFM-billedet.
Fukuma og kolleger overvejede situationen, hvor en AFM-spids sonderer en kugleformet biopolymer og modellerede både spids og molekyle som perler forbundet med fjedre (2.000 perler til molekylet, 50 perler til spidsen). De beregnede kraft-afstandskurverne ved at bruge den såkaldte Jarzynski-lighed, en ligning, der relaterer den frie energiforskel mellem to tilstande i et system til det arbejde (proportionalt med kraften), der kræves for at gå fra den ene tilstand til den anden. Det er vigtigt, at ligheden gælder for ikke-ligevægtssituationer.
Forskerne var i stand til at vise, at simuleringerne gengav den indre struktur af biopolymeren, hvor nogle fibertræk tydeligt kunne observeres. De så også på, hvordan scanningshastigheden påvirker simuleringsresultaterne, og fandt ud af, at der er et optimalt hastighedsområde for den vertikale (z) scanning. Endelig simulerede Fukuma og kolleger 3D-AFM-billeder af cytoskeletfibre, for hvilke der eksisterer eksperimentelt opnåede 3D-AFM-billeder, og fandt ud af, at simuleringerne stemmer godt overens med eksperimentet. Forskerne konkluderede derfor, at deres metode "er anvendelig til forskellige fibre i celler såsom DNA og så videre ved at ændre parametre såsom stivhed, hvilket giver en vigtig teoretisk base for sådanne eksperimentelle målinger." + Udforsk yderligere
Tildeling af bevægelige funktioner i højhastigheds atomkraftmikroskopi
 Varme artikler
Varme artikler
-
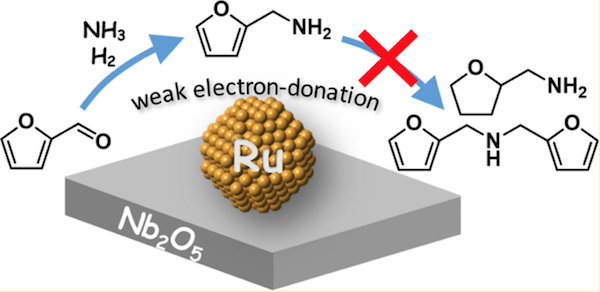 Genanvendelig ruthenium-baseret katalysator kunne være en game-changer for biomasseindustrienRuthenium (Ru) nanopartiklers svage elektrondonerende evne understøttet af niobiumpentoxid (Nb 2 O 5 ) menes at fremme reduktiv aminering og samtidig forhindre dannelsen af uønskede biprodukter.
Genanvendelig ruthenium-baseret katalysator kunne være en game-changer for biomasseindustrienRuthenium (Ru) nanopartiklers svage elektrondonerende evne understøttet af niobiumpentoxid (Nb 2 O 5 ) menes at fremme reduktiv aminering og samtidig forhindre dannelsen af uønskede biprodukter. -
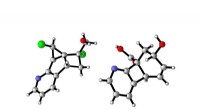 Kemikere bruger lysenergi til at producere små molekylære ringeEnkeltkrystal røntgenstrukturer af to produkter, der stammer fra cycloadditionsreaktionen. Især til venstre er et unikt fem-fire-treleddet kondenseret ringsystem synligt. Til højre kan en fire-seks-le
Kemikere bruger lysenergi til at producere små molekylære ringeEnkeltkrystal røntgenstrukturer af to produkter, der stammer fra cycloadditionsreaktionen. Især til venstre er et unikt fem-fire-treleddet kondenseret ringsystem synligt. Til højre kan en fire-seks-le -
 Ny teknik til at måle temperaturer i forbrændingsflammer kan føre til renere biobrændstofferMålte og simulerede kryptontaltætheder i en sodende metan/luftflamme. (A) Et fotografi af flammen dimensioneret til samme rumlige skala som (B). (B) Billedplot af eksperimentelle (venstre) og simulere
Ny teknik til at måle temperaturer i forbrændingsflammer kan føre til renere biobrændstofferMålte og simulerede kryptontaltætheder i en sodende metan/luftflamme. (A) Et fotografi af flammen dimensioneret til samme rumlige skala som (B). (B) Billedplot af eksperimentelle (venstre) og simulere -
 Hvordan virker fotosyntese i planter?Grønne planter bruger fotosyntese til at skabe energi fra kuldioxid og sollys. Denne energi, i form af glucose, bruges af planten til at vokse og brænde plantens nødvendige reproduktive aktiviteter. O
Hvordan virker fotosyntese i planter?Grønne planter bruger fotosyntese til at skabe energi fra kuldioxid og sollys. Denne energi, i form af glucose, bruges af planten til at vokse og brænde plantens nødvendige reproduktive aktiviteter. O
- Dyrkning af en dinosaur -middag
- Frysepunkt Forklaring til børn
- Molekylære nanocarboner med mekaniske bindinger
- Hvordan man forbedrer matematikfærdigheder i College Students
- Byrden af COVID-19-pandemien kan bidrage til udbrud af voldelige protester og anti-statslige føle…
- Skovregenerationsforsøg på 30 år giver resultater


