Ny teknik til at studere membran-associerede iboende forstyrrede proteiner
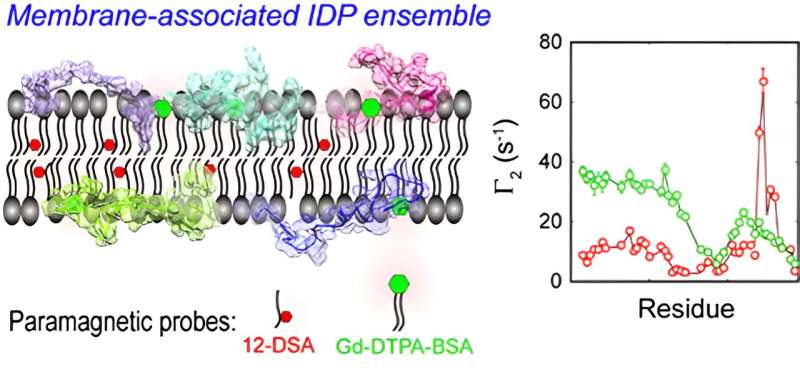
Intrinsically disordered proteiner (IDP'er) findes i vid udstrækning i eukaryotes proteomer og spiller nøgleroller i livsprocesser såsom transkription af genetisk information og signalering. Ud over at være normalt meget gentagne, hydrofile og elektrisk ladede samt koder for simple sekvenser af gener, udmærker IDP'er sig også i deres naturlige overflod og strukturelle aspekter, som bliver grundlaget for proteinernes "lidelse-funktionsparadigme".
I løbet af de sidste to årtier er IDP'ers rolle i humane sygdomme og som lægemiddelmål blevet aktivt undersøgt, mens hvordan man karakteriserer de meget fleksible og heterogene konformationer af IDP'er ved høj opløsning, er fortsat et nøglespørgsmål på dette område.
Mere end 15 % af IDP-molekylerne er membranbundne i celler, og deres indre dynamik og overordnede (translationelle og roterende) bevægelser i fosfolipid-dobbeltlagene er tæt forbundet med deres fysisk-kemiske egenskaber og biologiske funktioner, men disse dynamiske processer er svære at fange og kvantitativt karakteriseret ved konventionelle strukturanalysemetoder.
En gruppe forskere ledet af prof. Long Dong fra University of Science and Technology of China (USTC) har udviklet en IDP-spektroskopimetode baseret på membran paramagnetic relaxation enhancement (mPRE)-teknikken, som med succes har opnået højpræcisionsmodellering af interne konformationer, orienteringer og fordybelsesdybder af IDP'er. Resultaterne er blevet offentliggjort i Journal of the American Chemical Society .
I dette arbejde undersøgte forskerne i detaljer fleksibiliteten og mobiliteten af spinprobe-molekylerne i membranen til nøjagtig fortolkning af mPRE-spektraldataene og foreslog en vægtet tredimensionel (3D) gittermodel baseret på alle-atom-simuleringer til kvantitativt skildrer effekten af spin-probens dynamik på membranens paramagnetiske afslapningsforstærkningshastighed.
Ved at udnytte modellens høje beregningseffektivitet videreudviklede forskerne en algoritme, som er detaljeret i Supporting Information (SI) til optimering af orienteringer af de globale og interne grader af bevægelsesfrihed af membranbundne IDP'er via superposition af z- Kun koordinater, skræddersyet til mPRE-dataanalysen, konstruerer en alt-atom ensemblemodel af IDP i et implicit membranmiljø.
CD3ε er en komponent af T-celle receptor (TCR) komplekset ansvarlig for T-celle antigen genkendelse. CD'en3ε cytoplasmatisk domæne (CD3εCD ) indeholder immunoreceptor-tyrosin-baserede aktiveringsmotiver (ITAM'er), og det danner et fuzzy kompleks med lipid-dobbeltlag i en iboende forstyrret tilstand og regulerer signalaktiviteten af fuzzy kompleks ved at bruge dynamisk membranafskærmning af vigtige tyrosinsteder.
Forskerne løste ensemblet baseret på molekylær dynamik af CD3εCD i lipid-dobbeltlag ved at anvende en alt-atom ensemble modelopløsning af IDP i et implicit membranmiljø.
Ensemblet fra mPRE eksperimentelle parametre kortlægger den dynamiske fordeling af CD3εCD i forskellige områder af membranen på atomniveau og afslører vigtige forskelle i membraninteraktionerne mellem forskellige tyrosinsteder på ITAM, hvilket giver en ny mekanistisk forklaring på monophosphoryleringsmønsteret af ITAM.
Den mPRE-spektroskopiske analysemetode, der er etableret i dette arbejde, forventes bredt at lette atomopløsningsundersøgelser af forskellige funktionelle membran-IDP'er.
Flere oplysninger: Hong Jin et al., Quantitative Ensemble Interpretation of Membrane Paramagnetic Relaxation Enhancement (mPRE) for Studying Membrane-Associated Intrinsically Disordered Proteins, Journal of the American Chemical Society (2023). DOI:10.1021/jacs.3c10847
Journaloplysninger: Tidsskrift for American Chemical Society
Leveret af University of Science and Technology i Kina
 Varme artikler
Varme artikler
-
 Selvsamling påkrævet:Neutrons sonde ny polymeradfærd for biomedicinske materialerORNL-instrumentforsker Shuo Qian forbereder Bio-SANS-instrumentet på HFIR til et eksperiment ledet af LSU-professor Donghui Zhang. Zhang bruger neutronspredning til at studere alsidige proteinlignende
Selvsamling påkrævet:Neutrons sonde ny polymeradfærd for biomedicinske materialerORNL-instrumentforsker Shuo Qian forbereder Bio-SANS-instrumentet på HFIR til et eksperiment ledet af LSU-professor Donghui Zhang. Zhang bruger neutronspredning til at studere alsidige proteinlignende -
 Efter 150 år, et gennembrud i forståelsen af omdannelsen af CO2 til elektrobrændstofferVisualisering af et skift fra luftforurenende økonomi baseret på fossile brændstoffer til ren økonomi baseret på vedvarende energi, som lettes af elektrokatalytisk omdannelse af rigeligt CO 2 til br
Efter 150 år, et gennembrud i forståelsen af omdannelsen af CO2 til elektrobrændstofferVisualisering af et skift fra luftforurenende økonomi baseret på fossile brændstoffer til ren økonomi baseret på vedvarende energi, som lettes af elektrokatalytisk omdannelse af rigeligt CO 2 til br -
 Forskere laver syntetiske polymerer inde i levende cellerKredit:CC0 Public Domain Et team af forskere ved University of Edinburgh har vist, at det er muligt at skabe syntetiske polymerer inde i levende celler. I deres papir offentliggjort i tidsskriftet
Forskere laver syntetiske polymerer inde i levende cellerKredit:CC0 Public Domain Et team af forskere ved University of Edinburgh har vist, at det er muligt at skabe syntetiske polymerer inde i levende celler. I deres papir offentliggjort i tidsskriftet -
 Maling med halvlederePerovskite portræt af Marie Skłodowska-Curie. Kredit:Lukas Helmbrecht AMOLF-forskerne Lukas Helmbrecht og Wim Noorduin har udviklet en reaktiv blæk, der kan males på et lige så reaktivt lærred. Bl
Maling med halvlederePerovskite portræt af Marie Skłodowska-Curie. Kredit:Lukas Helmbrecht AMOLF-forskerne Lukas Helmbrecht og Wim Noorduin har udviklet en reaktiv blæk, der kan males på et lige så reaktivt lærred. Bl
- NuSTAR undersøger forvirrende galaksefusion
- Eksperter opfordrer til en gennemgang af matematikundervisningen efter undersøgelse viser, at børn…
- Fossil antyder aber, gamle verdens aber bevægede sig i modsatte retninger fra fælles forfader
- Facebooks aktie synker, så hvem skal købe det?
- Hvordan bliver en tarmmikrobe et patogen?
- Samsung-forskere:Mere effektive kvanteprikker uden tungmetaller


