Forskere demonstrerer kontinuerlig og kontrolleret translokation af DNA-polymer gennem en nanopore
Forskning offentliggjort i denne uge i JACS ( Journal of the American Chemical Society ) viser kontinuerlig og kontrolleret translokation af en enkeltstrenget DNA (ssDNA) polymer gennem et protein nanopore af et DNA polymerase enzym. Papiret fra forskere ved University of California Santa Cruz (UCSC) danner grundlaget for en molekylær motor, en væsentlig komponent i Strand Sequencing ved hjælp af nanoporer. Forskere ved UCSC samarbejder med den britiske virksomhed Oxford Nanopore Technologies, udviklere af en nanopore DNA-sekventeringsteknologi.
Den nye forskning fremmer tidligere arbejde, der viser, at DNA kunne flyttes gennem en nanopore ved hjælp af en polymerase. DNA-bevægelse i den tidligere undersøgelse blev udført af en række polymeraser og krævede kompleks elektronik til kontrol. Forbedringer noteret i JACS-papiret omfatter teknikker til at tillade kontinuerlig ssDNA-bevægelse, giver et uafbrudt signal, da strengen blev flyttet gennem nanoporen i realtid. Enzym-nanopore-konstruktionen var aktiv og målbar i et konstant elektronisk felt uden kompleks elektronik.
Kontrolleret initiering af polymerasebehandlingen på stedet for nanopore-enzymkomplekset muliggjorde sekventiel måling af flere ssDNA-molekyler ved hjælp af en enkelt eksperimentel opsætning. Ydermere udviste polymerasen ihærdig binding med DNA-polymeren, i modsætning til tidligere enzymer undersøgt under lignende forhold. Disse resultater viser, at kvaliteterne af phi29-DNA-polymerasen er i overensstemmelse med en strengsekventeringsteknologi.
I 'strengsekventering'-metoden til nanopore DNA-sekventering, ionstrøm gennem et protein nanopore måles, og strømforstyrrelser bruges til at identificere baser på en ssDNA-polymer i rækkefølge, da det translokerer poren. To nøgleudfordringer for denne metode er:konstruktion af en nanopore for at muliggøre identifikation af individuelle baser, når en ssDNA-polymer spænder over poren, og en mekanisme til styring af translokation af ssDNA med en ensartet og passende hastighed for at muliggøre baseidentifikation gennem elektroniske målinger. Translokationsteknikker beskrevet i dette papir er kompatible med basisidentifikationsteknologi, der udføres i laboratorierne hos Oxford Nanopore Technologies og dets samarbejdspartnere.
"Dette arbejde med phi29-polymerasen har givet os mulighed for at gøre vigtige fremskridt med et nøgleelement i DNA-strengsekventering, " sagde efterforsker professor Mark Akeson fra University of California, Santa Cruz. "Mens tidligere arbejde viste, at translokationskontrol var mulig i teorien, dette arbejde viser, at DNA-translokationskontrol er opnåelig under forhold, der er kompatible med en elektronisk sekventeringsteknologi. Vi ser frem til yderligere samarbejde med Oxford Nanopore for at realisere denne forskning."
"Strengsekventeringsmetoden til DNA-sekventering ved hjælp af en nanopore er blevet undersøgt i mange år, men dette papir viser for første gang, at DNA kan translokeres af et enzym ved hjælp af metoder, der er i overensstemmelse med en højkapacitets elektronisk teknologi, " sagde Dr Gordon Sanghera, direktør for Oxford Nanopore. "Vi er begejstrede for dette arbejde og dets potentiale, når det kombineres med yderligere nyere udviklinger inden for DNA-baseidentifikation på DNA-strenge, det andet kritiske element for streng-sekventering."
Sidste artikelFysikere bruger grafen til at afkode DNA
Næste artikelRedder liv med hurtig sepsis-test
 Varme artikler
Varme artikler
-
 Forskere udvikler en fleksibel transparent leder fri for refleksion og spredningFlexible Transparent Conductor er vist. Kredit:ICFO Gennemsigtige ledere er et af nøgleelementerne såsom elektroniske og optoelektroniske enheder som displays, lysdioder, fotovoltaiske celler og s
Forskere udvikler en fleksibel transparent leder fri for refleksion og spredningFlexible Transparent Conductor er vist. Kredit:ICFO Gennemsigtige ledere er et af nøgleelementerne såsom elektroniske og optoelektroniske enheder som displays, lysdioder, fotovoltaiske celler og s -
 Kemikere søger state-of-the-art lithium-svovl batterierHvornår kan vi forvente at køre hele Tyskland i en elbil uden at skulle fylde batteriet op? Kemikere ved NIM Cluster på LMU og ved University of Waterloo i Ontario, Canada, har nu syntetiseret et nyt
Kemikere søger state-of-the-art lithium-svovl batterierHvornår kan vi forvente at køre hele Tyskland i en elbil uden at skulle fylde batteriet op? Kemikere ved NIM Cluster på LMU og ved University of Waterloo i Ontario, Canada, har nu syntetiseret et nyt -
 SuperSTEM-forskning afslører, at grafen strikker sine huller igenSuperSTEM2, der er kun seks af disse usædvanligt følsomme instrumenter på verdensplan. Kredit:SuperSTEM Consortium (Phys.org) - Forskere ved University of Manchester og SuperSTEM-faciliteten ved S
SuperSTEM-forskning afslører, at grafen strikker sine huller igenSuperSTEM2, der er kun seks af disse usædvanligt følsomme instrumenter på verdensplan. Kredit:SuperSTEM Consortium (Phys.org) - Forskere ved University of Manchester og SuperSTEM-faciliteten ved S -
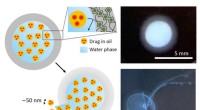 En ny platform til kontrolleret levering af vigtige lægemidler i nanoskala og mereDisse skemaer og fotografier illustrerer a) en lille kapsel indeholdende tusindvis af dråber i nanostørrelse fyldt med et lægemiddel eller en anden aktiv ingrediens; og b) hvordan dråberne brister fra
En ny platform til kontrolleret levering af vigtige lægemidler i nanoskala og mereDisse skemaer og fotografier illustrerer a) en lille kapsel indeholdende tusindvis af dråber i nanostørrelse fyldt med et lægemiddel eller en anden aktiv ingrediens; og b) hvordan dråberne brister fra
- Undersøgelse finder bevis på, at nanopartikler kan øge plante-DNA-skader
- Science Fair Project: Dehydrering Fruit
- Hydrologiske simuleringsmodeller, der informerer om politiske beslutninger, er vanskelige at fortolk…
- Sådan beregnes PH af bufferopløsninger
- Forskere afslører dynamisk sølvkrystallisering ved in-situ SEM
- Udbredelse af selvkørende biler kan forårsage mere forurening – medmindre elnettet ændrer sig rad…


