Største biokemiske kredsløb bygget ud af små syntetiske DNA-molekyler
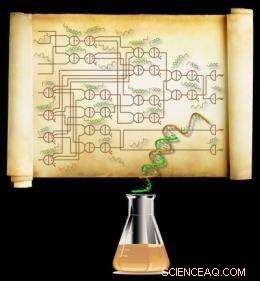
Et ledningsdiagram, der specificerer et system med 74 DNA-molekyler, der udgør det største syntetiske kredsløb af sin type, der nogensinde er lavet. Kredsløbet beregner kvadratroden af et tal op til 15 og runder ned til nærmeste heltal (den diskrete kvadratrod af et fire-bit heltal). Kredit:Caltech/Lulu Qian
På mange måder, livet er som en computer. En organismes genom er softwaren, der fortæller det cellulære og molekylære maskineri - hardwaren - hvad de skal gøre. Men i stedet for elektroniske kredsløb, liv er afhængig af biokemiske kredsløb - komplekse netværk af reaktioner og veje, der sætter organismer i stand til at fungere. Nu, forskere ved California Institute of Technology (Caltech) har bygget det mest komplekse biokemiske kredsløb, der nogensinde er skabt fra bunden, lavet med DNA-baserede enheder i et reagensglas, der er analoge med de elektroniske transistorer på en computerchip.
Konstruktion af disse kredsløb giver forskere mulighed for at udforske principperne for informationsbehandling i biologiske systemer, og at designe biokemiske veje med beslutningsevner. Sådanne kredsløb ville give biokemikere hidtil uset kontrol med at designe kemiske reaktioner til anvendelser inden for biologisk og kemisk teknik og industrier. For eksempel, i fremtiden kan et syntetisk biokemisk kredsløb indføres i en klinisk blodprøve, detektere niveauerne af en række molekyler i prøven, og integrere denne information i en diagnose af patologien.
"Vi forsøger at låne de ideer, der har haft stor succes i den elektroniske verden, såsom abstrakte repræsentationer af computeroperationer, programmeringssprog, og kompilatorer, og anvende dem til den biomolekylære verden, " siger Lulu Qian, en senior postdoktor i bioteknik ved Caltech og hovedforfatter på et papir offentliggjort i 3. juni-udgaven af tidsskriftet Videnskab .
Sammen med Erik Winfree, Caltech professor i datalogi, beregnings- og neurale systemer, og bioteknik, Qian brugte en ny slags DNA-baseret komponent til at bygge det største kunstige biokemiske kredsløb, der nogensinde er lavet. Tidligere laboratoriefremstillede biokemiske kredsløb var begrænsede, fordi de fungerede mindre pålideligt og forudsigeligt, når de blev skaleret til større størrelser, Qian forklarer. Den sandsynlige årsag bag denne begrænsning er, at sådanne kredsløb har brug for forskellige molekylære strukturer for at implementere forskellige funktioner, gør store systemer mere komplicerede og svære at fejlfinde. Forskernes nye tilgang, imidlertid, involverer komponenter, der er enkle, standardiseret, pålidelig, og skalerbar, hvilket betyder, at endnu større og mere komplekse kredsløb kan laves og stadig fungerer pålideligt.
"Du kan forestille dig, at i computerindustrien, du vil lave bedre og bedre computere, " siger Qian. "Dette er vores indsats for at gøre det samme. Vi ønsker at lave bedre og bedre biokemiske kredsløb, der kan udføre mere sofistikerede opgaver, får molekylære enheder til at virke på deres miljø."
At bygge deres kredsløb, forskerne brugte stykker DNA til at lave såkaldte logiske porte - enheder, der producerer on-off-outputsignaler som svar på on-off inputsignaler. Logiske porte er byggestenene i de digitale logiske kredsløb, der gør det muligt for en computer at udføre de rigtige handlinger på det rigtige tidspunkt. I en konventionel computer, logiske porte er lavet med elektroniske transistorer, som er koblet sammen for at danne kredsløb på en siliciumchip. Biokemiske kredsløb, imidlertid, bestå af molekyler, der flyder i et reagensglas med saltvand. I stedet for at være afhængig af elektroner, der strømmer ind og ud af transistorer, DNA-baserede logiske porte modtager og producerer molekyler som signaler. De molekylære signaler går fra en specifik port til en anden, forbinder kredsløbet, som om de var ledninger.
Winfree og hans kolleger byggede første gang et sådant biokemisk kredsløb i 2006. I dette arbejde, DNA-signalmolekyler forbundet flere DNA-logiske porte til hinanden, danner det, der kaldes et flerlagskredsløb. Men dette tidligere kredsløb bestod kun af 12 forskellige DNA-molekyler, og kredsløbet blev bremset med et par størrelsesordener, når det blev udvidet fra en enkelt logisk gate til et femlagskredsløb. I deres nye design, Qian og Winfree har konstrueret logiske porte, der er enklere og mere pålidelige, giver dem mulighed for at lave kredsløb mindst fem gange større.
Deres nye logiske porte er lavet af stykker af enten korte, enkeltstrenget DNA eller delvist dobbeltstrenget DNA, hvor enkeltstrenge stikker ud som haler fra DNA'ets dobbelthelix. De enkeltstrengede DNA-molekyler fungerer som input- og outputsignaler, der interagerer med de delvist dobbeltstrengede.
"Molekylerne flyder bare rundt i opløsning, støder ind i hinanden fra tid til anden, " Winfree forklarer. "Af og til, en indkommende streng med den rigtige DNA-sekvens vil zippe sig selv op til en streng, mens den samtidig udpakker en anden, frigiver det i opløsning og lader det reagere med endnu en streng." Fordi forskerne kan kode den DNA-sekvens, de ønsker, de har fuld kontrol over denne proces. "Du har denne programmerbare interaktion, " han siger.
Qian og Winfree lavede flere kredsløb med deres tilgang, men den største – der indeholder 74 forskellige DNA-molekyler – kan beregne kvadratroden af ethvert tal op til 15 (teknisk set, et hvilket som helst fire-bit binært tal) og rund ned svaret til nærmeste heltal. Forskerne overvåger derefter koncentrationerne af outputmolekyler under beregningerne for at bestemme svaret. Beregningen tager omkring 10 timer, så det vil ikke erstatte din bærbare computer på et øjeblik. Men formålet med disse kredsløb er ikke at konkurrere med elektronik; det er for at give videnskabsmænd logisk kontrol over biokemiske processer.
Deres kredsløb har flere nye funktioner, siger Qian. Fordi reaktioner aldrig er perfekte - molekylerne binder ikke altid ordentligt, for eksempel - der er iboende støj i systemet. Det betyder, at de molekylære signaler aldrig er helt tændt eller slukket, som det ville være tilfældet for ideel binær logik. Men de nye logiske porte er i stand til at håndtere denne støj ved at undertrykke og forstærke signaler - f.eks. booste et signal, der er på 80 procent, eller hæmme en, der er på 10 procent, hvilket resulterer i signaler, der enten er tæt på 100 procent til stede eller ikke-eksisterende.
Alle logiske porte har identiske strukturer med forskellige sekvenser. Som resultat, de kan standardiseres, så de samme typer komponenter kan kobles sammen for at lave et hvilket som helst kredsløb, du ønsker. Hvad mere er, Qian siger, du behøver ikke vide noget om det molekylære maskineri bag kredsløbet for at lave et. Hvis du vil have et kredsløb, sige, automatisk diagnosticerer en sygdom, du sender bare en abstrakt repræsentation af de logiske funktioner i dit design til en compiler, som forskerne leverer online, som derefter vil oversætte designet til de DNA-komponenter, der er nødvendige for at bygge kredsløbet. I fremtiden, en ekstern producent kan derefter lave disse dele og give dig kredsløbet, klar til at gå.
Kredsløbskomponenterne er også tunbare. Ved at justere koncentrationerne af typerne af DNA, forskerne kan ændre funktionerne af de logiske porte. Kredsløbene er alsidige, med plug-and-play-komponenter, der nemt kan omkonfigureres til at omkoble kredsløbet. Enkelheden af de logiske porte giver også mulighed for mere effektive teknikker, der syntetiserer dem parallelt.
"Som Moores lov for siliciumelektronik, som siger, at computere bliver eksponentielt mindre og mere kraftfulde hvert år, molekylære systemer udviklet med DNA-nanoteknologi er blevet fordoblet i størrelse omtrent hvert tredje år, " siger Winfree. Qian tilføjer, "Drømmen er, at syntetiske biokemiske kredsløb en dag vil opnå kompleksiteter, der kan sammenlignes med livet selv."
Sidste artikelMetode til at skabe enkelt-krystal arrays af grafen udviklet
Næste artikelNye uorganiske halvlederlag lover solenergi
 Varme artikler
Varme artikler
-
 Nanorørskov i en mikrofluidisk kanal kan hjælpe med at opdage sjældne proteiner og viraEn mønstret og cylindrisk struktur, der består af kulstof nanorør. Ingeniører ved MIT har udtænkt en ny teknik til at fange molekyler, der er svære at opdage, ved hjælp af skove af kulstof nanorør
Nanorørskov i en mikrofluidisk kanal kan hjælpe med at opdage sjældne proteiner og viraEn mønstret og cylindrisk struktur, der består af kulstof nanorør. Ingeniører ved MIT har udtænkt en ny teknik til at fange molekyler, der er svære at opdage, ved hjælp af skove af kulstof nanorør -
 Fremgangsmåde til visualisering af defekter i krystalfaste stoffer forstærket af kunstig intellige…Kredit:CEA Krystaller er allestedsnærværende:de fleste metaller, for eksempel, er krystallinske. Kendt for den næsten perfekte organisering af deres atomer, krystaller indeholder ikke desto mindre
Fremgangsmåde til visualisering af defekter i krystalfaste stoffer forstærket af kunstig intellige…Kredit:CEA Krystaller er allestedsnærværende:de fleste metaller, for eksempel, er krystallinske. Kendt for den næsten perfekte organisering af deres atomer, krystaller indeholder ikke desto mindre -
 Fremstilling af 3-D nanosuperledere med DNAEn illustration, der viser, hvordan højt nanostrukturerede 3-D superledende materialer kan skabes baseret på DNA-selvsamling. Kredit:Brookhaven National Laboratory Tredimensionelle (3-D) nanostruk
Fremstilling af 3-D nanosuperledere med DNAEn illustration, der viser, hvordan højt nanostrukturerede 3-D superledende materialer kan skabes baseret på DNA-selvsamling. Kredit:Brookhaven National Laboratory Tredimensionelle (3-D) nanostruk -
 Billeddannelse af nanoporøse metaller med elektronstråler giver dyb indsigt i usædvanlige optiske…Et nærbillede af nanoporøst sølv afslører uregelmæssige overfladetræk (øverst). A*STAR-forskere har genereret et kort over de forskellige farver, der absorberes lokalt af de nanoporøse strukturer (ned
Billeddannelse af nanoporøse metaller med elektronstråler giver dyb indsigt i usædvanlige optiske…Et nærbillede af nanoporøst sølv afslører uregelmæssige overfladetræk (øverst). A*STAR-forskere har genereret et kort over de forskellige farver, der absorberes lokalt af de nanoporøse strukturer (ned
- Dielektrisk sammenbrud Vs. Test for isolationsmodstand
- Irans netstop først for effektivt at isolere en hel nation
- Forskere udvikler billigere, hurtigere test for E. coli i drikkevand
- Skadelige effekter øger paradoksalt nok solcellernes effektivitet
- Fysikere tager stop-action-billeder af lysdrevet molekylær reaktion
- Elektronmikroskopi giver fingerpeg om den farverige kemi af guldsmedevinger


